Thiết kế nghiên cứu
Nghiên cứu này đã được phê duyệt bởi Văn phòng Đạo đức Nghiên cứu McGill (Ủy ban Đánh giá Thể chế, A03-M24-21B). Nó được dự định phát triển một nền tảng phổ quát để phát hiện phân tử các bệnh nhiễm trùng đường hô hấp tại điểm chăm sóc. Chúng tôi đã sử dụng các hạt vi rút bất hoạt nhiệt H1N1 cúm A, SARS-CoV-2 RNA và SARS-CoV-2 để chẩn đoán nhanh dựa trên các xét nghiệm RT-LAMP tăng cường plasmon. MERS-CoV RNA và HCoV-229E RNA được sử dụng làm biện pháp kiểm soát âm tính đối với xét nghiệm RT-LAMP. Hơn nữa, một xét nghiệm RCA đã được sử dụng để phát hiện cụ thể các biến thể SARS-CoV-2 đáng lo ngại. Cuối cùng, chúng tôi đã sử dụng 33 mẫu người dương tính với SARS-CoV-2 đã được xác định lại (đã được xác minh bằng RT-PCR) thu được từ Ngân hàng sinh học PRESERVE Pandemic Response của Mạng lưới Y tế Đại học và 15 mẫu âm tính với SARS-CoV-2 (đã được qPCR xác minh) làm đối chứng trong mẫu bệnh phẩm học. Bằng chứng về nguyên tắc, hồ sơ DNA của E. coli và MRSA được thực hiện bằng cách sử dụng các xét nghiệm LAMP. Pseudomonas aeruginosa đã được sử dụng như một biện pháp kiểm soát tiêu cực đối với các xét nghiệm LAMP. Nghiên cứu này được thực hiện ở Montreal, Canada, từ tháng 2020 năm 2022 đến tháng XNUMX năm XNUMX.
Vật liệu
Các vật liệu có nguồn gốc như sau: hạt nano polystyrene (hạt polystyrene (PS-R); Hạt vi mô); RNA SARS-CoV-2 tổng hợp (Bộ sưu tập văn hóa kiểu Mỹ (ATCC); Cedarlane); SARS-CoV-2 B.1.1.7 RNA biến thể Alpha (ATCC VR-3326D; Cedarlane); MERS-CoV (ATCC VR-3248SD; Cedarlane); SARS-CoV-2 bất hoạt bằng nhiệt (ATCC VR-1986HK; Cedarlane); chủng cúm A H1N1 bất hoạt bởi nhiệt, NY/01/09 (0810248CFHI; Cedarlane); Và E. coli (số 211540, Merlan Khoa học). Hỗn hợp chính RT-LAMP so màu và enzyme HiFi Taq DNA Ligase (New England Biolabs); PLP, mồi RCA và đích cDNA tổng hợp SARS-CoV-2, MgCl2, KCl, nicotinamide adenine dinucleotide (NAD) và Triton X-100 (Sigma Aldrich); và mồi LAMP và dithiothreitol (DTT) (Thermo Fisher Khoa học) đã được sử dụng. Nước bọt gộp của người khỏe mạnh (IRHUSL50ML) và nước bọt của một người hiến tặng khỏe mạnh (IRHUSLS5ML) được mua từ Innovative Research và được bảo quản khi đến nơi ở –80 °C. Các mẫu RNA của biến thể SARS-CoV-2 (Omicron, Delta, Eta và Gamma) được lấy từ phòng thí nghiệm hợp tác tại Đại học McGill (Phòng thí nghiệm Vidal). HCoV-229E RNA được lấy từ phòng thí nghiệm hợp tác tại Viện Nghiên cứu Y khoa Lady Davis ở Bệnh viện Đa khoa Jewish (phòng thí nghiệm C. Liang). DNA MRSA và P. aeruginosa DNA được lấy từ phòng thí nghiệm D. Nguyen tại viện nghiên cứu McGill University Health Centers (MUHC). Tất cả các xét nghiệm đã được chuẩn bị bằng cách sử dụng nước cất không chứa UltraPure DNase/RNase (Thermo Fisher Khoa học).
Mồi và đầu dò
Các mồi SARS-CoV-2 RT-LAMP được sử dụng trong nghiên cứu này được thiết kế bởi Yu et al.32 để nhắm mục tiêu RdRp gen trong ORF1ab của bộ gen (được WHO khuyến nghị là gen mục tiêu để phát hiện SARS-CoV-2)31. Độ chọn lọc của mồi được xác nhận thông qua thí nghiệm điện di trên gel (Hình bổ sung. 16a). Thành phần và nồng độ của mồi có sẵn trong Bảng bổ sung 6.
Các đoạn mồi H1N1 RT-LAMP được thiết kế bằng Công cụ thiết kế mồi LAMP của New England Biolabs nhắm vào các trình tự được bảo tồn cao của gen hemagglutinin (HA) của H1N1 IAV. Phần nhận dạng LAMP được đánh giá bởi Công cụ tìm kiếm căn chỉnh cục bộ cơ bản50và không có biến thể trình tự nào được nhìn thấy trong 100 lần truy cập được cung cấp bởi trang web của Trung tâm Thông tin Công nghệ Sinh học Quốc gia. Các đoạn mồi LAMP được chỉ định đã được chọn theo các tham số được tối ưu hóa do giao thức Primer Explorer V5 cung cấp. Độ chọn lọc của mồi được xác nhận thêm thông qua thí nghiệm điện di trên gel (Hình bổ sung. 16b). Thành phần và nồng độ của mồi có sẵn trong Bảng bổ sung 7.
Vị trí đột biến P681H và L452R được xác định theo công cụ CoV-GLUE-Viz và GISAID36,51và các bộ phận nhận dạng PLP được thiết kế dành riêng cho các đột biến36. Ngoài ra, trang đích PLP để phát hiện WT SARS-CoV-2 là RdRp gen, giống như vị trí nhắm mục tiêu cho đoạn mồi LAMP của SARS-CoV-2. Các PLP được thiết kế theo cách mà SNP được phân biệt bởi dòng ngược dòng của PLP, nhóm này cung cấp nhóm 3′-hydroxyl tại gốc nối nối thắt được ghép cặp bên cạnh đầu 5′ được phosphoryl hóa trên sợi đích. Tính xác thực của việc tuần hoàn hóa PLP khi phát hiện SNP đã được xác định bằng cách sử dụng Máy tính Nhiệt độ Phản ứng Ligase Ổn định Nhiệt do New England Biolabs cung cấp. Sau đó, các PLP được đánh giá bằng cách sử dụng máy chủ web Mfold để tránh cấu trúc thứ cấp không mong muốn, đặc biệt là trong trang web nhận dạng PLP. Các đoạn mồi tiến và lùi của RCA được thiết kế để lai với phần đệm của PLP kết nối hoàn toàn hai nhánh cụ thể của PLP. Tính chọn lọc của PLP đã được xác nhận thông qua thí nghiệm điện di trên gel (Hình bổ sung. 21). Thành phần và nồng độ PLP, cũng như thành phần và nồng độ mồi RCA và thành phần cDNA, có sẵn trong Bảng bổ trợ 8–10.
Các mồi LAMP vi khuẩn được sử dụng trong nghiên cứu này được thiết kế bởi Hill et al.52 để nhắm mục tiêu E. coli malB gen và Chen et al.53 để nhắm mục tiêu MRSA mecA gien. Thành phần và nồng độ mồi có sẵn trong Bảng bổ trợ 11 và 12.
chuẩn bị xét nghiệm
Đối với xét nghiệm RT-LAMP, thể tích phản ứng tiêu chuẩn là 20 µl đã được sử dụng. Thể tích này bao gồm 2 µl ×10 hỗn hợp mồi, 10 µl ×2 hỗn hợp chính, 7 µl nước không chứa RNase và 1 µl mẫu RNA. Đầu tiên, các mẫu vi rút bất hoạt bằng nhiệt được ly giải bằng nhiệt ở 95°C trong 3 phút và sau đó được trộn với xét nghiệm. Tiếp theo là ủ ở 65 ° C trong các khoảng thời gian khác nhau để hình dung sự thay đổi màu sắc theo thời gian đối với các mẫu khác nhau.
Phản ứng thắt PLP được thực hiện với thể tích cuối cùng là 10 μl bao gồm 1 μl cDNA tổng hợp của bộ gen RNA SARS-CoV-2, 1 μl 1 μM PLP, 2 μl nước cất UltraPure và 5 μl ×2 no-Tris-HCl HiFi Dung dịch thắt Taq DNA Ligase (20 mM MgCl2, 20 mM KCl, 2 mM NAD, 0.1% Triton X-100, 20 mM DTT, pH 8.50) và 1 μl enzyme HiFi Taq DNA Ligase. Hỗn hợp thắt đầu tiên được ủ ở 95°C trong 5 phút để làm biến tính DNA và sau đó được làm lạnh đến nhiệt độ ủ PLP (tương ứng là 60, 58 và 55°C đối với các PLP P681H, L452R và WT) để các PLP lai với cDNA và nối thông qua enzyme HiFi trong máy điều nhiệt (Analytik Jena). Sau đó, phản ứng thắt đã sử dụng 12 μl hỗn hợp chính WarmStart LAMP ×2 Master Mix và 1.6 μM mồi đảo ngược và chuyển tiếp RCA trong một thể tích cuối cùng là 24 μl. Phản ứng khuếch đại RCA được thực hiện ở 65°C trong các khoảng thời gian khác nhau để hình dung sự thay đổi màu sắc theo thời gian đối với các mẫu khác nhau.
Để tách chiết DNA vi khuẩn, E. coli các mẫu được nuôi cấy qua đêm ở 37 ° C trong môi trường Luria Broth. Nồng độ vi khuẩn được xác định bằng máy quang phổ Spectronic 21D. Aliquots của nồng độ khác nhau của 107 CFU ml-1, 105 CFU ml-1, 104 CFU ml-1, 103 CFU ml-1, 102 CFU ml-1 và 10 CFU ml-1 đã được chuẩn bị bằng cách đình chỉ E. coli cấy trong môi trường Luria Broth. E. coli DNA được chiết xuất bằng cách đun sôi môi trường nuôi cấy ở 95 ° C trong 10 phút. DNA MRSA được lấy từ Trung tâm Y tế Đại học McGill bằng phương pháp ly giải hóa học. Tất cả nồng độ mẫu DNA được đo bằng máy đo quang phổ Nanodrop 2000 và được treo trong Universal Buffer 48 (Bio Basic) để đạt được nồng độ mong muốn.
Xét nghiệm QolorEX đối với các dung dịch tăng vọt đã thử nghiệm SARS-CoV-2 (nước không có RNase và nước bọt khỏe mạnh) có kích thước 8 × 105 RNA sao chép μl-1 thành 5 bản sao RNA μl-1 của SARS-CoV-2 RNA và 90 hạt virus μl-1 đến 0.01 hạt virus μl-1 để SARS-CoV-2 bị bất hoạt bởi nhiệt phù hợp với phạm vi phù hợp về mặt sinh học. Tương tự, một nghiên cứu đã được thực hiện cho Delta B.1.617.2, Omicron B.1.1.529, Omicron BA.4, Omicron BA.2.21, Omicron BA.5.2, Omicron BA.5.1.1, Eta B.1.525 và Gamma P .1 với 8 × 105 RNA sao chép μl-1 để 104 RNA sao chép μl-1.
Xét nghiệm QolorEX cho H1N1 đã nghiên cứu các dung dịch có pha thêm (nước không có RNase và nước bọt khỏe mạnh) có kích thước 8 × 105 RNA sao chép μl-1 thành 5 bản sao RNA μl-1 RNA của cúm A H1N1. Đối với các nghiên cứu về tính chọn lọc, RNA từ nhiều loại vi-rút (SARS-CoV-2, MERS-CoV và HCoV-229E) đã được sử dụng ở kích thước 8 × 105 RNA sao chép μl-1.
Xét nghiệm QolorEX cho E. coli đã nghiên cứu các dung dịch tăng vọt (nước không có RNase) 7.2 × 106 bản sao gDNA ml-1 đến 7.2 gDNA sao chép ml-1, tương đương với 0.0343 ng μl-1 đến 3.43 × 10-8 ng μl-1 of E. coli ADN. Đối với các nghiên cứu chọn lọc, DNA từ nhiều vi khuẩn (E. coli, MRSA và P. aeruginosa) được kiểm tra ở nồng độ 102 gDNA sao chép μl-1.
Xét nghiệm QolorEX cho MRSA đã nghiên cứu các dung dịch có pha thêm (nước không có RNase) là 105 bản sao gDNA ml-1 đến 1 gDNA sao chép ml-1, tương đương với 3.05 × 10-4 ng μl-1 đến 3.05 × 10-9 ng μl-1 của DNA MRSA. Đối với các nghiên cứu chọn lọc, DNA từ nhiều loại vi khuẩn (MRSA, E. coli và P. aeruginosa) được kiểm tra ở nồng độ 102 gDNA sao chép μl-1.
Xét nghiệm QolorEX RCA được thực hiện trong các dung dịch có pha thêm (nước không có RNase và nước bọt khỏe mạnh) 8 × 105 cDNA sao chép µl-1 đến 5 bản sao cDNA µl-1 cDNA tổng hợp của các trình tự P681H, L452R và WT SARS-CoV-2. Về tính chọn lọc, P681H PLP được đánh giá với sự có mặt của P681H, WT-P681H và L452R cDNA. Chiến lược tương tự đã được sử dụng cho thử nghiệm tính chọn lọc của L452R PLP bằng cách sử dụng các đích cDNA L452R, WT- L452R và P681H cũng như WT PLP khi có các trình tự cDNA WT, P681H và L452R. Tất cả các mục tiêu cDNA được đánh giá ở nồng độ 105 cDNA sao chép µl-1.
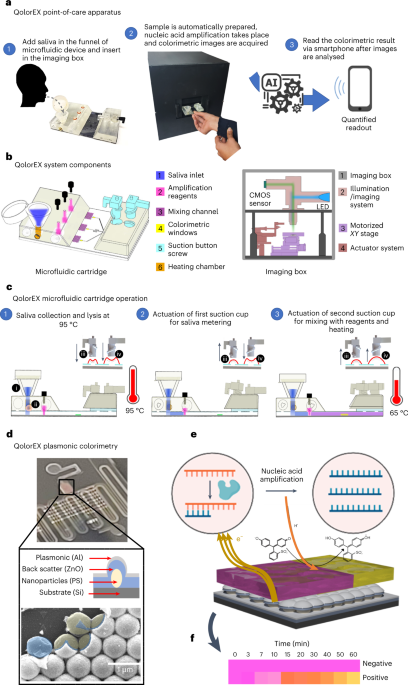
Hộp chụp ảnh và xử lý mẫu tự động QolorEX
Thiết lập hình ảnh di động có ba thành phần chính: thiết lập hình ảnh chiếu sáng epi, mô-đun xử lý chất lỏng và các thành phần tự động hóa (Hình bổ sung. 1). Vỏ ngoài của thiết lập được chế tạo hoàn toàn bằng cách in 3D mô hình lắng đọng nóng chảy (Prusa I3 Mk3, Prusa) ở độ phân giải lớp 0.3 mm.
Thiết lập hình ảnh chiếu sáng epi được thiết kế để ghi lại sự thay đổi so màu của dung dịch xét nghiệm. Thiết lập hình ảnh có mô-đun chiếu sáng và mô-đun xử lý và chụp ảnh. Vật kính ×20 (TU Plan Fluor EPI ×20, Nikon) được sử dụng làm thấu kính tụ quang để chụp ảnh. Tất cả các thành phần quang học được sử dụng đều được mua từ Thorlabs. Hình ảnh cuối cùng được chiếu lên cảm biến CMOS (Sony IMX477R, 12.3MP, Raspberry Pi.) và được xử lý thêm bởi Raspberry Pi 4 (Raspberry Pi).
Để tự động hóa quy trình, chúng tôi đã sử dụng năm bộ truyền động tuyến tính (Actuonix) thực hiện nhiều bước trong xử lý chất lỏng (Hình bổ sung. 1–3). Thiết lập sử dụng một mô-đun gia nhiệt, là một que hàn di động (TS-100), để ly giải mẫu. MỘT XY giai đoạn dịch mã được sử dụng để quét các vùng khác nhau trong và giữa các vùng của buồng phát hiện. Chúng tôi đã sử dụng một giai đoạn điều khiển số được vi tính hóa hướng dẫn tuyến tính được điều khiển bởi một động cơ bước (FUYU). Cả hệ thống truyền động và sân khấu đều được điều khiển bằng bộ vi điều khiển Arduino UNO (Arduino). Hai bộ phận làm nóng được điều khiển bằng mô-đun rơle hai kênh (Yizhet). Quá trình truyền và phân tích dữ liệu tự động trong hệ thống của chúng tôi được điều khiển bởi bộ vi điều khiển Raspberry Pi 4 (Raspberry Pi). Các kết quả được hiển thị trên một ứng dụng được thiết kế bởi MIT App Inventor (Hình bổ sung. 2).
Hộp mực vi lỏng và chế tạo nền tảng plasmonic
Quá trình chế tạo tuân theo một giao thức đã được thiết lập27,54,55 và được trình bày chi tiết trong Thông tin bổ sung (Hình bổ sung. 5). Tóm lại, hộp vi lỏng dựa trên một tấm wafer silicon sáu inch. Quy trình in thạch bản gồm ba bước được sử dụng để tạo mẫu cho phần tử gia nhiệt bị cản trở và các tính năng vi lỏng. Đầu tiên, một bước in thạch bản được thực hiện để chuyển các tính năng của bộ gia nhiệt (chiều rộng, 400 µm) và miếng đệm (chiều dài, 5 mm; chiều rộng, 2 mm) sang lớp cản quang thông qua mặt nạ quang với các mẫu mong muốn. Tiếp theo là quá trình ăn mòn oxit đệm để loại bỏ oxit tự nhiên và ăn mòn kali hydroxit cho quá trình ăn mòn silicon 200 nm. Tiếp theo, một quy trình nâng lên để lắng đọng có chọn lọc các phần tử gia nhiệt trong các rãnh khắc được thực hiện. Quá trình này bắt đầu bằng bước in thạch bản thứ hai, sau đó là sự lắng đọng chùm tia điện tử có bước sóng 240 nm Al bằng cách sử dụng Temescal BJD 1800. Theo đó, quá trình tách được hoàn thành bằng cách nhúng chìm trong chất tẩy phù hợp. Tiếp theo, bước in thạch bản cuối cùng được thực hiện để tạo mẫu cho các tính năng của thiết bị chất lỏng bao gồm các cổng đầu vào/đầu ra (đường kính 2 mm), buồng ly giải (chiều dài, 1.74 mm; chiều rộng, 1.5 mm; chiều sâu, 50 µm), các kênh trộn (chiều rộng, 200 µm; chiều cao, 50 µm) và cửa sổ plasmonic (chiều dài, 1.94 mm; chiều rộng, 1.5 mm; chiều cao, 50 µm) vào một lớp SU-8 (SU-8 2050). Sau đó, tấm bán dẫn mỏng 26.5 inch được cắt thành các chip riêng lẻ (chiều dài, 35 mm; chiều rộng, 3240 mm) bằng cách sử dụng cưa cắt hạt Disco DAD XNUMX.
Tiếp theo là sự tích hợp của các nền tảng nhạy cảm với màu sắc trong hộp mực vi lỏng bằng cách sử dụng kỹ thuật tạo mô hình nano không thể tin được. Một cách tiếp cận chung được sử dụng để phát triển một lớp đơn lớp hạt nano tự lắp ráp keo ở giao diện không khí nước56. Tiếp theo, các cấu trúc tổ ong thu được được chuyển đến buồng phát hiện màu trong hộp mực vi lỏng. Sau đó, một màng mỏng ZnO (120 nm) được lắng đọng, tiếp theo là một lớp Al mỏng (10 nm) để tạo ra cộng hưởng plasmon bề mặt cục bộ có thể điều chỉnh với nền trắng.
Polydimetylsiloxan (PDMS) được điều chế bằng cách sử dụng chất đàn hồi và chất liên kết ngang theo tỷ lệ 10:1, khử khí trong bình hút ẩm và ủ ở 65°C qua đêm. PDMS đã xử lý được cắt theo kích thước của hộp vi lỏng và các cổng đầu vào/đầu ra được đục lỗ bằng máy đục lỗ PDMS (Thermo Fisher Khoa học) và được sử dụng để niêm phong thiết bị. Quá trình hàn kín bao gồm xử lý plasma trong 50 giây, sau đó ủ qua đêm ở 105°C. Tiếp theo, một lớp cốc hút dựa trên PDMS được chế tạo bằng khuôn in 3D in 3D (SLA) (Mẫu XNUMX, Formlabs). Các giác hút sau đó được liên kết với lớp PDMS lỏng bằng liên kết được kích hoạt bằng plasma.
Hộp mực xử lý mẫu (được sản xuất bằng máy in 3D SLA ở độ phân giải 50 µm trên z trục) được liên kết với chip vi lỏng được phủ PDMS bằng cách sử dụng băng keo hai mặt có lợi cho liên kết được kích hoạt bằng plasma và được đặt trong lò ở 95 °C trong 90 phút. Trong hộp in 3D, có hai miếng kim loại bằng đồng thau (McMaster Carr) để ly giải mẫu.
Đang xử lý hình ảnh
Quá trình xử lý hình ảnh bắt đầu với một bộ dữ liệu bao gồm ba hình ảnh từ mỗi điều kiện được nghiên cứu, sẽ được chia thành các hình ảnh nhỏ để tạo tổng cộng 40 và 60 điểm dữ liệu tương ứng cho mỗi điều kiện về độ nhạy và độ chọn lọc. Cụ thể đối với các mẫu lâm sàng, mỗi mẫu được nghiên cứu trong ba buồng thu thập song song của chip vi lỏng; vì tất cả các hình ảnh được chụp ba lần và sau đó được chia ra, tổng cộng 90 điểm dữ liệu đã được nghiên cứu cho từng mẫu bệnh nhân. Tất cả quá trình xử lý hình ảnh bao gồm cắt xén 20% bên ngoài của hình ảnh RGB gốc để loại bỏ hiệu ứng vòng cà phê57, theo sau là ứng dụng bộ lọc màu xanh lam, trong đó các pixel có giá trị màu sắc nằm trong khoảng từ 85 đến 140 (phạm vi màu xanh lam) bị xóa và thay thế bằng giá trị trung bình của phần còn lại của hình ảnh57. Hình ảnh được lọc màu xanh lam sau đó được đặt ngưỡng bằng cách thay thế các pixel ít bão hòa hơn 25% bằng giá trị trung bình của phần còn lại của hình ảnh được lọc. Cuối cùng, hình ảnh được xử lý được cắt thành các hình ảnh phụ mà từ đó một số tính năng được trích xuất, do đó việc triển khai các công thức được trình bày chi tiết trong Sergyan58. Việc sửa đổi được thực hiện cho các công thức bao gồm hoán đổi cường độ thang độ xám với cường độ của từng kênh RGB58. Tổng cộng có 18 giá trị được trích xuất từ mỗi hình ảnh phụ tương ứng với giá trị màu trung bình, độ lệch chuẩn, chế độ, độ lệch, năng lượng và entropy cho từng kênh RGB.
Để tự động hóa các mẫu người thật, chúng tôi đã triển khai thuật toán học máy có giám sát để phân loại hình ảnh thành hai loại, không bị nhiễm và bị nhiễm (Hình bổ sung. 14). Một SVM với hạt nhân chức năng cơ sở xuyên tâm (RBF) đã được thiết lập với các siêu đường kính của nó C và gamma được đánh giá bằng tìm kiếm Bayes59 và sự vắng mặt quá mức được xác thực thông qua xác thực chéo năm lần. Cơ sở dữ liệu cho nghiên cứu này được tích hợp bởi 33 đối chứng bị nhiễm và 15 đối chứng không bị nhiễm; đối với mọi mẫu, các nghiên cứu được thực hiện ba lần, thu được tổng cộng chín hình ảnh cho mỗi thời điểm. Các bộ dữ liệu được chia thành hai loại: không bị nhiễm (âm tính) và bị nhiễm (dương tính). Sau đó, chúng được chia thành các tập huấn luyện và kiểm tra riêng biệt. Tập huấn luyện bao gồm hai phần ba vectơ từ bệnh nhân 1, 7, 9, 11, 15, 19, 21, 23, 25, 29 và 31 và âm tính 2, 3, 4, 5, 6, 7, 8, 9, 11, 13 và 15; tập kiểm tra được tích hợp bởi các vectơ còn lại. SVM đưa ra một dự đoán cho từng vectơ của bộ thử nghiệm là không bị nhiễm hoặc bị nhiễm.
Mẫu lâm sàng và tuyên bố đạo đức
Chúng tôi đã lấy các mẫu nước bọt thông qua Ngân hàng sinh học ứng phó với đại dịch PRESERVE của Mạng lưới Y tế Đại học (UHN REB 20-5364 và McGill IRB số A03-M24-21B). 33 mẫu lâm sàng SARS-CoV-2 được thu thập từ các bệnh nhân trưởng thành có các triệu chứng COVID-19 như sốt, mệt mỏi và ho khan. Tất cả các mẫu đã được xác định lại và cho kết quả dương tính với SARS-CoV-2 bằng RT-PCR. Hơn nữa, tải lượng vi-rút được đánh giá bởi qPCR (QuantStudio 12K Flex, Thermo Fisher Khoa học). Trình tự mồi qPCR được cung cấp trong Bảng bổ sung 13. Các mẫu được đánh giá tại một cơ sở Cấp 2+ nằm trong Viện Lady Davis tại Bệnh viện Đa khoa Do Thái.
phép đo điện hóa
Các phép đo điện hóa được thực hiện trong một tế bào ba điện cực thông thường bằng cách sử dụng bộ chiết áp/bộ điều chỉnh điện kế Autolab PGSTAT204. Các nền tảng plasmonic được sử dụng làm điện cực làm việc, trong khi Ag/AgCl và dây bạch kim lần lượt đóng vai trò là điện cực tham chiếu và đối kháng. Điện thế của các phép thử vôn kế tuần hoàn nằm trong khoảng từ −1 đến 1 V so với điện cực tham chiếu có tốc độ quét là 50 mV s-1. Phép đo phản ứng phát quang được thực hiện bằng cách sử dụng kỹ thuật đo ampe kế thời gian dưới ánh sáng khả kiến xung quanh được cắt nhỏ (chu kỳ bật-tắt đèn, 5 giây) ở điện thế phân cực 1 V so với Ag/AgCl trong dung dịch xét nghiệm khuếch đại axit nucleic dạng nước (10 phần LAMP Master Trộn, 2 phần ×10 mồi gốc, 7 phần nước không chứa RNase và 1 phần mẫu RNA đích).
Đặc tính
Đối với các nghiên cứu tối ưu hóa trên nền tảng plasmonic, các kỹ thuật mô tả đặc tính vật lý bao gồm kính hiển vi lực nguyên tử (AFM) và SEM đã được sử dụng. AFM được thực hiện với thiết bị Bruker MultiMode8 trong khi SEM được thực hiện bằng kính hiển vi điện tử quét môi trường FEI Quanta 450.
Các đặc tính quang học được thực hiện thông qua thiết bị nhìn thấy được bằng tia cực tím cận hồng ngoại Lambda750 trong đó các phép đo độ hấp thụ ánh sáng trắng trong 60 phút khuếch đại được thu thập từ 200 nm đến 850 nm. Cài đặt nghiên cứu bao gồm sự tích lũy của các giải pháp điểm thời gian trên nền tảng nơi các chùm ánh sáng tới và được thu thập có tỷ lệ bình thường đối với nền tảng. Sự phân bố điện trường cơ bản của nền tảng đã được nghiên cứu bằng cách sử dụng mô-đun miền thời gian sai phân hữu hạn (v.8.21.1781, Lumerical Solutions). Các đặc tính truyền nhiệt và dòng chất lỏng cơ bản của hộp vi lỏng đã được nghiên cứu bằng cách sử dụng COMSOL Multiphysics (v.5.6).
Phân tích thống kê
Kết quả được coi là giá trị trung bình ± sai số chuẩn của giá trị trung bình đối với các phép đo ba lần như được giải thích trong phần xử lý ảnh. Gói phần mềm OriginPro (OriginLab, 2021) đã được sử dụng để phân tích thống kê. Giới hạn phát hiện và phạm vi tuyến tính được tính toán bằng phương pháp hồi quy tuyến tính, bao gồm độ dốc của đường và sai số chuẩn của phần chặn. Ý nghĩa thống kê được đánh giá bằng cách sử dụng phân tích phương sai một chiều (ANOVA) với bài kiểm tra Tukey post hoc để so sánh trung bình. Sự khác biệt của tập dữ liệu được coi là có ý nghĩa thống kê đối với P < 0.001. Ứng dụng vẽ biểu đồ So sánh theo cặp (v.3.60, OriginLab) đã được sử dụng để tạo các số liệu bằng cách sử dụng thận trọng P các giá trị.
Tóm tắt báo cáo
Thông tin thêm về thiết kế nghiên cứu có sẵn trong Tóm tắt báo cáo danh mục đầu tư tự nhiên liên kết đến bài viết này.
- Phân phối nội dung và PR được hỗ trợ bởi SEO. Được khuếch đại ngay hôm nay.
- PlatoAiStream. Thông minh dữ liệu Web3. Kiến thức khuếch đại. Truy cập Tại đây.
- Đúc kết tương lai với Adryenn Ashley. Truy cập Tại đây.
- Mua và bán cổ phần trong các công ty PRE-IPO với PREIPO®. Truy cập Tại đây.
- nguồn: https://www.nature.com/articles/s41565-023-01384-5
- : có
- :là
- :Ở đâu
- ][P
- 1
- 10
- 100
- 11
- 12
- 13
- 14
- 15%
- 20
- 200
- 2008
- 2011
- 2016
- 2018
- 2019
- 2020
- 2021
- 2022
- 2023
- 2050
- 23
- 24
- 26
- 27
- 31
- 3d
- In ấn 3D
- 40
- 50
- 60
- 66
- 7
- 8
- 9
- a
- AC
- tăng tốc
- Theo
- cho phù hợp
- tích lũy
- Đạt được
- có được
- Ngoài ra
- Người lớn
- AL
- thuật toán
- Tất cả
- Alpha
- hoàn toàn
- Môi trường xung quanh
- American
- Khuếch đại
- an
- phân tích
- Neo
- và
- ứng dụng
- Các Ứng Dụng
- áp dụng
- phương pháp tiếp cận
- phê duyệt
- Tháng Tư
- Arduino
- LÀ
- cánh tay
- đến
- bài viết
- AS
- đánh giá
- At
- Tự động
- Tự động hóa
- có sẵn
- tránh
- Trục
- lý lịch
- Vi khuẩn
- dựa
- cơ bản
- cơ sở
- Bayesian
- BE
- được
- giữa
- thiên vị
- công nghệ sinh học
- máu
- Màu xanh da trời
- bảng
- cả hai
- mua
- thau
- một thời gian ngắn
- đệm
- by
- tính
- Canada
- Ung thư
- Các tế bào ung thư
- nắm bắt
- mà
- thực hiện
- mang
- Tế bào
- Trung tâm
- Trung tâm
- trung tâm
- CGI
- phòng
- thay đổi
- kênh
- đặc điểm
- hóa chất
- chen
- Chip
- Snacks
- các lớp học
- Phân loại
- Nhấp chuột
- Lâm sàng
- Cà Phê
- bộ sưu tập
- Chung
- so
- sự so sánh
- Hoàn thành
- hoàn toàn
- các thành phần
- sáng tác
- Bao gồm
- tập trung
- Liên quan
- điều kiện
- thực hiện
- trao
- XÁC NHẬN
- connect
- do hậu quả
- bảo thủ
- xem xét
- bao gồm
- điều khiển
- kiểm soát
- điều khiển
- thông thường
- bản sao
- Vi rút coronavirus
- Tương ứng
- Counter
- Covid-19
- văn hóa
- Cup
- Cắt
- chu kỳ
- Cha
- dữ liệu
- điểm dữ liệu
- Cơ sở dữ liệu
- bộ dữ liệu
- Davis
- Tháng mười hai
- đồng bằng
- ký gửi
- chiều sâu
- Thiết kế
- thiết kế
- mong muốn
- chi tiết
- Phát hiện
- xác định
- phát triển
- sai lệch
- thiết bị
- Thiết bị (Devices)
- sự khác biệt
- khác nhau
- khác biệt
- Phân biệt
- phân phối
- Chia
- dna
- thực hiện
- điều khiển
- khô
- suốt trong
- E&T
- mỗi
- hay
- thành phần
- các yếu tố
- việc làm
- sử dụng
- cho phép
- cuối
- năng lượng
- Nước Anh
- nâng cao
- môi trường
- EPI
- Trang thiết bị
- Tương đương
- lôi
- đặc biệt
- thành lập
- Ether (ETH)
- đạo đức
- đánh giá
- Mỗi
- thử nghiệm
- Giải thích
- người khám phá
- khai thác
- Cơ sở
- mệt mỏi
- Tính năng
- này
- Sốt
- Sung
- Số liệu
- Phim ảnh
- phim
- lọc
- cuối cùng
- Cuối cùng
- Tên
- phù hợp với
- dòng chảy
- chất lỏng
- sau
- sau
- Trong
- Buộc
- hình thức
- Forward
- từ
- chức năng
- cơ bản
- xa hơn
- Tổng Quát
- tạo ra
- gen
- Nhóm
- có
- Xử lý
- cho sức khoẻ
- khỏe mạnh
- cao
- cao
- Số lượt truy cập
- Bệnh viện
- HTTPS
- Nhân loại
- khinh khí
- i
- i3
- IEEE
- hình ảnh
- hình ảnh
- Hình ảnh
- thực hiện
- thực hiện
- in
- sự cố
- bao gồm
- Bao gồm
- ủ
- SỰ CỐ
- hệ thống riêng biệt,
- Nhiễm trùng
- Cúm
- thông tin
- sáng tạo
- Chèn
- Viện
- Thể chế
- tích hợp
- hội nhập
- Sự thông minh
- dự định
- Quốc Tế
- trong
- IT
- ITS
- phòng thí nghiệm
- phòng thí nghiệm
- phụ nữ
- Họ
- lớp
- học tập
- Chiều dài
- ống kính
- ít
- Cấp
- ánh sáng
- giới hạn
- Dòng
- LINK
- liên kết
- tải
- địa phương
- máy
- học máy
- thực hiện
- chính
- sản xuất
- chủ
- Mata
- vật liệu
- nghĩa là
- đo lường
- đo
- Phương tiện truyền thông
- y khoa
- nghiên cứu y học
- kim loại
- phương pháp
- phương pháp
- Kính hiển vi
- Kính hiển vi
- phút
- MIT
- hỗn hợp
- Trộn
- hỗn hợp
- Chế độ
- mô hình hóa
- mô-đun
- phân tử
- giám sát
- Montreal
- Hơn thế nữa
- động cơ
- MRSA
- nhiều
- Đột biến
- công nghệ nano
- quốc dân
- tự nhiên
- Thiên nhiên
- tiêu cực
- Mới
- tiếp theo
- Nguyễn
- NIH
- Không
- bình thường
- Mục tiêu
- thu được
- of
- cung cấp
- Office
- on
- Thành phần quang học
- tối ưu hóa
- tối ưu hóa
- or
- cơ quan
- nguyên
- vfoXNUMXfipXNUMXhfpiXNUMXufhpiXNUMXuf
- ra
- qua đêm
- gói
- tập giấy
- ghép đôi
- đại dịch
- Song song
- thông số
- một phần
- hạt
- các bộ phận
- bệnh nhân
- bệnh nhân
- Họa tiết
- mô hình
- thực hiện
- kinh nguyệt
- vật lý
- kế hoạch
- Plasma
- nền tảng
- Nền tảng
- bạch kim
- plato
- Thông tin dữ liệu Plato
- PlatoDữ liệu
- Điểm
- điểm
- danh mục đầu tư
- cổng
- tích cực
- Bài đăng
- tiềm năng
- dự đoán
- chuẩn bị
- sự hiện diện
- mồi
- nguyên tắc
- in ấn
- QUY TRÌNH
- quá trình
- Quá trình Tự động hóa
- xử lý
- xử lý
- profiling
- dự
- bằng chứng
- giao thức
- cho
- cung cấp
- cung cấp
- định lượng
- phạm vi
- nhanh
- Mâm xôi
- Raspberry Pi
- Tỷ lệ
- tỉ lệ
- rbf
- phản ứng
- thực
- thời gian thực
- công nhận
- đề nghị
- hồi quy
- Đặt lại
- phát hành
- có liên quan
- còn lại
- tẩy
- Đã loại bỏ
- thay thế
- Báo cáo
- nghiên cứu
- Độ phân giải
- cộng hưởng
- tương ứng
- phản ứng
- phản ứng
- REST của
- kết quả
- Kết quả
- đảo ngược
- xem xét
- RGB
- Nhẫn
- RNA
- s
- Nước bọt
- tương tự
- SARS-CoV 2
- quy mô
- quét
- quét
- SCI
- Tìm kiếm
- Thứ hai
- trung học
- Phần
- đã xem
- chọn
- chọn lọc
- SEM
- nhạy cảm
- Độ nhạy
- Trình tự
- định
- bộ
- thiết lập
- một số
- chia sẻ
- Sigma
- ý nghĩa
- có ý nghĩa
- Silicon
- Tương tự
- kể từ khi
- website
- Các trang web
- Kích thước máy
- nghiêng
- Slope
- nhỏ
- Phần mềm
- giải pháp
- Giải pháp
- Sony
- nguồn gốc
- riêng
- đặc biệt
- Traineeship
- Tiêu chuẩn
- bắt đầu
- thống kê
- Bước
- Các bước
- cổ phần
- lưu trữ
- Chủng
- Chiến lược
- cấu trúc
- nghiên cứu
- nghiên cứu
- Học tập
- Sau đó
- như vậy
- phù hợp
- Bề mặt
- đình chỉ
- Hội nghị chuyên đề
- Các triệu chứng
- sợi tổng hợp
- hệ thống
- bàn
- Lấy
- dùng
- Mục tiêu
- nhắm mục tiêu
- mục tiêu
- kỹ thuật
- thử nghiệm
- Kiểm tra
- kiểm tra
- việc này
- Sản phẩm
- sau đó
- Đó
- họ
- điều này
- số ba
- ba bước
- Thông qua
- thời gian
- đến
- công cụ
- Tổng số:
- Hội thảo
- chuyển
- chuyển
- Dịch
- điều trị
- Triton
- hai
- hai phần ba
- kiểu
- Dưới
- phổ cập
- trường đại học
- UNO
- trên
- đã sử dụng
- sử dụng
- xác nhận
- giá trị
- Các giá trị
- biến thể
- xác minh
- linh hoạt
- Versus
- thông qua
- virus
- virus
- có thể nhìn thấy
- khối lượng
- là
- Nước
- Đường..
- we
- web
- máy chủ web
- Website
- TỐT
- là
- cái nào
- trong khi
- trắng
- CHÚNG TÔI LÀ
- sẽ
- Dây điện
- với
- đang làm việc
- thế giới
- Tổ chức Y tế thế giới
- X
- zephyrnet