Incubação
Todos os processos de incubação foram realizados em um ThermoMixer C (Eppendorf), exceto as Figuras Suplementares. 1 e 9 (Incubadora Dry Bath FB15103, Fisher Scientific) e para a Fig. 1c e Figs Suplementares. 3-6 (QuantStudio5, Biossistemas Aplicados da Thermo Fisher Scientific).
Automontagem isotérmica de origamis de DNA 2D
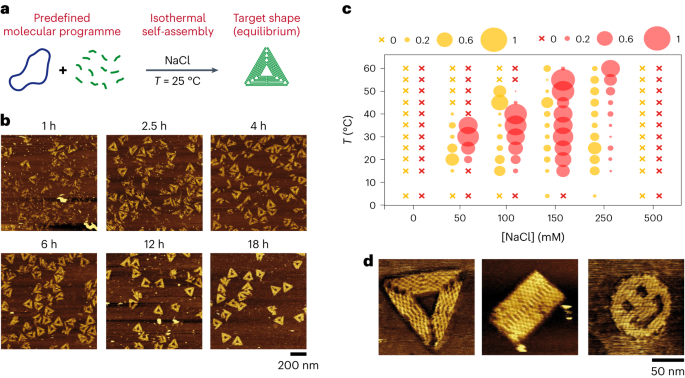
Veja a Fig. 1 e Figs Suplementares. 1-9 e 11-14. Usamos o coquetel básico sem qualquer pré-tratamento térmico e o misturamos diretamente com o tampão desejado antes de um breve vórtice e adição do modelo M13 (1 nM) à solução e mistura suave para cima e para baixo com uma pipeta. A solução foi deixada incubar, sem mais mistura, a uma temperatura fixa durante o período de tempo desejado.
Recozimento térmico de origamis de DNA em tampão TANa
Veja a Fig. Suplementar. 10. Montamos o modelo M13 (1 nM) com uma mistura de grampos (40 nM cada grampo) em TANa suplementado com 100 mM de NaCl. A amostra foi incubada por 10 min a 90 °C e depois submetida a uma rampa térmica em um termociclador peqSTAR 2X (Peqlab) de 70 °C a 20 °C a uma taxa de -1 °C por 10 min.
Purificação por precipitação de PEG
Veja as Figuras Suplementares. 13 e 14. Origamis de DNA obtidos por montagem isotérmica em tampão TANa ([NaCl] = 100 mM) a 25 °C foram purificados a partir de suas cadeias básicas por precipitação com PEG. O método foi inspirado no protocolo apresentado em relatório anterior35. Os origamis de DNA foram diluídos três vezes com uma solução de PEG 8000 e NaCl para atingir concentrações finais de 4% p/v e 500 mM, respectivamente. Após mistura suave, a solução foi deixada incubar por 15 min em temperatura ambiente e centrifugada a 15,000g por 15 minutos. O sobrenadante foi removido e os origamis foram ressuspensos ao seu volume inicial em tampão TANa ([NaCl] = 100 mM). Se necessário, o processo foi repetido para uma segunda purificação consecutiva.
Eletroforese em gel de origamis purificados
Veja a Fig. Suplementar. 13. Preparamos 50 ml de gel de agarose (tipo I baixo EEO, Sigma Aldrich) a 1.5% contendo 4 μl de GR-Green 10,000× (Excellgen) em tampão TBE 1×. Após o resfriamento do gel, introduzimos, em cada poço, 18 µl de 100 pb DNA Ladder (New England Biolabs) ou 18 µl de amostra suplementada com 1× solução SDS de corante de carregamento de DNA (Thermo Scientific). A migração foi realizada a 100 V por 1 h em uma célula de eletroforese de 7 cm preenchida com tampão TBE 1×.
Montagem isotérmica gradual
Veja a Fig. Suplementar. 24. Os grampos do triângulo foram montados em três lotes separados, cada um codificando o canto superior, a parte intermediária e a borda oposta. Um lote (40 nM cada grampo) foi misturado com M13 (1 nM) em tampão TANa ([NaCl] = 100 mM) e o sistema foi deixado para incubar a 25 °C sem mistura adicional. A cada 24 h, removemos o volume necessário para a imagem AFM, adicionamos um lote codificando para uma parte adicional do triângulo (40 nM cada grampo) e deixamos o sistema incubar a 25 °C em tampão TANa ([NaCl] = 100 mM) . Realizamos duas formas diferentes de montagem passo a passo, do canto para o lado oposto e de um lado para o canto oposto.
Preparação isotérmica de triângulos modificados com estreptavidina
Veja a Fig. 2a e Figs Suplementares. 15 e 16. No mesmo tubo, misturamos 1 nM de M13, 40 nM de cada um dos grampos, incluindo os biotinilados, e 2 µM de estreptavidina em tampão TANa suplementado com 100 mM de NaCl. A amostra foi deixada incubar a 25 °C sem mistura adicional por 24 h.
Preparação isotérmica de retângulos SST R4
Veja a Fig. 2b. Misturamos todas as cadeias do retângulo R4 no tampão até uma concentração final de 100 nM em cada cadeia em TANa suplementado com NaCl 100 mM. A amostra foi deixada incubar a 25 °C sem mistura adicional por 24 h.
Eletroforese em gel de retângulos SST R4
Veja a Fig. 2b. Um gel de agarose a 1.5% (EEO baixo tipo I, Sigma Aldrich) foi preparado em tampão TBE 0.5× suplementado com MgCl11 XNUMX mM2 e coloração de DNA verde GB. A eletroforese em gel foi realizada em banho de água gelada por 2 horas a 100 V de voltagem usando uma escada de DNA de 1 kb. Para purificação, a banda alvo do gel foi cortada em pequenos pedaços e colocada em um tubo com coluna de rotação, e a coluna foi submetida à centrifugação a 5,000g por 10 minutos. Para imagens de AFM, a amostra eluída foi diretamente adsorvida em uma placa de mica por 10 minutos dentro da câmara com atmosfera controlada. A amostra foi então enxaguada com 1 ml de 0.5× TBE + 11 mM MgCl2 e observado usando AFM em 0.5× TBE + 11 mM MgCl2.
Preparação isotérmica de nanorredes de DNA
Veja a Fig. 2c e Fig. 17. Misturamos os nove oligonucleotídeos (1 µM de cada nucleotídeo) em tampão TANa suplementado com 100 mM ou 150 mM de NaCl. A amostra foi deixada incubar a 25 °C sem mistura adicional por 24 h.
Recozimento térmico de origamis 3D
Veja a Fig. 3a e Fig. 18. O andaime (7,560 nt M13 para Tb, 8,064 nt M13 para T1) e a mistura básica (10x excesso em cada grampo) foram misturados em tampão contendo 5 mM Tris-HCl, pH 8.0, 1 mM EDTA e 18 mM MgCl2. A mistura foi aquecida a 65 °C por 15 min para desnaturar todas as cadeias de DNA antes de ser resfriada lentamente em um gradiente de 60 °C a 40 °C, ao longo de 41 h para recozer e montar as nanoestruturas de origami 3D.
TEM com coloração negativa
Veja a Fig. 3 e Fig. 18. Para caracterização de TEM, as nanoestruturas de DNA foram primeiro purificadas a partir de 1% de agarose (0.5 × TBE, Tris-borato 45 mM, EDTA 1 mM, pH 8.3) suplementado com MgCl11 XNUMX mM.2 e 0.5 mg ml-1 Sybr SEGURO. As amostras foram migradas no gel por 3 horas com um tampão de corrida de 0.5 × TBE, 11 mM MgCl2 a 2.85 V cm-1 à temperatura ambiente. Bandas correspondentes a estruturas automontadas foram excisadas e transferidas para uma coluna giratória de extração de gel de DNA (Merck) e centrifugadas a 5,000 g por 5 min a 4 °C. Os origamis purificados foram então depositados por adsorção em uma grade revestida de carbono com descarga luminosa (Quantifoil Micro Tools), corados por 60 s com uma solução aquosa de acetato de uranila a 2% (p/v) (Merck) e depois secos com papel de filtro sem cinzas (VWR ). As observações TEM foram realizadas em um microscópio JEM-1400 Flash Tungsten trabalhando a 120 kV, equipado com uma câmera Gatan OneView.
Competição isotérmica
Veja a Fig. Suplementar. 20. Nós agitamos brevemente as misturas de dois conjuntos de grampos que codificam triângulos e retângulos (concentração final de 40 nM para cada grampo) com o tampão (TANa suplementado com NaCl 100 mM) antes de adicionar M13 à mistura (concentração final de 1 nM) e mistura suave com uma pipeta. A amostra foi deixada incubar a 25 °C sem mistura adicional.
Observação AFM de nanoestruturas de DNA em líquido
Observações ambientais de AFM de alta resolução no buffer de amostra foram usadas para todos os dados e imagens de AFM mostrados neste artigo. Exceto a Fig. 4 (veja o protocolo específico abaixo), nanoestruturas de DNA obtidas em tampão TANa (origamis de DNA com ou sem modificação de proteína, retângulos SST R4, nanorredes de DNA) foram adsorvidas em discos de mica recém-clivados de 10 mm de diâmetro (grau Nano-Tec V-1 Muscovite, Micro to Nano Innovative Microscopy Supplies) previamente colados em um disco de metal e tratados com 20 µl de solução de tetracloreto de espermina (0.1 M em água MilliQ) por 10 min e lavados abundantemente, primeiro com água MilliQ e depois com o tampão TANa. Para adsorção da amostra, 15–20 µl de amostra foram depositados na mica recém-tratada com espermina e deixadas para adsorver por 10 min, exceto para experimentos de transformação isotérmica (Fig. 5 e Figs Suplementares. 21-23) onde o tempo foi aumentado para 20 min devido à menor concentração de origamis. A placa de mica foi então enxaguada suavemente com 200 µl do tampão para remover o excesso de grampos e objetos não adsorvidos. Para evitar a secagem da amostra durante a manipulação da mica, deixamos uma fina camada de tampão no topo da amostra adsorvida e a mantivemos à temperatura ambiente em uma câmara com atmosfera controlada (um recipiente selado contendo um pedaço de lenço Kimtech umedecido com MilliQ água). O mesmo protocolo foi feito com amostras contendo magnésio (Fig. Complementar 1, tampões TAEMg e TAMg), exceto que as amostras foram diretamente adsorvidas por 5 min na mica recém-clivada sem qualquer tratamento. As amostras foram observadas com um microscópio de força atômica Cypher ES (Oxford Instruments) em modo de rosqueamento com frequência de ressonância de 17–45 kHz em líquido e 0.09 N m-1 ponta constante de força (BL-AC40TS, Olympus), usando o modo de excitação fototérmica blueDrive. As imagens brutas foram submetidas à subtração polinomial de fundo, correção em nível de plano, alinhamento de linha usando vários métodos e correção de cicatriz horizontal em Gwyddion.
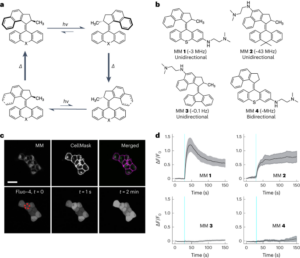
Imagem em tempo real da evolução isotérmica Λ → Δ em uma superfície de bicamada lipídica
Veja a Fig. 4, Texto Suplementar 3 e filmes complementares 1-4. Bicamadas lipídicas suportadas (SLBs) foram obtidas de lipossomas DOPC através do nosso método descrito anteriormente37. As vesículas foram preparadas a partir de um estoque de clorofórmio de DOPC. Após a evaporação do clorofórmio sob uma corrente de gás nitrogênio, os lipídios foram reidratados em água MilliQ para atingir uma concentração lipídica final de 2 mg ml-1. A mistura lipídica foi então agitada em vórtice e sonicada por 60 minutos para produzir pequenas vesículas unilamelares. Para evitar a secagem das bicamadas, os seguintes passos foram realizados numa câmara com atmosfera controlada. Os SLBs foram formados depositando 2 ml da solução de vesículas em discos de mica recém-clivados (previamente colados em uma placa metálica magnética com cola), seguido de 2 µl de TAEMg (Tris-acetato 1×, [EDTA] = 1 mM, [MgCl2] = 12.5 mM). Após 30 min de adsorção, a amostra foi enxaguada com 2 μl de tampão TAEMg para remover lipossomas não adsorvidos e este processo de adsorção foi repetido uma segunda vez para garantir a cobertura ideal da superfície da mica pela bicamada. Ao final do processo de adsorção, a amostra foi enxaguada com 5 µl de TAENa (Tris-acetato 1×, [EDTA] = 1 mM, [NaCl] = 100 mM) para garantir que não haverá Mg livre restante.2+ íons na amostra, o que impediria o dobramento isotérmico dos origamis.
Origamis em forma de Λ modificados com colesterol (Fig. 19) foram preparados misturando uma solução de 10 nM de M13 no tampão TAENa com 20 nM de cada um dos grampos e recozimento reduzindo a temperatura de 70 °C para 4 °C a uma taxa de -0.1 °C min-1. A solução de origami resultante foi usada sem purificação adicional. Em seguida, 2 µl da solução de Λ origamis modificada com colesterol foram depositados no SLB pré-formado, seguido por 2 µl de tampão TAENa. A amostra foi incubada por 60 minutos à temperatura ambiente na câmara com atmosfera controlada, e a superfície foi então fotografada diretamente à temperatura ambiente (T = 26 °C) por AFM em 20 µl de tampão TAENa sem enxágue da superfície. Depois de selecionar uma posição contendo um número suficiente de origamis Λ adsorvidos no SLB, 8 μl dos grampos do lado A em TAENa foram adicionados à amostra sem movê-la ou removê-la do estágio AFM. A solução pendente sobre a amostra foi então suavemente misturada movendo lentamente a sonda AFM para cima e para baixo várias vezes para acelerar a difusão nos grampos do lado A em direção à superfície da mica. A mesma posição foi então digitalizada em média a cada 3 minutos durante 223 minutos. A resolução da imagem foi 512 × 512 de t = 0 para t = 41 min e foi então alterado para 640 × 640. z a escala das imagens foi de 0–10 nm de t = 0 para t = 47 min e 0–7 nm depois. Devido à evaporação durante o processo de imagem, tampão suplementar (8 µl) foi adicionado a t = 63 minutos, t = 144 min e t = 161 min, e 8 µl dos grampos do lado A foram adicionados após t = 170 min.
As imagens AFM foram obtidas em TAENa à temperatura ambiente usando um microscópio de força atômica Brücker Fast Scan no modo de toque. Todas as experiências de AFM foram realizadas utilizando sondas Olympus. As imagens obtidas por este protocolo são exibidas na Fig. 4 e filmes complementares 1-4.
Transformação morfológica isotérmica
Veja a Fig. 5 e Figs Suplementares. 21-23. Origamis de retângulo de DNA, sem ou com grampos encurtados, foram primeiro preparados por recozimento térmico: após montagem de 1 nM do modelo M13 com uma mistura de grampos (40 nM cada) em tampão TANa (com NaCl 100 mM ou 150 mM), o a amostra foi incubada por 10 min a 90 °C e depois submetida a uma rampa térmica em um termociclador peqSTAR 2X (Peqlab) de 70 °C a 20 °C a uma taxa de -0.1 °C por 2 min. Os grampos triangulares foram então misturados diretamente à amostra, sem qualquer purificação (os grampos retangulares são mantidos no sistema), com uma pipeta até a concentração desejada (10 ou 100 nM de cada grampo) com uma concentração final de 0.25 nM em [M13] e 10 nM em cada grampo retangular. A amostra resultante foi deixada incubar a 25 °C ou 30 °C sem mistura adicional.
Estatística e reprodutibilidade
Com exceção do experimento mostrado na Fig. 4, que foi realizado apenas uma vez devido à complexidade da configuração, todas as outras investigações foram replicadas várias vezes para garantir a reprodutibilidade. Todas as análises de imagens AFM foram realizadas em um grande número n de origamis individuais retirados de diferentes imagens, em diferentes posições de amostras obtidas nas mesmas condições. Todos os origamis detectados foram analisados. Nenhum origami, devidamente dobrado ou não, foi excluído destas análises. O número n de objetos analisados em cada condição mostrada nas diferentes figuras é exibida em Tabelas Suplementares 1-5. Nenhum método estatístico foi utilizado para predeterminar o tamanho da amostra. Nenhum dado foi excluído das análises. Os experimentos não foram randomizados. Os investigadores não ficaram cegos quanto à alocação durante os experimentos e avaliação dos resultados.
- Conteúdo com tecnologia de SEO e distribuição de relações públicas. Seja amplificado hoje.
- PlatoData.Network Gerativa Vertical Ai. Capacite-se. Acesse aqui.
- PlatoAiStream. Inteligência Web3. Conhecimento Amplificado. Acesse aqui.
- PlatãoESG. Automotivo / EVs, Carbono Tecnologia Limpa, Energia, Ambiente, Solar, Gestão de resíduos. Acesse aqui.
- BlockOffsets. Modernizando a Propriedade de Compensação Ambiental. Acesse aqui.
- Fonte: https://www.nature.com/articles/s41565-023-01468-2
- :é
- :não
- :onde
- $UP
- 1
- 10
- 100
- 11
- 12
- 15%
- 2%
- 20
- 200
- 2014
- 2015
- 24
- 25
- 26
- 2D
- 30
- 3d
- 40
- 50
- 500
- 60
- 65
- 7
- 70
- 8
- 90
- a
- acelerar
- adicionado
- acrescentando
- Adição
- Adicional
- Depois de
- depois
- Todos os Produtos
- alocação
- quantidade
- an
- Apresentadora
- e
- qualquer
- aplicado
- SOMOS
- artigo
- montado
- Montagem
- avaliação
- At
- média
- fundo
- BANDA
- BE
- Porque
- ser
- abaixo
- BP
- brevemente
- amortecer
- by
- Câmera
- transportado
- célula
- Câmara
- mudado
- clique
- coquetel
- Codificação
- Coluna
- complexidade
- concentração
- condição
- condições
- consecutivo
- constante
- Recipiente
- Canto
- Correspondente
- cobertura
- Cortar
- cifra
- dados,
- denso
- depositado
- descrito
- desejado
- detectou
- diferente
- Distribuição
- diretamente
- exibido
- dna
- feito
- down
- secar
- dois
- durante
- e
- cada
- ed
- borda
- final
- Inglaterra
- garantir
- equipado
- Éter (ETH)
- Cada
- evolução
- Exceto
- excesso
- excluídos
- experimentar
- experimentos
- RÁPIDO
- Figo
- Figura
- figuras
- preenchida
- filtro
- final
- Primeiro nome
- fixado
- Flash
- seguido
- seguinte
- Escolha
- força
- formado
- Gratuito
- Frequência
- da
- mais distante
- Além disso
- GAS
- suave
- grau
- Verde
- Grade
- tinha
- de alta resolução
- Horizontal
- HTTPS
- i
- if
- imagem
- imagens
- Imagiologia
- in
- Incluindo
- aumentou
- incubado
- incubação
- incubadora
- Individual
- do estado inicial,
- inovadores
- dentro
- inspirado
- instrumentos
- Nível intermediário
- para dentro
- introduzido
- investigações
- Investigadores
- IT
- manteve
- escada
- grande
- camada
- esquerda
- LINK
- Líquido
- carregamento
- lote
- Baixo
- diminuir
- Manipulação
- Martin
- material
- Merck
- metal
- método
- métodos
- Mica
- microfone
- Microscópio
- Microscopia
- migrou
- migração
- minutos
- misturar
- misto
- Misturando
- mistura
- ML
- Moda
- Filmes
- em movimento
- nano
- nanotecnologia
- Natureza
- necessário
- Novo
- não
- número
- objetos
- observado
- obtido
- of
- Olimpo
- on
- uma vez
- ONE
- queridos
- só
- oposto
- ideal
- or
- Outros
- A Nossa
- Fora
- Resultado
- Acima de
- Oxford
- Papel
- parte
- Cavilha
- para
- realizada
- peça
- peças
- platão
- Inteligência de Dados Platão
- PlatãoData
- posição
- abertas
- preparação
- preparado
- evitar
- anterior
- anteriormente
- Prévio
- sonda
- processo
- processos
- produzir
- devidamente
- Proteína
- protocolo
- colocar
- Rampa
- Randomizado
- Taxa
- Cru
- alcançar
- redução
- remanescente
- remover
- Removido
- removendo
- repetido
- replicado
- Resolução
- ressonância
- respectivamente
- resultando
- Quarto
- LINHA
- corrida
- s
- seguro
- mesmo
- escalável
- Escala
- digitalização
- científico
- Segundo
- Vejo
- selecionando
- separado
- Conjuntos
- vários
- encurtado
- mostrando
- lado
- Sigma
- Tamanho
- Lentamente
- pequeno
- solução
- Soluções
- específico
- Spin
- Etapa
- estatístico
- Passos
- estoque
- Vertentes
- transmitir canais
- suficiente
- Suportado
- superfície
- .
- T
- T1
- tomado
- tocando
- Target
- modelo
- que
- A
- deles
- então
- Lá.
- térmico
- Este
- isto
- três
- tempo
- vezes
- tipo
- para
- ferramentas
- topo
- para
- transferido
- Transformação
- tratado
- tratamento
- dois
- tipo
- para
- usava
- utilização
- vário
- via
- Voltagem
- volume
- foi
- Água
- maneiras
- we
- BEM
- foram
- qual
- precisarão
- Limpar
- de
- sem
- trabalhar
- seria
- zefirnet