inkubasjon
Alle inkubasjonsprosesser ble utført i en ThermoMixer C (Eppendorf) bortsett fra tilleggsfigurer. 1 og 9 (Dry Bath FB15103 inkubator, Fisher Scientific) og for Fig. 1c og utfyllende fig. 3-6 (QuantStudio5, Applied Biosystems av Thermo Fisher Scientific).
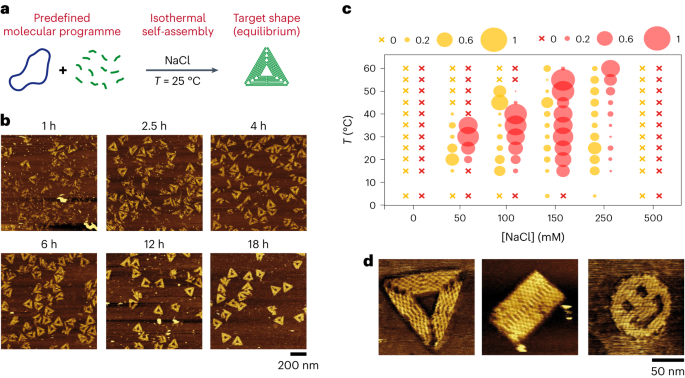
Isotermisk selvmontering av 2D DNA-origamier
Se fig. 1 og utfyllende fig. 1-9 og 11-14. Vi brukte stiftcocktailen uten noen termisk forbehandling og blandet den direkte med ønsket buffer før kort vortexing og tilsetning av M13-malen (1 nM) til løsningen og forsiktig opp-og-ned-blanding med en pipette. Løsningen ble latt inkubere, uten ytterligere blanding, ved en fast temperatur i ønsket tidsperiode.
Termisk annealing av DNA-origamier i TANa-buffer
Se tilleggsfig. 10. Vi satt sammen M13-malen (1 nM) med en blanding av stifter (40 nM hver stift) i TANa supplert med 100 mM NaCl. Prøven ble inkubert i 10 minutter ved 90 °C og deretter utsatt for en termisk rampe i en peqSTAR 2X termosykler (Peqlab) fra 70 °C til 20 °C med en hastighet på -1 °C per 10 minutter.
Rensing ved PEG-utfelling
Se tilleggsfigurer. 13 og 14. DNA-origamier oppnådd ved isotermisk montering i TANa-buffer ([NaCl] = 100 mM) ved 25 °C ble renset fra stifttrådene ved PEG-utfelling. Metoden er inspirert av protokollen introdusert i en tidligere rapport35. DNA-origamiene ble fortynnet tre ganger med en løsning av PEG 8000 og NaCl for å nå endelige konsentrasjoner på henholdsvis 4% w/v og 500 mM. Etter forsiktig blanding ble løsningen inkubert i 15 minutter ved romtemperatur og sentrifugert ved 15,000g i 15 min. Supernatanten ble fjernet og origamiene ble resuspendert til det opprinnelige volumet i TANa-buffer ([NaCl] = 100 mM). Om nødvendig ble prosessen gjentatt for en andre påfølgende rensing.
Gelelektroforese av renset origamis
Se tilleggsfig. 13. Vi tilberedte 50 ml agarose (type I lav EEO, Sigma Aldrich) gel ved 1.5 % inneholdende 4 μl GR-Green 10,000 1× (Excellgen) i TBE 18× buffer. Etter at gelen var avkjølt, introduserte vi, i hver brønn, 100 µl 18 bp DNA Ladder (New England Biolabs) eller 1 µl prøve supplert med 100 x DNA-ladningsfarge SDS-løsning (Thermo Scientific). Migreringen ble utført ved 1 V i 7 time i en 1 cm elektroforesecelle fylt med TBE XNUMX× buffer.
Isotermisk trinnvis montering
Se tilleggsfig. 24. Trekantens stifter ble satt sammen i tre separate partier, hver kodet for det øverste hjørnet, den mellomliggende delen og den motsatte kanten. Ett parti (40 nM hver stift) ble blandet med M13 (1 nM) i TANa-buffer ([NaCl] = 100 mM) og systemet ble inkubert ved 25 °C uten ytterligere blanding. Hver 24. time fjernet vi volumet som var nødvendig for AFM-avbildning, la til ett parti som koder for en ekstra del av trekanten (40 nM hver stift) og lot systemet inkubere ved 25 ° C i TANa-buffer ([NaCl] = 100 mM) . Vi utførte to forskjellige måter for trinnvis montering, fra hjørnet til motsatt side og fra den ene siden til motsatt hjørne.
Isotermisk fremstilling av streptavidin-modifiserte trekanter
Se fig. 2a og utfyllende fig. 15 og 16. I det samme røret blandet vi 1 nM M13, 40 nM av hver av stiftene inkludert biotinylerte, og 2 µM streptavidin i TANa-buffer supplert med 100 mM NaCl. Prøven ble inkubert ved 25 °C uten ytterligere blanding i 24 timer.
Isotermisk fremstilling av SST R4 rektangler
Se fig. 2b. Vi blandet alle tråder av R4-rektangelet i bufferen til en sluttkonsentrasjon på 100 nM i hver tråd i TANa supplert med 100 mM NaCl. Prøven ble inkubert ved 25 °C uten ytterligere blanding i 24 timer.
Gelelektroforese av SST R4 rektangler
Se fig. 2b. En 1.5 % agarosegel (type I lav EEO, Sigma Aldrich) ble fremstilt i TBE 0.5× buffer supplert med 11 mM MgCl2 og GB grønn DNA-farge. Gelelektroforese ble utført i et isvannbad i 2 timer ved 100 V spenning ved bruk av en 1 kb DNA-stige. For rensing ble målbåndet til gelen kuttet i små biter og satt inn i et rør med en spinnkolonne, og kolonnen ble utsatt for sentrifugering ved 5,000g i 10 min. For AFM-avbildning ble eluert prøve direkte adsorbert på en glimmerplate i 10 minutter inne i det atmosfærekontrollerte kammeret. Prøven ble deretter skylt med 1 ml i 0.5× TBE + 11 mM MgCl2 og observert ved bruk av AFM i 0.5 × TBE + 11 mM MgCl2.
Isotermisk fremstilling av DNA-nanonett
Se fig. 2c og tilleggsfig. 17. Vi blandet de ni oligonukleotidene (1 µM av hvert nukleotid) i TANa-buffer supplert med 100 mM eller 150 mM NaCl. Prøven ble inkubert ved 25 °C uten ytterligere blanding i 24 timer.
Termisk utglødning av 3D-origamier
Se fig. 3a og tilleggsfig. 18. Stillaset (7,560 13 nt M8,064 for Tb, 13 1 nt M10 for T5) og stiftblandingen (8.0 x overskudd i hver stift) ble blandet i buffer som inneholdt 1 mM Tris-HCl, pH 18, XNUMX mM EDTA og XNUMX mM MgCl2. Blandingen ble oppvarmet til 65 ° C i 15 minutter for å denaturere alle DNA-tråder før den ble sakte avkjølt i en gradient fra 60 ° C til 40 ° C, over 41 timer for å anneale og sette sammen 3D origami nanostrukturene.
Negativ-flekk TEM
Se fig. 3 og tilleggsfig. 18. For TEM-karakterisering ble DNA-nanostrukturer først renset fra 1 % agarose (0.5 × TBE, 45 mM Tris-borat, 1 mM EDTA, pH 8.3) supplert med 11 mM MgCl2 og 0.5 mg ml-1 Sybr TRYGT. Prøver ble migrert på gelen i 3 timer med en løpende buffer på 0.5 × TBE, 11 mM MgCl2 ved 2.85 V cm-1 i romtemperatur. Bånd som tilsvarer selvmonterte strukturer ble skåret ut og overført til en DNA-gelekstraksjonsspinkolonne (Merck) og sentrifugert ved 5,000 g i 5 minutter ved 4 °C. Rensede origamier ble deretter avsatt ved adsorpsjon på glødutladet karbonbelagt gitter (Quantifoil Micro Tools), farget i 60 s med en 2 % (vekt/volum) vandig uranylacetat (Merck) løsning og deretter tørket med askefritt filterpapir (VWR) ). TEM-observasjoner ble utført på et JEM-1400 Flash Tungsten-mikroskop som arbeidet ved 120 kV, utstyrt med et Gatan OneView-kamera.
Isotermisk konkurranse
Se tilleggsfig. 20. Vi virvlet kort blandingene av to stiftesett som koder for trekanter og rektangler (40 nM sluttkonsentrasjon for hver stift) med bufferen (TANa supplert med 100 mM NaCl) før vi tilsatte M13 til blandingen (1 nM sluttkonsentrasjon) og forsiktig blanding med en pipette. Prøven ble inkubert ved 25 °C uten ytterligere blanding.
AFM-observasjon av DNA-nanostrukturer i væske
Miljømessige høyoppløselige AFM-observasjoner i prøvebuffer ble brukt for alle AFM-data og bilder vist i denne artikkelen. Bortsett fra fig. 4 (se den spesifikke protokollen nedenfor), DNA-nanostrukturer oppnådd i TANa-buffer (DNA-origamis med eller uten proteinmodifikasjon, SST R4-rektangler, DNA-nanogrids) ble adsorbert på nyspaltede 10 mm-diameter glimmerskiver (Nano-Tec V-1 klasse Muscovite, Micro to Nano Innovative Microscopy Supplies) tidligere limt til en metallskive og behandlet med 20 µl av en spermintetrakloridløsning (0.1 M i MilliQ-vann) i 10 minutter og vasket rikelig, først med MilliQ-vann og deretter med TANa-bufferen. For prøveadsorpsjon ble 15–20 µl prøve avsatt på den ferske sperminbehandlede glimmeren og latt adsorbere i 10 minutter, bortsett fra isotermiske transformasjonseksperimenter (fig. 5 og utfyllende fig. 21-23) hvor tiden ble økt til 20 minutter på grunn av den lavere konsentrasjonen av origamis. Glimmerplaten ble deretter forsiktig skylt med 200 ul av bufferen for å fjerne overskudd av stifter og ikke-adsorberte gjenstander. For å forhindre tørking av prøven under glimmermanipulering, la vi et tynt lag med buffer på toppen av den adsorberte prøven og holdt den ved romtemperatur i et atmosfærekontrollert kammer (en forseglet beholder som inneholder et stykke Kimtech-serviett fuktet med MilliQ vann). Den samme protokollen ble utført med prøver som inneholdt magnesium (tillegg Fig. 1, TAEMg og TAMg-buffere) bortsett fra at prøvene ble direkte adsorbert i 5 minutter på den nyspaltede glimmeren uten noen behandling. Prøvene ble observert med et Cypher ES atomkraftmikroskop (Oxford Instruments) i tappemodus med 17–45 kHz resonansfrekvens i væske og en 0.09 Nm-1 kraft konstant spiss (BL-AC40TS, Olympus), ved bruk av blueDrive fototermisk eksitasjonsmodus. Råbilder ble utsatt for polynomisk bakgrunnssubtraksjon, plannivåkorreksjon, radjustering ved bruk av forskjellige metoder og horisontal arrkorrigering i Gwyddion.
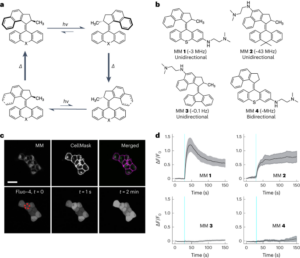
Sanntidsavbildning av den Λ → Δ isotermiske evolusjonen på en lipid-dobbeltlagsoverflate
Se fig. 4, Tilleggstekst 3 og tilleggsfilmer 1-4. Støttede lipid-dobbeltlag (SLB) ble oppnådd fra DOPC-liposomer via vår tidligere beskrevne metode37. Vesikler ble fremstilt fra en kloroformstam av DOPC. Etter fordampning av kloroformen under en strøm av nitrogengass, ble lipidene rehydrert i MilliQ vann for å oppnå en endelig lipidkonsentrasjon på 2 mg ml-1. Lipidblandingen ble deretter vortexet og sonikert i 60 minutter for å produsere små unilamellære vesikler. For å forhindre tørking av dobbeltlagene ble følgende trinn utført i et atmosfærekontrollert kammer. SLB-er ble dannet ved å deponere 2 ml av vesikkelløsningen på nyspaltede glimmerskiver (tidligere festet på en magnetisk metallplate med lim), etterfulgt av 2 µl TAEMg (Tris-acetat 1×, [EDTA] = 1 mM, [MgCl2] = 12.5 mM). Etter 30 minutter med adsorpsjon ble prøven skylt med 2 ul TAEMg-buffer for å fjerne uadsorberte liposomer, og denne adsorpsjonsprosessen ble gjentatt en gang til for å sikre optimal dekning av glimmeroverflaten av dobbeltlaget. På slutten av adsorpsjonsprosessen ble prøven skylt med 5 µl TAENa (Tris-acetat 1×, [EDTA] = 1 mM, [NaCl] = 100 mM) for å sikre at det ikke vil være gjenværende fritt Mg2+ ioner på prøven, noe som ville forhindre isotermisk folding av origamiene.
Kolesterol-modifisert Λ-formet origamis (tillegg Fig. 19) ble fremstilt ved å blande en løsning av 10 nM M13 i TAENa-bufferen med 20 nM av hver av stiftene og gløde ved å redusere temperaturen fra 70 °C til 4 °C med en hastighet på -0.1 °C min.-1. Den resulterende origami-løsningen ble brukt uten ytterligere rensing. Deretter ble 2 µl av den kolesterolmodifiserte Λ origamis-løsningen avsatt på den forhåndsformede SLB, etterfulgt av 2 µl TAENa-buffer. Prøven ble inkubert i 60 minutter ved romtemperatur i det atmosfærekontrollerte kammeret, og overflaten ble deretter direkte avbildet ved romtemperatur (T = 26 ° C) av AFM i 20 µl TAENa-buffer uten overflateskylling. Etter å ha valgt en posisjon som inneholder et tilstrekkelig antall Λ-origamier adsorbert på SLB, ble 8 µl av stiftene på A-siden i TAENa tilsatt prøven uten å flytte den eller fjerne den fra AFM-stadiet. Løsningen som overhengte prøven ble deretter forsiktig blandet ved sakte å bevege AFM-sonden opp og ned flere ganger for å akselerere diffusjonen på A-sidestiftene mot glimmeroverflaten. Den samme posisjonen ble deretter skannet i gjennomsnitt hvert 3. min i 223 min. Bildeoppløsningen var 512 × 512 fra t = 0 til t = 41 min og ble deretter endret til 640 × 640. The z skalaen til bildene var 0–10 nm fra t = 0 til t = 47 min og 0–7 nm etterpå. På grunn av fordampningen under avbildningsprosessen ble tilleggsbuffer (8 µl) tilsatt kl t = 63 min, t = 144 min og t = 161 min, og 8 ul av stiftene på A-siden ble videre tilsatt etter t = 170 min.
AFM-bilder ble oppnådd i TAENa ved romtemperatur ved bruk av et Brücker Fast Scan atomkraftmikroskop i tappemodus. Alle AFM-eksperimenter ble utført ved bruk av Olympus-sonder. Bilder hentet med denne protokollen vises i fig. 4 og tilleggsfilmer 1-4.
Isotermisk morfologisk transformasjon
Se fig. 5 og utfyllende fig. 21-23. DNA rektangel origamis, uten eller med forkortede stifter, ble først fremstilt ved termisk annealing: etter montering av 1 nM av M13-malen med en blanding av stifter (40 nM hver) i TANa-buffer (med 100 mM eller 150 mM NaCl), prøven ble inkubert i 10 minutter ved 90 °C og deretter utsatt for en termisk rampe i en peqSTAR 2X termosykler (Peqlab) fra 70 °C til 20 °C med en hastighet på -0.1 °C per 2 minutter. Trekantstifter ble deretter blandet direkte til prøven, uten noen rensing (rektangelstifter beholdes i systemet), med en pipette til ønsket konsentrasjon (10 eller 100 nM av hver stift) med en sluttkonsentrasjon på 0.25 nM i [M13] og 10 nM i hver rektangelstift. Den resulterende prøven ble inkubert ved 25 °C eller 30 °C uten ytterligere blanding.
Statistikk og reproduserbarhet
Bortsett fra eksperimentet vist i fig. 4, som bare ble utført én gang på grunn av kompleksiteten i oppsettet, ble alle andre undersøkelser replikert flere ganger for å sikre reproduserbarhet. Alle AFM-bildeanalysene ble utført på et stort antall n av individuelle origamier tatt fra forskjellige bilder, ved forskjellige posisjoner av prøver tatt under de samme forholdene. Alle påviste origamier ble analysert. Ingen origami, riktig brettet eller ikke, ble ekskludert fra disse analysene. Antallet n av analyserte objekter i hver tilstand vist i de forskjellige figurene vises i tilleggstabeller 1-5. Ingen statistisk metode ble brukt for å forhåndsbestemme prøvestørrelse. Ingen data ble ekskludert fra analysene. Eksperimentene ble ikke randomisert. Undersøkerne ble ikke blindet for tildeling under eksperimenter og resultatvurdering.
- SEO-drevet innhold og PR-distribusjon. Bli forsterket i dag.
- PlatoData.Network Vertical Generative Ai. Styrk deg selv. Tilgang her.
- PlatoAiStream. Web3 Intelligence. Kunnskap forsterket. Tilgang her.
- PlatoESG. Bil / elbiler, Karbon, CleanTech, Energi, Miljø, Solenergi, Avfallshåndtering. Tilgang her.
- BlockOffsets. Modernisering av eierskap for miljøkompensasjon. Tilgang her.
- kilde: https://www.nature.com/articles/s41565-023-01468-2
- :er
- :ikke
- :hvor
- $OPP
- 1
- 10
- 100
- 11
- 12
- 15%
- 2%
- 20
- 200
- 2014
- 2015
- 24
- 25
- 26
- 2D
- 30
- 3d
- 40
- 50
- 500
- 60
- 65
- 7
- 70
- 8
- 90
- a
- akselerere
- la til
- legge
- tillegg
- Ytterligere
- Etter
- etterpå
- Alle
- allokering
- beløp
- an
- Anchor
- og
- noen
- anvendt
- ER
- Artikkel
- montert
- Montering
- evaluering
- At
- gjennomsnittlig
- bakgrunn
- BAND
- BE
- fordi
- være
- under
- BP
- kort
- buffer
- by
- rom
- gjennomført
- celle
- Chamber
- endret
- klikk
- cocktail
- Koding
- Kolonne
- kompleksitet
- konsentrasjon
- tilstand
- forhold
- påfølgende
- konstant
- Container
- Corner
- Tilsvarende
- dekning
- Kutt
- cypher
- dato
- tett
- deponert
- beskrevet
- ønsket
- oppdaget
- forskjellig
- kringkasting
- direkte
- vises
- dna
- gjort
- ned
- tørk
- to
- under
- e
- hver enkelt
- ed
- Edge
- slutt
- England
- sikre
- utstyrt
- Eter (ETH)
- Hver
- evolusjon
- Unntatt
- overskytende
- ekskludert
- eksperiment
- eksperimenter
- FAST
- Fiken
- Figur
- tall
- fylt
- filtrere
- slutt~~POS=TRUNC
- Først
- fikset
- Blitz
- fulgt
- etter
- Til
- Tving
- dannet
- Gratis
- Frekvens
- fra
- videre
- Dess
- GAS
- skånsom
- klasse
- Grønn
- Grid
- HAD
- høy oppløsning
- Horisontal
- HTTPS
- i
- if
- bilde
- bilder
- Imaging
- in
- Inkludert
- økt
- inkubert
- INKUBASJON
- inkubator
- individuelt
- innledende
- innovative
- innsiden
- inspirert
- instrumenter
- Mellom
- inn
- introdusert
- Undersøkelser
- etterforskerne
- IT
- holdt
- stigen
- stor
- lag
- venstre
- LINK
- Flytende
- lasting
- Lot
- Lav
- lavere
- Manipulasjon
- Martin
- materiale
- Merck
- metall
- metode
- metoder
- MiCA
- micro
- Mikroskop
- mikros~~POS=TRUNC
- migrert
- migrasjon
- minutter
- bland
- blandet
- Blanding
- blanding
- ML
- Mote
- Filmer
- flytting
- nano
- nanoteknologi
- Natur
- nødvendig
- Ny
- Nei.
- Antall
- gjenstander
- observerte
- innhentet
- of
- olympus
- on
- gang
- ONE
- seg
- bare
- motsatt
- optimal
- or
- Annen
- vår
- ut
- Utfallet
- enn
- Oxford
- Papir
- del
- Peg
- for
- utført
- brikke
- stykker
- plato
- Platon Data Intelligence
- PlatonData
- posisjon
- stillinger
- forberedelse
- forberedt
- forebygge
- forrige
- tidligere
- Før
- probe
- prosess
- Prosesser
- produsere
- riktig
- Protein
- protokollen
- sette
- Rampe
- randomisert
- Sats
- Raw
- å nå
- redusere
- gjenværende
- fjerne
- fjernet
- fjerne
- gjentatt
- replikert
- oppløsning
- resonans
- henholdsvis
- resulterende
- rom
- RAD
- rennende
- s
- trygge
- samme
- skalerbar
- Skala
- skanne
- vitenskapelig
- Sekund
- se
- velge
- separat
- sett
- flere
- forkortet
- vist
- side
- Sigma
- Størrelse
- Sakte
- liten
- løsning
- Solutions
- spesifikk
- Snurre rundt
- Scene
- statistisk
- Steps
- lager
- Tråder
- stream
- tilstrekkelig
- Støttes
- overflaten
- system
- T
- T1
- tatt
- tappe
- Target
- mal
- Det
- De
- deres
- deretter
- Der.
- termisk
- Disse
- denne
- tre
- tid
- ganger
- typen
- til
- verktøy
- topp
- mot
- overføres
- Transformation
- behandlet
- behandling
- to
- typen
- etter
- brukt
- ved hjelp av
- ulike
- av
- Spenning
- volum
- var
- Vann
- måter
- we
- VI VIL
- var
- hvilken
- vil
- tørke
- med
- uten
- arbeid
- ville
- zephyrnet