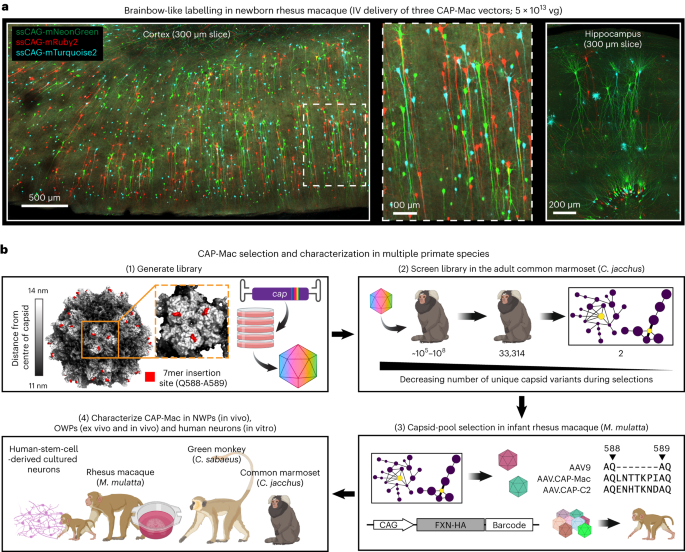
모든 실험과 절차는 현지 규제 위원회 및 위원회의 승인을 받았으며 연구 프로토콜을 준수해야 했습니다. 모든 마우스 절차는 Caltech 기관 동물 관리 및 사용 위원회(IACUC, 프로토콜 1738)의 승인을 받은 Caltech에서 수행되었습니다. 마모셋(프로토콜 TGC-03) 및 성체 원숭이(프로토콜 LN-14) 절차는 NIH에서 이루어졌으며 NIH IACUC의 승인을 받았습니다. Marmoset 절차는 캘리포니아 샌디에이고 대학교(UCSD)(프로토콜 S09147)에서도 완료되었으며 UCSD IACUC를 준수하고 승인되었습니다. 유아 원숭이 절차는 캘리포니아 대학교 데이비스 캠퍼스의 캘리포니아 국립 영장류 연구 센터에서 이루어졌으며 지역 IACUC(프로토콜 22525)의 승인을 받았습니다. Green Monkey 절차는 Virscio에서 이루어졌으며 지역 IACUC의 승인을 받았습니다.
AAV DNA 라이브러리 생성
이 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.5jyl8jy89g2w/v1). 우리는 처음에 DNA 수준에서 다양성을 생성한 다음 이전에 자세히 설명한 대로 AAV 캡시드 라이브러리를 생성하기 위한 형질감염 물질을 생성하는 데 사용했습니다.16. 588차 라이브러리의 경우, 우리는 아미노산 589과 XNUMX 사이에 삽입된 축퇴 뉴클레오티드를 포함하는 프라이머를 사용하여 이러한 유전적 다양성을 도입했습니다. 12,13,16) (VP1 번호 매기기; 보충 그림. 1a). 우리는 7 축퇴 뉴클레오티드([NNK] × 7)를 포함하는 역방향 프라이머를 사용하여 독특한 7mer 서열을 포함하는 중합효소 연쇄 반응(PCR) 단편을 무작위로 생성했습니다. 캡 유전자. 66,628차 DNA 라이브러리의 경우 합성 올리고 풀(Twist Bioscience)을 역방향 프라이머로 사용하여 추가 스크리닝을 위해 선택된 변이체만 인코딩했습니다(총 33,314 DNA 올리고, 20차 선택 후 회수된 5 변이체 및 코돈 변형 각각 복제). 모든 역방향 프라이머에는 상보적인 XNUMXbp XNUMX' 오버행이 포함되어 있습니다. 캡 AgeI 제한 효소 서열 근처의 서열과 XbaI 제한 효소 서열 근처의 20bp 5' 오버행을 포함하는 정방향 프라이머와 짝을 이루었습니다. 그런 다음 다양한 영역을 포함하는 PCR 단편을 Gibson 어셈블리를 통해 rAAV-ΔCAP-in-cis-Lox 플라스미드에 삽입하여 NEBuilder HiFi DNA 어셈블리를 사용하여 결과 AAV DNA 라이브러리, 즉 rAAV-CAP-in-cis-Lox를 생성했습니다. 마스터 믹스(New England Biolabs, E2621).
AAV 캡시드 라이브러리 제작
이 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.5jyl8jyz9g2w/v1). 이전에 게시된 프로토콜에 따라 AAV 캡시드 라이브러리를 생성했습니다.16,70. 간단히 말하면, 우리는 형질감염 등급 선형 폴리에틸렌이민(PEI, Polysciences)을 사용하여 293mm 조직 배양 플레이트에서 HEK3216T 세포(ATCC, cat # CRL-0063, RRID: CVCL_150)를 형질감염시켰습니다. 각 플레이트에서 우리는 1개의 플라스미드를 형질감염시켰습니다: (2) AAV 캡슐화에 필요한 역전된 말단 반복이 옆에 있는 조립된 rAAV-Cap-in-cis-Lox AAV DNA 라이브러리; (2) AAV9/XNUMX REP-AAP-ΔCAP, 이는 AAV 생산에 필요한 REP 및 AAP 보충 단백질을 코딩하며 C 말단을 갖습니다. 캡 AAV DNA 라이브러리와의 재조합 및 복제 가능 AAV의 후속 생산을 방지하기 위해 절제된 유전자; (3) AAV 생산에 필요한 필수 아데노바이러스 단백질을 코딩하는 pHelper; 및 (4) pUC18(Addgene ID: 50004; RRID: Addgene_50004)은 포유동물 발현 벡터를 포함하지 않지만 최적의 PEI 형질감염을 위해 적절한 질소-인산염 비율을 달성하기 위한 필러 DNA로 사용됩니다. PEI-DNA 혼합물을 준비하는 동안 우리는 10mm 접시마다 150ng의 AAV DNA 라이브러리(rAAV-Cap-in-cis-Lox)를 추가하고 AAV2/9 REP-AAP-ΔCAP, pUC18 및 pHelper를 한 접시에 결합했습니다. 1:1:2 비율(40 mm 접시당 총 DNA 150 μg). 형질감염 후 60시간에 폴리에틸렌 글리콜 침전 및 요오딕사놀 구배 초원심분리를 사용하여 세포 펠릿과 배지 모두에서 AAV 캡시드 라이브러리를 정제했습니다. 그런 다음 정량적 PCR을 사용하여 확립된 프로토콜에 따라 선형화된 게놈 표준에 비해 DNaseI 내성 바이러스 게놈을 증폭하여 AAV 캡시드 라이브러리의 역가를 결정했습니다.70.
마모셋 실험
Capsid 라이브러리 선택
이 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.bp2l695zklqe/v2). 모든 마모셋(C. 자커스) 절차는 국립정신건강연구소(NIMH)에서 수행되었으며 지역 IACUC의 승인을 받았습니다. 마모셋은 NIMH 식민지에서 태어나고 자랐으며 27°C 및 50% 습도의 표준 조건에서 가족 그룹으로 사육되었습니다. 그들은 NIH에서 NHP를 위한 영장류 농축 프로그램의 일환으로 자유롭게 먹이를 주고 농축을 받았습니다. 이 연구에 사용된 모든 마모셋에 대해 IV 주입 전 1:5 혈청 희석에서 검출 가능한 중화 항체가 없었습니다(펜실베니아 대학의 Penn Vector Core에 의해 분석됨). 그런 다음 그들은 며칠 동안 개별적으로 수용되었고 주사 전에 새로운 방에 적응되었습니다. 도서관 심사에는 2명의 성인 남성이 사용되었으며, 10차 및 XNUMX차 도서관에는 각각 XNUMX명이 사용되었습니다. 주입 전날, 동물의 사료를 제거했습니다. 동물을 산소 중의 이소플루란으로 마취시키고, 대퇴 정맥 위의 피부를 면도하고 이소프로판올 스크럽과 XNUMX × XNUMX12 AAV 캡시드 라이브러리의 vg를 몇 분에 걸쳐 주입했습니다. 마취를 중단하고 동물이 활성화될 때까지 모니터링한 후 다시 우리에 넣었습니다. 활동과 행동은 다음 3일 동안 면밀히 모니터링되었으며 그 이후에는 매일 관찰되었습니다.
주사 후 4주째에 마모셋을 안락사시키고(Euthanasia, VetOne) 1X 인산염 완충 식염수(PBS)를 관류했습니다. 2차 라이브러리 이후 뇌를 32631개의 관상 블록으로 절단하고 80-메틸부탄(Sigma-Aldrich, M80)에 급속 냉동한 다음 드라이아이스로 냉각하고 장기 보관을 위해 -XNUMX°C에 보관했습니다. XNUMX차 라이브러리 이후 뇌를 XNUMX개의 관상 블록으로 절단하고 척수 및 간 섹션과 함께 급속 냉동하여 장기 보관을 위해 -XNUMX°C에 보관했습니다.
마모셋에서 AAV의 개별 특성 분석
이 섹션의 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1 및 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1). 성체 커먼마모셋 두 마리(C. 자커스)를 이 실험에 사용했습니다: Conan(남성, 2.8세, 0.386 kg) 및 Sandy(여성, 5.8세, 0.468 kg)(보충표) 3 자세한 내용을 제공합니다). 그들은 음식과 물에 자유롭게 접근할 수 있는 27°C 및 50% 습도의 표준 조건에서 보관되었습니다. 모든 동물은 그룹 수용되었으며 실험은 UCSD의 피질 시스템 및 행동 실험실에서 수행되었습니다. 모든 실험은 UCSD IACUC의 승인을 받았습니다. 주입 전날, 동물의 사료를 제거했습니다.
동물을 케타민(Ketaset, Zoetis 043-304, 20 mg kg)으로 마취시켰습니다.-1), 복재 정맥 위의 피부를 면도하고 이소프로판올 스크럽과 2 × 1013 vgkg-1 AAV를 5분에 걸쳐 주입했습니다. 동물이 활성화될 때까지 모니터링한 후 다시 우리에 넣었습니다. 활동과 행동은 다음 3일 동안 면밀히 모니터링되었으며 그 이후에는 매일 관찰되었습니다. 혈장 내 바이러스 농도를 측정하기 위해 1, 7, 14, 21 및 31일에 혈액 샘플을 채취했습니다.
주사 후 31일째에 마모셋을 앞서 설명한 대로 케타민으로 마취한 후 안락사시켰습니다(Euthasol, Virbac 200-071, 1 ml kg).-1) 및 1× PBS로 관류하였다. 뇌와 장기를 반으로 자르고 반은 2-메틸부탄(Sigma-Aldrich, M32631)에 급속 냉동한 후 드라이아이스로 냉각하고 -80°C에 보관했습니다. 나머지 절반은 4% 파라포름알데히드(PFA)(Thermo Scientific, J19943-K2)에 밤새 고정한 후 PBS 아지드(Sigma-Aldrich, S4-2002G, 100%)에 넣어 0.025°C에서 보관했습니다. 그런 다음 샘플을 분석을 위해 캘리포니아 공과대학(Caltech)으로 배송했습니다. GLUT1 염색을 위해 우리는 토끼 항-GLUT1(1:200; Millipore-Sigma, cat # 07-1401; RRID: AB_1587074)로 슬라이스를 배양하고 PBS로 1~200회 세척을 수행하고 당나귀 항토끼 IgG(711: 605, Jackson ImmunoResearch Labs, 카탈로그 번호 152-2492288-0.1, RRID: AB_100) 장착 전 8787~10회 세척했습니다. 우리는 모든 항체를 희석하고 017% Triton X-000(Sigma-Aldrich, T121) 및 2337258% 정상 당나귀 혈청(Jackson ImmunoResearch Labs, cat # XNUMX-XNUMX-XNUMX; RRID: AB_XNUMX)이 보충된 PBS를 사용하여 밤새 모든 인큐베이션을 수행했습니다. 흔들리는 실온.
바이러스 라이브러리 DNA 추출 및 NGS 샘플 준비
이 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.bp2l695zklqe/v2). 우리는 이전에 TRIzol을 사용하여 수성상에서 핵산을 침전시켜 바이러스 라이브러리 DNA와 내인성 숙주 RNA를 분리할 수 있다고 보고했습니다.12,16. 따라서 마모셋 조직에서 바이러스 라이브러리 DNA를 추출하기 위해 BeadBug(Benchmark Scientific, D100)를 사용하여 TRIzol(Life Technologies, 15596)에서 척수, 간 및 뇌의 각 관상 블록 1036mg을 균질화하고 핵산을 분리했습니다. 제조업체가 권장하는 프로토콜에 따라 수성상을 처리합니다. 우리는 재구성된 침전물을 RNase(Invitrogen, AM2288)로 처리하고 SmaI로 분해하여 PCR을 통한 하류 바이러스 DNA 회수를 개선했습니다. 소화 후 제조업체의 권장 프로토콜에 따라 Zymo DNA Clean and Concentrator 키트(D4033)로 정제하고 정제된 바이러스 DNA를 -20°C에 보관했습니다.
다양한 영역 측면에 Illumina 어댑터를 추가하기 위해 먼저 추출된 총 바이러스 DNA의 7%를 주형(50사이클)으로 사용하여 25mer 삽입이 포함된 영역을 PCR 증폭했습니다. Zymo DNA 정제 후 샘플을 1:100으로 희석하고 7600주기의 PCR을 통해 가변 영역 주변을 추가로 증폭시켜 다음 PCR 반응을 위한 결합 영역을 추가했습니다. 마지막으로 2회 이상의 PCR 주기를 통해 NEBNext Dual Index Primers(New England Biolabs, E16520050)를 사용하여 Illumina 플로우 셀 어댑터와 고유 인덱스를 추가했습니다. 그런 다음 210% 저융점 아가로스 겔(Thermo Fisher Scientific, XNUMX)을 사용하여 최종 PCR 산물을 겔 정제하고 XNUMXbp 밴드를 회수했습니다.
XNUMX차 라이브러리의 경우에만 NGS용 캡슐화된 AAV 라이브러리 ssDNA를 분리하여 라이브러리 농축 점수를 계산했습니다. 이는 라이브러리의 다양한 변이체 역가의 차이를 정규화하는 데 사용한 정량적 측정 기준입니다(참고 문헌 참조). 16 및 'NGS 판독 정렬, 분석 및 네트워크 그래프 생성' 섹션). 캡슐화 된 바이러스 게놈을 분리하기 위해 AAV 캡시드 라이브러리를 DNaseI로 처리하고 단백질 분해 효소 K를 사용하여 캡시드를 소화했습니다. 그런 다음 페놀-클로로포름을 사용하여 ssDNA를 정제하고 두 가지 PCR 증폭 단계로 바이러스 도입 유전자를 증폭하여 Illumina NGS에 대한 어댑터와 인덱스를 추가하고 정제했습니다. 겔 전기영동을 이용하여 이 바이러스 라이브러리 DNA는 조직에서 추출한 바이러스 DNA와 함께 Illumina HiSeq 2500 시스템(Millard and Muriel Jacobs Genetics and Genomics Laboratory, Caltech)을 사용하여 심층 시퀀싱을 위해 전송되었습니다.
NGS 읽기 정렬, 분석 및 네트워크 그래프 생성
NGS 실행의 원시 FASTQ 파일은 맞춤형 스크립트(https://github.com/GradinaruLab/protfarm 및 https://github.com/GradinaruLab/mCREATE)16. 33,314차 라이브러리의 경우 이러한 데이터 세트를 처리하는 파이프라인에는 품질이 낮은 읽기를 제거하기 위한 필터링, 각 시퀀스에 대한 품질 점수 활용, PCR로 유발된 돌연변이 또는 높은 GC 함량으로 인한 편향 제거가 포함되었습니다. 그런 다음 필터링된 데이터세트를 완벽한 문자열 일치 알고리즘으로 정렬하고 다듬어 정렬 품질을 향상시켰습니다. 그런 다음 각 조직 내에서 시퀀싱을 실행하는 동안 각 변종에 대한 절대 판독 횟수를 표시했으며, 뇌에서 발견된 모든 XNUMX개의 변이가 XNUMX차 선택을 위해 선택되었습니다.
두 번째 라운드 선택 후 동일한 분석을 수행하여 주입된 바이러스 라이브러리와 각 조직 내의 각 변이체의 변이체 절대 판독 수를 표시했습니다. 또한, 우리는 라이브러리 농축을 계산했습니다.16 각 조직 내의 각 변종에 대해:
$${overline{{rm{RC}}}}_{x,{rm{주입}},{rm{라이브러리}}}=,frac{{{rm{RC}}}_{x,{rm{ 주입}},{rm{라이브러리}}}}{mathop{sum }nolimits_{i=1}^{{N}_{{rm{삽입}},{rm{라이브러리}}}}{{rm{RC }}}_{i,{rm{주입},{라이브러리}}}},$$
(1)
$${overline{{rm{RC}}}}_{x,{rm{조직}}}=,frac{{{rm{RC}}}_{x,{rm{바이러스}}}}{mathop {sum }nolimits_{i=1}^{{N}_{{rm{조직}}}}{{rm{RC}}}_{i,{rm{조직}}}},$$
(2)
$${rm{라이브러리},{보강}}=,{로그 }_{10}left(frac{{overline{{rm{RC}}}}_{x,rm{{주입},{라이브러리}} }}{{overline{{rm{RC}}}}_{x,{rm{조직}}}}right),$$
(3)
주어진 샘플에 대해 y (예: 주입된 바이러스 라이브러리 또는 조직 샘플), RCx,y 변종의 절대 읽기 횟수입니다. x, Ny 복구된 변종의 총 개수입니다. ({윗줄{{rm{RC}}}}_{x,{y}}) 정규화된 읽기 횟수입니다.
CAP-Mac 시퀀스 클러스터링 그래프를 구성하기 위해 다음 기준에 따라 1차 NGS 데이터를 필터링했습니다. (100) 주입된 라이브러리 샘플의 읽기 수 ≥24,186(33,314/2 변종), (0.7) ≥415 라이브러리 농축 3개 이상의 뇌 샘플(0.7 변종)에서 점수를 매기고 (0.7) 라이브러리 농축이 -323 미만인 뇌 샘플(2 변종)보다 라이브러리 농축이 ≥1인 뇌 샘플이 100개 이상 더 많습니다. CAP-C2 시퀀스 그래프를 구성하기 위해 우리는 다음 기준에 따라 0.7차 NGS 데이터를 필터링했습니다. (95) 주입된 라이브러리 샘플의 읽기 수 ≥XNUMX 및 (XNUMX) 두 개 이상의 뇌 샘플에 존재하는 두 코돈 복제물 ≥XNUMX 라이브러리 강화(XNUMX 변종). 그런 다음 이러한 변형을 독립적으로 처리하여 쌍별 역방향 해밍 거리(https://github.com/GradinaruLab/mCREATE) 및 이전에 자세히 설명한 대로 Cytoscape(v. 3.9.0; RRID: SCR_003032)를 사용하여 클러스터링되었습니다.16. 제시된 네트워크는 쌍별 역방향 해밍 거리가 ≥3인 경우 가장자리로 연결된 캡시드 변형(노드)을 보여줍니다.
개별 AAV 캡시드 변종 복제
이 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.n2bvj87ebgk5/v1). 단일 변형 특성화를 위해 MscI 및 AgeI를 사용하여 pUCmini-iCAP-PHP.eB(Addgene ID: 103005; RRID: Addgene_103005) 백본의 수정된 버전을 소화하여 새로운 변형 플라스미드를 복제했습니다. 우리는 각 캡시드 변이체에 대해 원하는 100bp 삽입과 소화된 백본과 겹치는 ~21bp의 AAV9 템플릿에 상보적인 영역을 포함하는 20bp 프라이머를 설계했습니다. 그런 다음 NEBuilder HiFi DNA Assembly Master Mix를 사용하여 변종 플라스미드를 조립하고 반응 혼합물에서 5μl의 200nM 프라이머와 30ng의 소화된 백본을 결합했습니다. AAV.CAP-Mac을 생산하는 데 사용되는 캡시드 플라스미드는 Addgene(Addgene ID: 200658; RRID: Addgene_200658)에서 구할 수 있습니다.
개별 AAV 생산 및 정제
이 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.14egn2dqzg5d/v1). 풀 테스트를 위한 변형을 생성하기 위해 이전에 게시된 프로토콜을 따랐습니다.70 150 mm 조직 배양 접시를 사용합니다. 생체 내 및 시험관 내 개별 AAV.CAP-Mac 및 AAV9 특성화를 위해 우리는 공개된 프로토콜을 채택하여 3320층 CellSTACK(Corning, 150)을 활용하여 붉은 털 원숭이와 녹색 원숭이를 투여하기 위해 높은 역가에서 바이러스를 효율적으로 생산했습니다. 구체적으로, 우리는 형질감염 70시간 전에 24층 CellSTACK에 약 150% 컨플루언시로 72개의 150mm 접시를 통과시켰습니다. 형질감염 당일에 우리는 120개의 15575020mm 접시에 대한 PEI-DNA 형질감염 혼합물을 준비하고 형질감염 혼합물을 배지와 결합하고 CellSTACK에 대한 완전한 배지 교체를 수행했습니다. 우리는 10mm 접시에서 생산하는 것과 유사하게 형질감염 후 37시간에 미디어를 수집하고 변경했습니다. 형질감염 후 20시간에 에틸렌디아민테트라아세트산(Invitrogen, XNUMX)을 최종 농도가 XNUMXmM이 되도록 첨가하고 XNUMX°C에서 XNUMX분 동안 배양했으며 때때로 CellSTACK의 측면을 휘젓고 두드려 세포를 분리했습니다. 그런 다음 배지와 세포 혼합물을 제거하고 AAV 정제 프로토콜을 진행했습니다.70. 참고로, 초원심분리 후 완충액 교환 단계에서는 Amicon 여과 장치 대신 폴리에테르술폰 막이 있는 원심분리 단백질 농축기(Thermo Scientific, 88533)를 사용하고 0.001% Pluronic F-68(Gibco, 24040032)이 보충된 Dulbecco의 PBS를 사용했습니다.
설치류 실험
모든 설치류 절차는 Caltech에서 수행되었으며 지역 IACUC의 승인을 받았습니다. 우리는 C57BL/6J(균주 번호: 000664; RRID: IMSR_JAX:000664), BALB/cJ(균주 번호: 000651; RRID: IMSR_JAX:000651) 및 DBA/2J(균주 번호: 000671; RRID: IMSR_JAX:000671) 마우스를 구입했습니다. (모두 수컷, 생후 6~8주) The Jackson Laboratory. 생쥐에게 IV 투여를 위해 우리는 5 × 1011 후안와동을 통한 바이러스의 vg70,71 31게이지 인슐린 주사기(BD, 328438)를 사용합니다. 마우스에서 AAV의 안와후 주사에 대한 자세한 내용은 프로토콜을 참조하세요(https://doi.org/10.17504/protocols.io.3byl4joy8lo5/v1). 쥐의 뇌실내(ICV) 투여를 위해 5.0 × 1010 또는 1.5×1011 vg를 측뇌실로 삽입합니다. 간단히 말해서, 우리는 5% O 이소플루란(유도용 1%, 유지용 3-95%)을 사용하여 마우스를 마취했습니다.2/ 5 % CO2 (1 XNUMX 분-1) 그리고 생쥐의 머리는 정위 프레임에 고정되었습니다. 머리를 면도하고 클로로헥시딘으로 소독한 후 0.05 mg ml 2.5 ml를 피하주사하였다.-1 부피바카인을 투여하고 정중선을 절개하고 두개골에서 혈액과 결합 조직을 제거했습니다. 머리를 수평으로 맞춘 후 측면 심실 위에 버 구멍을 양측으로 뚫었습니다(브레그마 후방 0.60mm, 정중선에서 1.15mm). 10 게이지 미세주사 바늘을 사용하여 바이러스 벡터를 33 μl NanoFil 주사기(World Precision Instruments)에 흡인하고 바늘을 측뇌실(연막 표면에서 1.6 mm)로 천천히 내렸습니다. 바늘을 약 5 분 동안 제자리에 놓아두고 마이크로 주사기 펌프(World Precision Instruments, UMP3) 및 펌프 컨트롤러(World Precision Instruments, Mircro5)를 사용하여 3 nl min의 속도로 3~300 μl의 바이러스 벡터를 주입했습니다.-1. 모든 쥐는 수술 중에 1 mg kg을 투여받았습니다.-1 부프레노르핀 SR 및 5 mg kg-1 케토프로펜 피하 투여 및 30 mg kg-1 이부프로펜 및 60 mg kg-1 수술 후 5일 동안 트리메토프림/설파메톡사졸을 투여합니다. 마우스에서 AAV의 ICV 주사에 대한 자세한 내용은 Protocols.io를 참조하십시오(https://doi.org/10.17504/protocols.io.5qpvorm4dv4o/v1). 발현 3주 후, 모든 마우스에 PBS를 관류하고 4% PFA에 고정시켰다. 모든 장기를 추출하고 4.00% PFA에서 밤새 배양한 후 0.01% 아지드화 나트륨이 보충된 PBS로 옮겨 장기 보관을 위해 4°C에 보관했습니다. 우리는 Prolong Diamond Antifade(Invitrogen, P100)에 장착된 vibratome(Leica Biosystems, VT1200S)을 사용하여 뇌를 36970μm 섹션으로 자르고 ZEN(Black edition)을 사용하여 공초점 현미경(Zeiss, LSM 880)을 사용하여 이미지화했습니다. 조직 취급에 대한 자세한 내용은 Protocols.io를 참조하십시오(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1 및 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1).
붉은털원숭이 실험
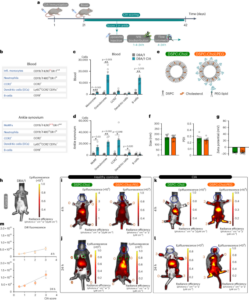
이 섹션의 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1). 갓 태어난 원숭이(0.45~1.40 kg)는 태어나자마자 젖을 뗐습니다. 첫 달 이내에 원숭이에게 AAV 벡터를 정맥 내 또는 척수강 내로 주입했습니다. 이 연구에 포함된 모든 성인 원숭이(8~17세, 4.65~11kg)에는 IV 투여를 통해서만 AAV가 주입되었습니다. IV 주사를 위해 동물을 케타민(0.10 ml)으로 마취시키고 복재 정맥 위의 피부를 면도하고 소독했습니다. AAV(2 × 10 사이13 및 1 × 1014 vgkg-1)를 PBS <1 ml에 ~0.75 분에 걸쳐 복재 정맥에 천천히 주입했습니다. ICM 주사의 경우, 동물에게 진정제를 근육내 투여하고 목 피부 부위를 면도하고 무균적으로 준비했습니다. 주입된 수액의 양에 비례하여 소량의 CSF를 제거하기 위해 바늘을 대수조 안으로 전진시켰습니다. 그런 다음 AAV(1.5 × 10)의 멸균 제제가 들어 있는 멸균 주사기를 사용합니다.12 또는 2.5×1013 vgkg-1) 채취한 수액의 양에 비례하여 무균적으로 부착하여 천천히 주입하였다. 모든 동물을 진정으로부터 회복하는 동안 하루 종일 모니터링한 다음 매일 부작용 결과를 모니터링했습니다. 모든 원숭이는 동종의 시야와 소리 내에 개별적으로 수용되었습니다. 주사 후 4~11주 후에 조직을 수집했습니다. 캘리포니아 국립 영장류 연구 센터(California National Primate Research Center)의 동물 인도적 안락사 지침에 따라 동물을 깊게 마취하고 펜토바르비탈 나트륨을 투여했습니다. 붉은털원숭이에 주입된 모든 물질에는 내독소가 없었습니다(<0.1 EU ml).-1), 단백질 순도는 나트륨 도데실 황산염-폴리아크릴아미드 겔 전기영동에 의해 확인되었습니다. 보충표 4 및 5 각 실험에 대한 투여 경로, AAV 변이체, 바이러스 용량, 유전적 화물 및 발현 기간을 나열합니다.
붉은 털 원숭이의 풀 테스트
이 섹션의 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1, https://doi.org/10.17504/protocols.io.3byl4jo68lo5/v1 및 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1). 신생아 원숭이 풀 실험(RM-001~RM-004)은 UC Davis의 CNPRC에서 수행되었으며 지역 IACUC의 승인을 받았습니다. 성인 원숭이 풀 실험(RMN-001 및 RMN-002)은 NIMH에서 수행되었으며 현지 IACUC의 승인을 받았습니다. 원숭이에게 얼음처럼 차가운 RNase가 없는 PBS를 주입했습니다. 관류 시 뇌의 한쪽 반구를 급속 냉동하고 다른 반구를 4mm 관상 블록으로 절단한 후 4% PFA에 48시간 동안 후고정한 후 추가 처리를 위해 Caltech로 옮겼습니다. HA 염색을 위해 우리는 토끼 항-HA(1:200; Cell Signaling Technology, cat # 3724; RRID: AB_1549585)로 슬라이스를 배양하고 PBS로 1~200회 세척을 수행하고 당나귀 항-토끼 IgG(711:605; Jackson ImmunoResearch Labs, 카탈로그 번호 152-2492288-0.1, RRID: AB_100) 장착 전 8787~10회 세척했습니다. 우리는 모든 항체를 희석하고 017% Triton X-000(Sigma-Aldrich, T121) 및 2337258% 정상 당나귀 혈청(Jackson ImmunoResearch Labs, cat # XNUMX-XNUMX-XNUMX; RRID: AB_XNUMX)이 보충된 PBS를 사용하여 모든 인큐베이션을 수행했습니다. 흔들어주면서 실온에서 밤새도록.
바이러스 DNA와 전체 RNA를 분리하기 위해 뇌와 간에서 100mg 조각을 BeadBug(Benchmark Scientific, D15596)를 사용하여 TRIzol(Life Technologies, 1036)에서 균질화하고 제조업체의 권장 프로토콜에 따라 전체 DNA와 RNA를 회수했습니다. . 회수된 DNA를 RNase로 처리하고 SmaI로 제한효소 분해한 후 Zymo DNA Clean and Concentrator 키트(D4033)로 정제했습니다. 회수된 RNA를 DNase로 처리하고 제조사의 권장 프로토콜에 따라 SuperScript III(Thermo Fisher Scientific, 18080093) 및 올리고(dT) 프라이머를 사용하여 mRNA로부터 cDNA를 생성했습니다. 우리는 50ng의 바이러스 DNA 또는 cDNA를 주형으로 사용하여 PCR을 사용하여 바코드 영역을 증폭했습니다. Zymo DNA 정제 후 샘플을 1:100으로 희석하고 Illumina NGS용 어댑터를 추가하기 위한 프라이머를 사용하여 바코드 영역을 추가로 증폭했습니다. 정리 후, 이들 생성물은 Illumina 시퀀싱용 NEBNext Dual Index Primers(New England Biolabs, E7600)를 사용하여 2주기 동안 추가로 증폭되었습니다. 그런 다음 16520050% 저융점 아가로스 겔(Thermo Fisher Scientific, XNUMX)을 사용하여 최종 PCR 산물을 겔 정제했습니다. 풀 테스트 강화는 라이브러리 강화와 동일하게 계산되었지만 그림 XNUMX에 표시되어 있습니다. 2b,c 선형 규모로.
붉은 털 원숭이에서 CAP-Mac의 개별 특성 분석
이 섹션의 절차에 대한 자세한 내용은 Protocols.io(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1 및 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1). 신생아 원숭이에게 PBS 및 4% PFA를 관류시켰습니다. 뇌를 4mm 관상 블록으로 절단하고 모든 조직을 PBS에 보관하기 전 4일 동안 3% PFA에 후고정했습니다. 생체 내 개별 특성 분석에 사용된 단일 성체 원숭이(RM-020; 17세, 11kg)에 RNase가 없는 PBS를 관류하고 반구의 한쪽을 급속 냉동하고 다른 반구를 4mm 관상 블록과 4% PFA로 후고정. 모든 조직은 추가 처리를 위해 Caltech로 옮겨졌습니다. 뇌와 간은 vibratome을 사용하여 100μm 조각으로 절단되었습니다. 추가로, 뇌 및 척수의 섹션을 30% 수크로스에서 밤새 배양하고 O.C.T.에 삽입했습니다. 화합물(Scigen, 4586)을 사용하고 저온 유지 장치(Leica Biosystems, CM50)를 사용하여 1950μm 슬라이스로 절단했습니다. 모든 슬라이스는 Prolong Diamond Antifade를 사용하여 장착하고 공초점 현미경을 사용하여 이미지화했습니다. 척추강내 투여된 원숭이의 척수 및 뇌 절편을 GFP 염색하기 위해 우리는 절편을 닭 항-GFP(1:500; Aves Labs, cat # GFP-1020; RRID: AB_10000240)와 함께 배양하고 PBS로 1~200회 세척을 수행했습니다. , 당나귀 항닭 IgY(703:605; Jackson ImmunoResearch Lab, cat # 155-2340379-0.1; RRID: AB_100)와 함께 배양하고 장착하기 전에 8787~10회 세척했습니다. 우리는 모든 항체를 희석하고 017% Triton X-000(Sigma-Aldrich, T121) 및 XNUMX% 일반 당나귀 혈청(Jackson ImmunoResearch, XNUMX-XNUMX-XNUMX)이 보충된 PBS를 사용하여 밤새 실온에서 진탕하면서 모든 인큐베이션을 수행했습니다.
형태학적 재구성을 위해 뇌를 300μm 섹션으로 분할하고 굴절률 매칭 솔루션에서 배양했습니다.72 굴절률 매칭 용액에 담근 슬라이드에 장착하기 전에 72 시간 동안. 침지액으로 25% 글리세롤을 사용하는 공초점 현미경과 ×25 대물렌즈(LD LCI Plan-Apochromat ×0.8/100 Imm Corr DIC)를 사용하여 영상을 촬영했습니다. 우리는 타일을 캡처했습니다 Z 관심 있는 셀 주위에 스택(제안된 캡처 설정을 사용하여 각 프레임에 대해 1,024 × 1,024)을 쌓고 추적을 위해 적절한 시야를 자릅니다. 추적은 반자동 및 자동화 방법을 사용하여 Imaris(Oxford Instruments, RRID: SCR_007370)에서 수행되었습니다.
뉴런(NeuN) 및 성상교세포(S100β) 정량화를 위해 슬라이스를 항-NeuN(EPR12763) 항체(1:200; Abcam, cat # ab177487; RRID: AB_2532109) 또는 항-S100β 항체(1:200; Abcam)를 사용하여 염색했습니다. , 카탈로그 번호 ab52642; RRID: AB_882426) 0.1% Triton X-100 및 10% 일반 당나귀 혈청이 보충된 PBS에서 하룻밤 동안. 슬라이스를 PBS로 647~1회 세척하고 PBS + 200%의 Alexa Fluor 711(605:152; Jackson ImmunoResearch Labs, cat # 2492288-0.1-100; RRID: AB_10)과 접합된 항토끼 IgG 항체에서 밤새 배양했습니다. 트리톤 X-XNUMX + XNUMX% 노멀 당나귀 세럼. Prolong Diamond Antifade를 사용하여 XNUMX~XNUMX회 세척하고 장착한 후, Z 공초점 현미경과 x25 대물렌즈를 사용하여 스택을 쌓습니다. Python(RRID: SCR_008394) 및 Cellpose(https://www.cellpose.org/; RRID: SCR_021716)73.
생체 외 XNUMX광자 이미징
이미징에 적합한 크기의 뇌 조각은 vibratome을 사용하여 더 큰 조각에서 400μm의 두께로 준비되었으며 이전에 게시된 프로토콜에서 설명한 대로 XNUMX광자 이미징 전에 카보겐 가스로 버블링된 인공 뇌척수액에 저장되었습니다.74,75. GCaMP8s 반응을 테스트하기 위해 표시된 수의 펄스를 사용한 전기 자극(4–5 V, 80 Hz, 0.3 s 지속 시간)이 이미지화된 뉴런에서 100~200 μm 떨어진 곳에 배치된 세포외 단극 전극을 사용하여 전달되었습니다. 이미징의 프레임 속도는 30Hz였습니다. 분할된 관심 영역의 흔적을 Δ로 표시했습니다.F/F0 = (F(t) - F0)/F0어디로 F0 전기 자극 전의 모든 형광 값의 평균으로 정의됩니다. 상승 시간은 신호의 상승 위상이 피크의 10%에서 피크의 90%에 도달하는 데 필요한 시간으로 정의됩니다. 감쇠 시간 상수는 지수의 합을 신호의 감쇠 단계에 맞춰서 얻어졌습니다. 신호 대 잡음비는 신호의 피크 진폭을 전기 자극 전 형광 흔적의 표준 편차로 나누어 얻은 것입니다.
성인 붉은 털 원숭이 조각의 특성
워싱턴 국립 영장류 연구 센터의 성체 붉은털원숭이(14세 1개월, 체중 10.83 kg) 한 마리가 일상적인 안락사를 위해 계획되었으며, 시설의 조직 배포 프로그램의 일환으로 뇌가 수집되었습니다. 상측두회 블록을 300μm 슬라이스로 절단하고 해당 슬라이스를 회수했습니다.74 이전에 설명한대로 공기-액체 막 경계면에서 배양되었습니다.76. 슬라이스를 플레이팅한 후 약 30분 후에 AAV(1 × 2) 5–10 μl를 투여했습니다.13 vg ml-1 AAV9 또는 AAV.CAP-Mac 패키징(ssCAG-FXN-HA 또는 ssCAG-eGFP). 실험은 각 조건에 대해 생물학적으로 48중으로 수행되었으며, 형질도입 후 8일에 조직 수집까지 배양 배지를 20시간마다 새로 고쳤습니다. 조직 수집 당일, 형질도입을 확인하기 위해 슬라이스를 이미지화하고, 슬라이스를 반으로 자르고, 각 절반 슬라이스를 드라이아이스-에탄올 욕조에서 급속 냉동했습니다. 샘플은 추가 처리가 완료될 때까지 -XNUMX °C에서 보관되었습니다.
각 절반 조각이 처리되었습니다(DNA 및 RNA 복구를 위해 각각 하나씩). Qiagen DNeasy 혈액 및 조직 키트(Qiagen, 카탈로그 번호 69504)를 사용하여 DNA를 분리하고 TRIzol(Thermo Fisher Scientific, 카탈로그 번호 15596026) 및 PureLink RNA Mini 키트(Thermo Fisher Scientific, 카탈로그 번호 12183018A)를 사용하여 RNA를 회수했습니다. PureLink RNA Mini 키트의 첫 번째 세척을 다음과 같이 수정하여 RNA 샘플에서 DNA를 제거했습니다. 350μl의 세척 완충액 1로 세척한 다음 RDD 완충액(Qiagen 카탈로그 # 80)에 79254μl의 RNase-free DNaseI를 추가하고 배양합니다. 컬럼을 실온에서 15분 동안; 그런 다음 프로토콜을 진행하기 전에 350μl의 세척 완충액 1로 다시 세척하십시오. Promega GoScript Reverse Transcription 키트(Promega, 카탈로그 번호 A400)를 사용하여 20μl 반응에서 5000ng 총 RNA로부터 첫 번째 가닥 cDNA 합성을 수행했습니다.
그런 다음 Roche Lightcycler II에서 정량적 PCR을 사용하여 각 샘플에서 발견된 벡터 게놈과 바이러스 전사체를 평가했습니다. 여기서 100ng의 DNA는 Thermo Fisher Scientific의 TaqMan 프로브(EGFP-FAM 프로브, 분석 ID Mr20_mr, 카탈로그 #04097229, 맞춤형 게놈 참조 프로브 CN4331182-2386-VIC, 분석 ID ARH2DUK, 카탈로그 번호)를 사용하여 6μl 증폭 반응에 사용되었습니다. 4448512, 두 가지 모두를 대상으로 설계됨 M. 물라타 및 마 카카 네 메스 티나).
녹색 원숭이 실험
모든 녹색 원숭이 (C. 사바에우스) 절차는 Virscio에서 수행되었으며 IACUC의 승인을 받았습니다. 모든 원숭이에 대해 중화 항체를 검사한 결과 <1:5 역가를 갖는 것으로 확인되었습니다. 대략 7~8개월령(1.0~1.3 kg)에 원숭이에게 정맥 투여를 실시했습니다(보충 표). 6). 투여 전 최소 10분 동안, 최대 60분 동안 투여량 제제를 대략 실온으로 평형화되도록 했습니다. IV 용량 부피는 0일째 체중을 기준으로 했습니다. 동물을 케타민(8.0 mg kg)으로 진정시켰습니다.-1) 및 자일라진(1.6 mg kg)-1). 주사 부위를 면도하고 클로로헥시딘과 70% 이소프로판올로 준비한 후 IV 카테터를 삽입하기 전에 외과적으로 문지릅니다. 투여는 AAV(7.5 × 10)의 단일 IV 주입으로 발생했습니다.13 또는 7.6×1013 vgkg-1) 0일째에 휴대용 주입 장치를 사용하여 1 ml min의 목표 속도로 복재 정맥을 통해 투여-1. 일반적인 건강 상태는 투여 1주 전부터 케이지 측면 관찰을 통해 하루 8회 확인되었습니다. 예정된 안락사 시간에 원숭이들은 케타민(10–XNUMX mg kg)으로 진정되었습니다.-1 근육 내) 및 나트륨 펜토바르비탈(100 mg kg)로 안락사-1 IV 효과). 각막 반사가 소실된 경우, 연동 펌프 세트를 사용하여 냉각된 PBS로 약 100ml min의 속도로 경심장 관류(좌심실)를 수행했습니다.-1 조직 수집 전에 누출되는 체액이 깨끗해 질 때까지. 조직의 큐브는 좌뇌 반구 및 기타 다양한 기관에서 수집되었으며 생체분포를 위한 추가 처리를 위해 액체 질소의 증기상에서 냉동되었습니다. 우뇌 반구를 제거하고 ~4mm 관상 조각으로 자르고 실온에서 약 20시간 동안 약 10%의 24% 중성 완충 포르말린으로 그대로 후고정했습니다.
Thermo Fisher MagMax DNA Ultra 2.0 추출 키트(카탈로그 번호 A36570)를 사용하여 CNS 및 말초 조직에서 게놈 DNA를 추출했습니다. Qubit dsDNA 분석을 사용한 형광 정량화를 통해 DNA 수율을 평가했습니다. 약 20ng의 DNA를 각 20μl 반응에 로딩하고 플레이트를 BioRad CFX Connect Real-Time PCR 검출 시스템(카탈로그 번호 1855201)에서 실행했습니다. 바이러스 복제수 분석은 단일 증폭 산물의 검출을 통해 특이성을 검증했습니다. 반응당 XNUMX개 복사본보다 큰 검출 하한을 평가하여 민감도를 평가합니다. 표준곡선을 확보하여 선형성 확보 R2 >0.95였습니다. 반응물은 FastStart Universal SYBR Green Master(Rox)(카탈로그 카탈로그 번호 4913850001)에서 조립되었습니다. 프라이머의 서열은 ACGACTTCTTCAAGTCCGCC(순방향) 및 TCTTGTAGTTGCCGTCGTCC(역방향)였습니다. PCR 프로토콜은 95초 동안 180 °C의 초기 변성 단계를 사용한 다음, 40초 동안 95 °C, 15초 동안 60 °C의 60주기를 사용했으며, 각 60 °C 주기 후에 이미징 단계를 수행했습니다. 1 × 10에서 바이러스에 존재하는 GFP 주형 서열을 포함하는 선형화된 플라스미드로 표준 곡선을 생성했습니다.8 1×10까지10 매트릭스 효과를 제어하기 위해 본 연구의 샘플과 동일한 키트를 사용하여 준비된 미처리 미처리 원숭이 DNA 샘플에 희석된 사본. 바이러스 DNA의 사본은 가장 적합한 선에 대한 방정식을 사용하여 표준 곡선으로부터 계산되었습니다. 감염 다중도 값은 반응당 숙주 세포 DNA의 측정된 총 게놈 중량을 기준으로 계산되었습니다.
고정 후, 조직을 10 °C에서 각각 20 h 동안 30% > 24% > 4% 자당에 넣은 다음 O.C.T에 삽입했습니다. 화합물을 만들고 냉동절편할 때까지 -80 °C에서 보관했습니다. 조직 블록을 저온 유지 장치에서 -20°C까지 올려 30μm 슬라이스로 절단하고 저온 절단 후 슬라이드에 건조 장착했습니다. 절편화한 후 슬라이드를 실온에서 밤새 방치하여 건조시켰습니다. 뉴런 정량화를 돕기 위해 우리는 토끼 항-GFP(1:100; Millipore-Sigma, cat # AB3080; RRID: AB_91337) 및 마우스 항-NeuN(A60)(1:500; Millipore-Sigma, 고양이 번호 MAB377, RRID: AB_2298772). 488차 항체 염색에는 다음과 같은 1차 항체와 농도가 사용되었습니다: 당나귀 항토끼 Alexa Fluor 500(21206:2535792; Thermo Fisher Scientific, 카탈로그 # A-647; RRID: AB_1) 및 당나귀 항-마우스 Alexa Fluor 500(31571 :162542, Thermo Fisher Scientific, 카탈로그 번호 A-1, RRID: AB_0.25). 모든 항체를 100% Triton X-5.00(PBST) 및 2% 정상 당나귀 혈청이 보충된 1X PBS로 희석했습니다. 10,000차 항체 인큐베이션을 실온에서 밤새 방치했습니다. 그런 다음 섹션을 PBST로 세척했습니다. 1306차 항체 인큐베이션은 실온에서 5시간 동안 수행되었습니다. 절편을 PBST로 XNUMX회 세척했습니다. 절편을 DAPI 용액(XNUMX:XNUMX, Invitrogen, DXNUMX)에서 실온에서 XNUMX분 동안 배양한 다음 세척했습니다. 섹션은 Prolong Diamond Antifade를 사용하여 커버슬립되었습니다.
동물당 세 부분을 염색하고 이미지화했습니다. 각 섹션은 800중으로 이미지화되었으며 각 관심 영역에는 총 1개의 이미지가 있습니다. 관심 조직 영역은 다음 획득 매개변수를 사용하여 Keyence BZ-X500으로 이미지화되었습니다: GFP(5/1 s), Cy1(12 s), DAPI(XNUMX/XNUMX s), 고해상도 Z 1.2 μm 피치로 스택합니다. 다음 뇌 하위 영역이 영상화되었습니다: 전두엽, 정수리, 측두엽, 후두엽 피질, 소뇌, 꼬리뼈, 피각 및 시상(내측, 복부 측면 및 복부 후방 핵). 정량화를 위해 ImageJ(RRID: SCR_003070)를 사용하여 반자동 세포 계수 방법을 수행했습니다. 임계값과 입자 분석을 사용하여 NeuN 양성 및 DAPI 양성 세포를 정량화했습니다. ImageJ의 세포 계수기를 사용하여 GFP 양성 세포와 GFP 및 NeuN 이중 양성 세포를 수동으로 계산했습니다.
iPSC 실험
제조사의 프로토콜에 따라 Stemdiff Forebrain Differentiation and Maturation 키트(각각 StemCell # 08600 및 # 08605)를 사용하여 iPSC 유래 신경 전구 세포를 분화 및 성숙시켜 신경 배양을 생성했습니다. 신경 전구 세포는 Stemdiff SMADi 신경 유도 키트(StemCell l#1019)를 사용하여 포피 섬유아세포 유래 iPSC 계통: ACS-0100(ATCC# DYS-499; RRID: CVCL_X08581)의 분화, Stemdiff 신경 로제트 선택을 통한 선택에 의해 생산되었습니다. 제조업체의 프로토콜에 따라 Stemdiff Neural Progenitor Media(StemCell l#05832)에서 시약(StemCell l#05833) 및 확장. 뉴런은 형질도입을 위해 리플레이팅하기 전 최소 8일 동안 성숙되었습니다.
폴리오르니틴 및 라미닌 코팅된 검은색 벽 15,000웰 광학 플레이트에 웰당 96개의 세포를 접종한 성숙한 뉴런 배양물을 형질도입 전 추가 4일 동안 배양했습니다. 복제 웰은 90% 성숙 배지 및 10% OptiPRO SFM에서 4자릿수로 연속 희석된 바이러스로 형질도입되었습니다. 형질도입 1일 후, 배양물을 XNUMX% PFA로 고정하고 XNUMX μg ml로 대조염색했습니다.-1 Hoechst 33322. 형질도입된 세포의 식별은 CellInsight CX60 HCS 플랫폼에서 386채널 형광 검출(Hoechst, ex440/em485; eGFP, ex521/em5)을 사용하여 웰당 XNUMX 필드를 이미징하여 결정되었습니다. 개별 세포는 핵의 Hoechst 검출과 각 세포에 크기 및 접촉 제한 링 마스크를 적용하여 식별되었습니다. 개별 링 마스크 내에서 임계값 수준 이상의 eGFP 형광을 측정하여 세포 형질도입을 결정했습니다. 각 모집단에 대해 형질도입된 세포의 백분율을 적용된 용량에 대해 표시했습니다. 곡선 맞춤 및 EC50 값은 Prism GraphPad(RRID: SCR_002798) 작용제 대 반응(5,000개 매개변수) 회귀 방법을 사용하여 결정되었습니다. 셀당 eGFP 발현 효율성을 보고하기 위해 eGFP 스팟 형광 강도는 웰당 최소 XNUMX개의 셀에 걸쳐 각 링 마스크의 평균을 구했습니다. 농도 회귀 방법으로 Prism GraphPad Biphasic X를 사용하여 곡선 맞춤을 얻었습니다.
통계 및 재현성
대표 이미지의 경우 각 샘플에서 최소 2.38개의 개별 슬라이스를 이미징을 위해 장착했습니다. 단일 동물의 각 뇌 영역 내에서 최소 2.38개의 서로 다른 시야가 확보되었습니다(타일링 후 최소 시야, 50 mm × XNUMX mm, 슬라이스 두께, XNUMX μm). 이는 세 개의 뇌 조각에 걸쳐 XNUMX개의 별도 시야에 해당합니다. , 이미징 샘플 전반에 걸쳐 일관성을 보장합니다.
보고 요약
연구 설계에 대한 추가 정보는 Nature 포트폴리오 보고 요약 이 기사에 링크되어 있습니다.
- SEO 기반 콘텐츠 및 PR 배포. 오늘 증폭하십시오.
- PlatoData.Network 수직 생성 Ai. 자신에게 권한을 부여하십시오. 여기에서 액세스하십시오.
- PlatoAiStream. 웹3 인텔리전스. 지식 증폭. 여기에서 액세스하십시오.
- 플라톤ESG. 자동차 / EV, 탄소, 클린테크, 에너지, 환경, 태양광, 폐기물 관리. 여기에서 액세스하십시오.
- BlockOffsets. 환경 오프셋 소유권 현대화. 여기에서 액세스하십시오.
- 출처: https://www.nature.com/articles/s41565-023-01419-x
- :이다
- :어디
- $UP
- 000
- 1
- 10
- 100
- 11
- 12
- 13
- 14
- 15%
- 16
- 17
- 180
- 2%
- 20
- 200
- 2011
- 2015
- 2016
- 2017
- 2018
- 2019
- 2020
- 2021
- 24
- 25
- 27
- 30
- 300
- 31
- 33
- 39
- 40
- 50
- 500
- 60
- 66
- 7
- 70
- 72
- 75
- 8
- 80
- 9
- 95%
- a
- 위의
- 절대
- ACCESS
- 일치
- 에 따르면
- 달성
- 취득
- 가로질러
- 활동적인
- 활동
- Ad
- 더하다
- 추가
- 추가
- 또한
- 관리 된
- 관리
- 채택
- 성인
- 많은
- 불리한
- 후
- 다시
- 나이
- AL
- 알렉사
- 연산
- 정렬 된
- All
- 수
- 따라
- 또한
- 양
- 확대
- 증폭
- 증폭
- an
- 분석
- 닻
- 및
- 동물
- 동물
- 항체
- 어떤
- 적용된
- 적용
- 적당한
- 인가 된
- 대략
- 지역
- 약
- 기사
- 인조의
- AS
- 조립
- 조립
- 평가
- 평가
- 지원
- At
- 자동화
- 가능
- 평균
- 떨어져
- 등뼈
- 밴드
- 기반으로
- BD
- BE
- 되었다
- 전에
- 처음
- 존재
- 기준
- BEST
- 사이에
- 바이어스
- 제본
- 검정
- 블록
- 블록
- 피
- 몸
- 타고난
- 두
- BP
- 뇌
- 두뇌
- 간단히
- 가져
- 버퍼
- 비자 면제 프로그램에 해당하는 국가의 시민권을 가지고 있지만
- by
- 케이지
- 계산하다
- 계산 된
- 캘리포니아
- CAN
- 포착
- 캡처
- 한
- 뱃짐
- CAT
- 세포
- 셀
- 세포의
- 센터
- 중심적인
- 체인
- 찬
- 이전 단계로 돌아가기
- 변경
- 선택
- 선명한
- 클릭
- 면밀히
- 클러스터링
- 수집
- 단
- 결합 된
- 결합
- 위원회
- 공통의
- 보완적인
- 완전한
- 진행완료
- compliance
- 준수
- 화합물
- 집중
- 조건
- 조건
- 확인하기
- 확인 됨
- 연결하기
- 연결
- 상수
- 구축
- 포함
- 이 포함되어 있습니다
- 제어
- 제어 장치
- 사본들
- 핵심
- 화관
- 계수기
- 기준
- 문화
- 곡선
- 관습
- 맞춤형
- 절단
- 주기
- 주기
- 매일
- 데이터
- 데이터 세트
- 데이비스
- 일
- 일
- 깊은
- 한정된
- 전달
- 배달
- 기술 된
- 디자인
- 설계
- 원하는
- 세부설명
- Detection System
- 결정
- 결정된
- 일탈
- 장치
- 디바이스
- 다이아몬드
- 샌디에고
- 차이
- 다른
- 희석
- 디스플레이
- 표시된
- 거리
- 뚜렷한
- 분포
- 다양
- 다양성
- DNA
- 한
- 선량
- 투약
- 드라이브
- 건조
- 지속
- ...동안
- e
- E & T
- 마다
- 이전
- 판
- 효과
- 효과
- 효율성
- 효율적인
- 효율적으로
- 중
- 요소
- 제거
- 임베디드
- 영국
- 확인
- 보장
- 확립 된
- 에테르 (ETH)
- EU
- 평가
- 모든
- 예
- 교환
- 확장
- 실험
- 실험
- 표현
- 추출물
- 추출
- 가족
- 연방 준비 은행
- 여자
- 들
- Fields
- 무화과
- 그림
- 파일
- 필터링
- 최후의
- 최종적으로
- 결과
- 먼저,
- 맞게
- 입어 보기
- 다섯
- 고정
- 플래시
- 흐름
- 유체
- 다음에
- 수행원
- 다음
- 식품
- 럭셔리
- 수율을 위해
- 앞으로
- 발견
- 사
- FRAME
- 무료
- 에
- 겨울 왕국
- 기능의
- 추가
- 가스
- 계량기
- 일반
- 생성
- 생성
- 세대
- 유전학
- 게놈
- 유전체학
- 주어진
- 그래프
- 큰
- 초록색
- 그룹
- 여러 떼
- 가이드 라인
- 반
- 처리
- 있다
- 데
- 머리
- 건강
- 여기에서 지금 확인해 보세요.
- 높은
- 높은 해상도
- 구멍
- 균질화 된
- 주인
- HTTPS
- 사람의
- ICE
- ID
- 같은
- 식별
- 확인
- if
- ii
- III
- 형상
- 영상
- 잠긴
- 담금
- 개선
- in
- 포함
- 배양
- 독립하여
- 색인
- 표시된
- 색인
- 개인
- 개별적으로
- 유도
- 감염
- 정보
- 주입 된
- 주입
- 처음에는
- 처음에는
- 를 받아야 하는 미국 여행자
- 학회
- 기관
- 악기
- 관심
- 인터페이스
- 으로
- 정맥
- 소개
- 참여
- 외딴
- 잭슨
- 키트
- 실험실
- 실험실
- 실험실
- 큰
- ld
- 가장 작은
- 왼쪽 (left)
- 적게
- 레벨
- 도서관
- 도서관
- 생활
- 제한
- 라인
- LINK
- 연결
- 리퀴드
- 명부
- 간
- 지방의
- 장기
- 오프
- 절감
- 낮춘
- 만든
- 유지
- 수동으로
- 매핑
- 마스크
- 마스크
- 석사
- 경기
- 어울리는
- 자료
- 매트릭스
- 측정
- 측정
- 측정
- 미디어
- 매질
- 정신
- 정신 건강
- 방법
- 방법
- 메트릭
- 생쥐
- 현미경
- 분
- 최저한의
- 분
- 혼합
- 혼합물
- ML
- 수정
- 모니터링
- 달
- 개월
- 배우기
- mRNA
- MSCI
- 즉
- 나노 기술
- 국가의
- 자연
- 가까운
- 필요한
- 네트워크
- 네트워크
- 신경
- 신경
- 뉴런
- 신제품
- 다음 것
- NIH
- 아니
- 노드
- 표준
- 번호
- 목표
- 획득
- 발생
- of
- 낡은
- on
- ONE
- 만
- 최적의
- 최적화
- or
- 명령
- 기타
- 우리의
- 위에
- 밤새
- 옥스퍼드
- 산소
- 포장
- 쌍을 이루는
- 매개 변수
- 부품
- 입자
- PBS
- PCR
- 피크 (캐노피 지붕쪽)
- 펜
- 펜실베니아
- 용
- tỷ lệ phần trăm
- 완전한
- 수행
- 주위의
- 상
- 관로
- 피치
- 장소
- 계획
- 혈장
- 플랫폼
- 플라톤
- 플라톤 데이터 인텔리전스
- 플라토데이터
- ...을 더한
- 풀
- 인구
- 유가 증권
- Precision
- 준비
- 준비
- 제시
- 제시
- 예방
- 이전에
- 일차
- 뇌관
- 탐침
- 순서
- 절차
- 방법
- 처리
- 처리
- 생산
- 생산
- 프로덕트
- 생산
- 제품
- 선조
- 프로그램
- 보호하는
- 단백질
- 단백질
- 프로토콜
- 프로토콜
- 제공
- 출판
- 펌프
- 구매 한
- Python
- 품질
- 정량적 인
- 큐빗
- 토끼
- 높인
- 율
- 비율
- 도달
- 반응
- 반응
- 읽기
- 실시간
- 접수
- 추천
- 회복
- 지방
- 지역
- 되돌아옴
- 규정하는
- 상대적인
- 제거
- 제거됨
- 복제
- 신고
- 보고
- 통계 보고서
- 대리인
- 대표되는
- 필수
- 연구
- 각기
- 응답
- 응답
- 제한
- 결과
- 역
- 연락해주세요
- 반지
- 상승
- 상승
- RNA
- 강력한
- 록
- 방
- 길
- 달리기
- 실행
- s
- 같은
- 산
- 샌디에고
- 모래의
- 규모
- 예약
- SCI
- 과학적인
- 점수
- 점수
- 전형
- 스크립트
- 보조
- 섹션
- 섹션
- 참조
- 분할
- 선택된
- 선택
- 선택적
- 감도
- 전송
- 별도의
- 순서
- 시퀀싱
- 세럼
- 세트
- 설정
- 몇몇의
- 제공
- 표시
- 뒷면
- 시각
- 신호
- 비슷한
- 단일
- 앉다
- SIX
- 크기
- 피부
- 일부분
- 슬라이드
- 슬라이드
- 천천히
- 작은
- 나트륨
- 해결책
- 소리
- 구체적으로
- 특성
- Spot
- 스택
- 스택
- 표준
- 단계
- 단계
- 저장
- 저장
- 끈
- 교육과정
- 후속의
- 적당한
- 우수한
- 표면
- 인조
- 체계
- 전신의
- 시스템은
- 테이블
- 촬영
- 도청
- 목표
- 대상
- 대상
- 기술
- Technology
- 이 템플릿
- 안색
- 단말기
- 지원
- 보다
- 그
- XNUMXD덴탈의
- 지역
- 그들의
- 그들
- 그때
- 그곳에.
- 따라서
- Bowman의
- 그들
- 이
- 세
- 임계값
- 을 통하여
- 도처에
- 시간
- 시대
- 조직
- 에
- 했다
- 금액
- 더듬다
- 트레이싱
- 이전
- 전송
- 처리 된
- 소라 고둥
- 두번
- 트위스트
- 두
- 유형
- 울트라
- 아래에
- 유일한
- 보편적 인
- 대학
- University of California
- 펜실베니아 대학
- 까지
- 사용
- 익숙한
- 사용
- 활용
- 활용
- 검증 된
- 마케팅은:
- 변형
- 여러
- 버전
- 대
- 를 통해
- 관측
- 바이러스의
- 바이러스
- 바이러스
- 생체
- 볼륨
- 였다
- 워싱턴
- 물
- we
- 주
- 주
- 무게
- 잘
- 웰스
- 했다
- 어느
- 모든
- 펼친
- 과
- 이내
- 세계
- X
- 년
- 수율
- 산출량
- 선
- 제퍼 넷