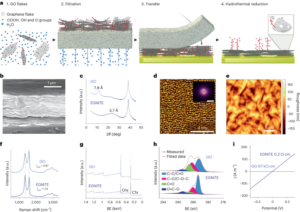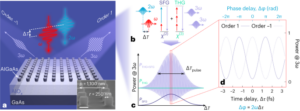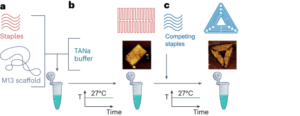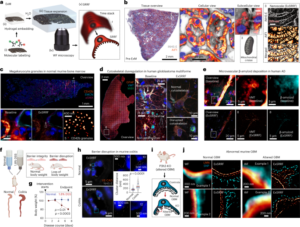
Levine, AJ & Oren, M. I primi 30 anni di p53: sempre più complessi. Nat. Rev. cancro 9, 749-758 (2009).
Shaw, P. H. Il ruolo di p53 nella regolazione del ciclo cellulare. Pathol. Ris. Pratica. 192, 669-675 (1996).
Vousden, K. H. & Lane, D. P. p53 in salute e malattia. Naz. Rev. Mol. Biologia cellulare. 8, 275-283 (2007).
Vousden, K. H. & Ryan, K. M. p53 e metabolismo. Nat. Rev. cancro 9, 691-700 (2009).
La rete di ricerca Cancer Genome Atlas Analisi genomiche integrate del carcinoma ovarico. Natura 474, 609-615 (2011).
Olivier, M., Hollstein, M. & Hainaut, P. Mutazioni TP53 nei tumori umani: origini, conseguenze e uso clinico. Cold Spring Harb. Perspect. Biol. 2, a001008 (2010).
Bertheau, P. et al. p53 nei sottotipi di cancro al seno e nuove informazioni sulla risposta alla chemioterapia. Seno 22, S27 – S29 (2013).
Freed-Pastor, W. A. & Prives, C. Mutant p53: un nome, molte proteine. Genes Dev. 26, 1268-1286 (2012).
Muller, P. A. & Vousden, K. H. mutazioni p53 nel cancro. Naz. Biologia cellulare. 15, 2-8 (2013).
Yue, X. et al. P53 mutante nel cancro: accumulo, guadagno di funzione e terapia. J.Mol. Biol. 429, 1595-1606 (2017).
Lukashchuk, N. & Vousden, K. H. Ubiquitinazione e degradazione del mutante p53. Mol. Biolo cellulare. 27, 8284-8295 (2007).
Schulz-Heddergott, R. et al. L'ablazione terapeutica del mutante p53 con guadagno di funzione nel cancro del colon-retto inibisce la crescita e l'invasione del tumore mediata da Stat3. Cancer Cell 34, 298-314 (2018).
Zhang, C. et al. Mutante p53 con guadagno di funzione nella progressione e nella terapia del cancro. J.Mol. Biolo cellulare. 12, 674-687 (2020).
Bykov, V. J. N., Eriksson, S. E., Bianchi, J. & Wiman, K. G. Targeting del mutante p53 per una terapia efficace contro il cancro. Nat. Rev. cancro 18, 89-102 (2018).
Parrales, A. & Iwakuma, T. Targeting del mutante oncogenico p53 per la terapia del cancro. Davanti. Oncol. 5, 288 (2015).
Zhang, YJ et al. Degradazione proteasomica dipendente dalla glutationilazione delle proteine p53 mutanti ad ampio spettro mediante struttura zeolitica imidazolata ingegnerizzata-8. biomateriali 271, 120720 (2021).
Qian, J. et al. Miglioramento della chemioterapia del cancro con mutazione p53 attraverso la degradazione proteasomale ubiquitinazione-dipendente delle proteine p53 mutanti mediante nanoparticelle ZnFe-4 ingegnerizzate. Adv. Funz. Mater. 30, 2001994 (2020).
Kocaturk, N. M. & Gozuacik, D. Diafonia tra l'autofagia dei mammiferi e il sistema ubiquitina-proteasoma. Davanti. Cell Dev. Biol. 6, 128 (2018).
Jing, M. et al. Nanocarrier fotoresponsivo assemblato con PAMAM caricato con inibitore dell'autofagia per la terapia sinergica del cancro. Piccolo 17, e2102295 (2021).
Lee, CW et al. L'autofagia selettiva degrada i complessi dei pori nucleari. Naz. Biologia cellulare. 22, 159-166 (2020).
Zhang, Y. et al. Sfruttare l’autofagia pro-sopravvivenza indotta da nanoparticelle di tetrapodi in lega di rame-palladio per la terapia fototermica ottimizzata del cancro resistente ai farmaci. Nat. Commun. 9, 4236 (2018).
Khaminets, A., Behl, C. & Dikic, I. Segnali dipendenti e indipendenti dall'ubiquitina nell'autofagia selettiva. Tendenze Cellule Biol. 26, 6-16 (2016).
Kirkin, V. & Rogov, V. V. Una diversità di recettori autofagici selettivi determina la specificità del percorso autofagico. Mol. Cellula 76, 268-285 (2019).
Shaid, S., Brandts, C. H., Serve, H. & Dikic, I. Ubiquitinazione e autofagia selettiva. La morte cellulare differisce. 20, 21-30 (2013).
Sarraf, SA et al. La perdita dell'autofagia diretta da TAX1BP1 provoca un accumulo di aggregati proteici nel cervello. Mol. Cellula 80, 779-795 (2020).
Jo, C. et al. Nrf2 riduce i livelli di proteina tau fosforilata inducendo la proteina adattatrice dell'autofagia NDP52. Nat. Commun. 5, 3496 (2014).
Pankiv, S. et al. p62/SQSTM1 si lega direttamente ad Atg8/LC3 per facilitare la degradazione degli aggregati proteici ubiquitinati mediante autofagia. J. Biol. Chem. 282, 24131-24145 (2007).
Guida, E. et al. Gli aptameri peptidici che prendono di mira la p53 mutante inducono l'apoptosi nelle cellule tumorali. Cancer Res. 68, 6550-6558 (2008).
Man, N., Chen, Y., Zheng, F., Zhou, W. & Wen, LP Induzione di vera autofagia da parte di lipidi cationici nelle cellule di mammifero. L'autofagia 6, 449-454 (2010).
Roberts, R. et al. L'autofagia e la formazione di autofagosomi tubulovescicolari forniscono una barriera contro il rilascio di geni non virali. L'autofagia 9, 667-682 (2013).
Li, M. et al. Mono- versus poliubiquitinazione: controllo differenziale del destino di p53 da parte di Mdm2. Scienze 302, 1972-1975 (2003).
Li, M., Luo, J., Brooks, C. L. & Gu, W. L'acetilazione di p53 inibisce la sua ubiquitinazione da parte di Mdm2. J. Biol. Chem. 277, 50607-50611 (2002).
Maeda, H., Nakamura, H. & Fang, J. L'effetto EPR per la somministrazione di farmaci macromolecolari a tumori solidi: miglioramento dell'assorbimento del tumore, riduzione della tossicità sistemica e imaging tumorale distinto in vivo. Adv. Drug Deliv. rev. 65, 71-79 (2013).
Peer, D. et al. Nanovettori come piattaforma emergente per la terapia del cancro. Naz. Nanotecnologie. 2, 751-760 (2007).
Alexandrova, EM et al. Migliorare la sopravvivenza sfruttando la dipendenza del tumore dal mutante stabilizzato p53 per il trattamento. Natura 523, 352-356 (2015).
Ghosh, M. et al. Il mutante p53 sopprime la segnalazione immunitaria innata per promuovere la tumorigenesi. Cancer Cell 39, 494-508 (2021).
Baslan, T. et al. Evoluzione ordinata e deterministica del genoma del cancro dopo la perdita di p53. Natura 608, 795-802 (2022).
Parrales, A. et al. DNAJA1 controlla il destino del mutante p53 mal ripiegato attraverso la via del mevalonato. Naz. Biologia cellulare. 18, 1233-1243 (2016).
Proia, D. A. & Bates, R. C. Ganetespib e HSP90: tradurre ipotesi precliniche in promesse cliniche. Cancer Res. 74, 1294-1300 (2014).
Padmanabhan, A. et al. La via lisosomiale dipendente dall'USP15 controlla il turnover di p53-R175H nelle cellule di cancro ovarico. Nat. Commun. 9, 1270 (2018).
Garufi, A. et al. Degradazione della proteina mutante p53H175 da parte di Zn(II) attraverso l'autofagia. Cell Death Dis. 5, e1271 (2014).
Li, Z. et al. Abbassamento allele-selettivo della proteina HTT mutante mediante composti linker HTT-LC3. Natura 575, 203-209 (2019).
Inverno, GE et al. Coniugazione con ftalimide come strategia per la degradazione delle proteine bersaglio in vivo. Scienze 348, 1376-1381 (2015).
Li, HJ et al. Nanoparticelle clusterizzate che rispondono agli stimoli per una migliore penetrazione nel tumore e un’efficacia terapeutica. Proc. Natl Acad. Sci. Stati Uniti d'America 113, 4164-4169 (2016).
- Distribuzione di contenuti basati su SEO e PR. Ricevi amplificazione oggi.
- PlatoData.Network Generativo verticale Ai. Potenzia te stesso. Accedi qui.
- PlatoAiStream. Intelligenza Web3. Conoscenza amplificata. Accedi qui.
- PlatoneESG. Carbonio, Tecnologia pulita, Energia, Ambiente, Solare, Gestione dei rifiuti. Accedi qui.
- Platone Salute. Intelligence sulle biotecnologie e sulle sperimentazioni cliniche. Accedi qui.
- Fonte: https://www.nature.com/articles/s41565-023-01562-5
- ][P
- 003
- 01
- 07
- 08
- 09
- 1
- 10
- 11
- 12
- 13
- 14
- 15%
- 16
- 17
- 19
- 1996
- 20
- 2008
- 2010
- 2011
- 2012
- 2013
- 2014
- 2015
- 2016
- 2017
- 2018
- 2019
- 2020
- 2021
- 2022
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 35%
- 36
- 39
- 40
- 41
- 43
- 7
- 72
- 75
- 8
- 9
- a
- accumulazione
- Dopo shavasana, sedersi in silenzio; saluti;
- contro
- aggregato
- aggregati
- AL
- Lega
- an
- analisi
- ed
- articolo
- AS
- atlante
- barriera
- fra
- biomateriali
- Cervello
- Cancro al seno
- by
- Cancro
- Cellule cancerogene
- tumori
- cella
- Celle
- chemioterapia
- chen
- clicca
- Info su
- complesso
- Conseguenze
- di controllo
- controlli
- ciclo
- Morte
- consegna
- dipendenza
- determina
- Dev
- differire
- direttamente
- Malattia
- distinto
- Diversità
- droga
- Consegna farmaci
- e
- E&T
- effetto
- efficacia
- efficiente
- emergenti del mondo
- progettato
- migliorando
- Etere (ETH)
- EVER
- evoluzione
- facilitare
- destino
- Nome
- Nel
- formazione
- genoma
- genuino
- Crescita
- Crescita
- Sfruttamento
- Salute e benessere
- http
- HTTPS
- umano
- i
- ii
- Imaging
- immune
- migliorata
- miglioramento
- miglioramento
- in
- studente indipendente
- induzione
- innato
- intuizioni
- integrato
- ai miglioramenti
- invasione
- SUO
- Corsia
- livelli
- LINK
- spento
- abbassamento
- molti
- MOL
- Scopri di più
- Nome
- nanotecnologia
- Natura
- Rete
- New
- nucleare
- of
- on
- ONE
- ottimizzati
- origini
- Tumore ovarico
- sentiero
- penetrazione
- piattaforma
- Platone
- Platone Data Intelligence
- PlatoneDati
- preclinico
- progressione
- PROMETTIAMO
- promuoverlo
- Proteine
- Proteine
- fornire
- R
- riduce
- riferimento
- Regolamento
- riparazioni
- risposta
- Risultati
- Ruolo
- Ryan
- s
- Studioso
- SCI
- selettivo
- servire
- Segnali
- solido
- specificità
- primavera
- Strategia
- sopravvivenza
- sinergico
- sistema
- sistemico
- T
- Target
- mira
- Il
- Terapeutico
- terapia
- Attraverso
- a
- trattamento
- tumore
- tumori
- turnover
- comprensione
- uso
- contro
- vivo
- W
- con
- X
- anni
- zefiro