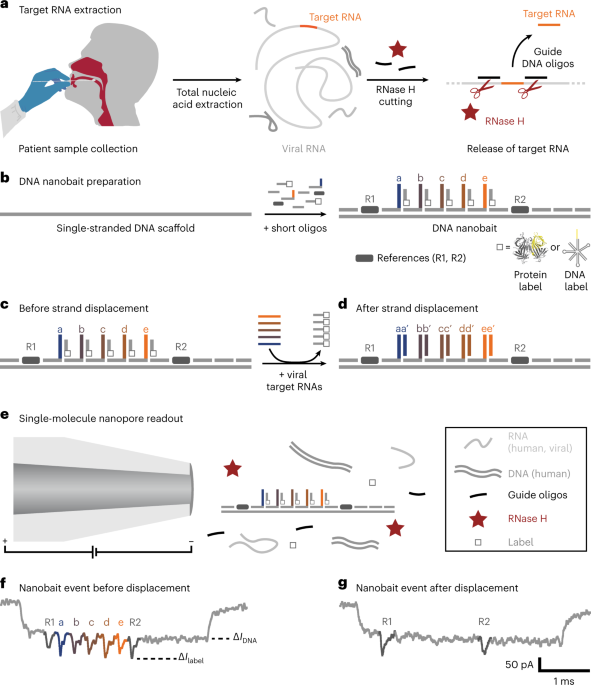
Betegmintagyűjtés
A betegek torkának (oropharyngealis tampon) letörlésével vett mintákat vettünk a korábban leírtak szerint.26. A mintákat COVID-19-szerű klinikai képpel rendelkező betegektől gyűjtöttük, és nukleinsav-extrakció után qRT-PCR-rel teszteltük. Röviden, az összegyűjtés után a tamponokat egy lízispuffert (4 M guanidin-tiocianátot, 25 mM Tris-HCl-t, 0.5% β-merkaptoetanolt és MS2 RNS-t (200 ng µl) tartalmazó MSXNUMX RNS-t tartalmazó mintacsőbe helyeztük.-1; Roche)). A csövet finoman megráztuk, hogy biztosítsuk a lízispuffer egyenletes eloszlását. A biztonsági lépéseket korábban leírták, és egy tanúsított CL2 laboratóriumban végezték el26.
Nukleinsav extrakció
A teljes nukleinsavat spin-oszlop alapú rendszerekkel és standardizált qRT-PCR teszttel extraháltuk.26. A belső erősítésvezérlő (MS2 (~6 × 104 PFU ml-110 ml lízispufferenként) hozzáadtuk a feltöltött lízispufferhez (25 µl/10 ml lízispuffer). A mintát 100 µl nukleázmentes vízben eluáltuk (nfH2O; Invitrogen) és 1 percig állni hagyjuk, majd 1 percig 21,130 XNUMX×-on centrifugáljuk.g (15,000 80 ford./perc) asztali mikrofugában. Az eluált mintákat közvetlenül qRT-PCR-nek vetettük alá. A fennmaradó nukleinsav-kivonatokat –XNUMX °C-on tároltuk, és a továbbiakban nanobait–nanopórus érzékelésre használtuk.
qRT-PCR a SARS-CoV-2-hez
A SARS-CoV-2 kimutatását a korábban leírtak szerint végeztük26. Reakciónként a mesterkeverék 12.5 µl 2× Luna Universal Probe One-Step reakciókeveréket, 0.5 µl 20 µM Wu forward primert (5′-ATGGGTTGGGATTATCCTAAATGTGA-3′), 0.5 µl 20 uM reverse primert tartalmazott. -GCAGTTGTGGCATCTCCTGATGAG-5'), 3 µl 0.3 µM MGB Probe 10 fluoreszcein (3'-ATGCTTAGAATTATGGCCTCAC-5'), 3 µl 0.5 µM belső kontroll 10M belső kontroll RNS-ből 2, 0.5 MS10 forward primer, 2. primer for MS0.3 RNS, 10 µl 2 µM belső próba (MS1 ROX), 3.9 µl Luna WarmStart RT Enzyme Mix és XNUMX µl nfH2O. Ezután a mesterkeverékből 20 µl-t aliquot részekre osztunk egy 96 lyukú lemez minden egyes üregébe, majd egyesítjük az egyes kivonatokból 5 µl-rel. A teljes extrakciós protokollon átesett MS2 belső extrakciós és amplifikációs kontroll negatív extrakciós kontrollként szerepelt a qRT-PCR lemez legalább két üregében. A lehetséges szennyeződés meghatározásához a qRT-PCR folyamatban 5 µl nfH2Az O-t qRT-PCR negatív kontrollként alkalmaztuk. Ezután 5 µl tüskés SARS-CoV-2 templát plazmidot helyeztünk egyetlen lyukba, mint a qRT-PCR pozitív kontrollt. Miután minden mintából 5 µl-t adtunk a kijelölt lyukhoz, a lemezt optikailag átlátszó műanyag tömítéssel lezártuk. A lemezt 1 percig 2,000-szeres sebességgel centrifugáltukg (1,000 ford./perc) 4 °C-on, majd behelyezzük a qRT-PCR gépbe (QuantStudio, Thermo Fisher Scientific), és a futtatást paraméterezzük. A fluoreszcein (FAM) és a karboxirodamin (ROX) jeleit megszereztük. A belső MS2 kontroll kimutatására ROX-ot, a SARS-CoV-2 RNS kimutatására fluoreszceint használtunk. A vizsgálatot 2 percig 25 °C-on, 15 percig 50 °C-on (a reverz transzkriptázhoz), 2 percig 90 °C-on végeztük, 45 ciklus előtt 95 °C-on 3 másodpercig, majd 60 °C-on 30 másodpercig. . Az eredményeket a helyes pozitív kontrollok megerősítése (a plazmid amplifikációja), az összes minta extrakciója és amplifikációs kontrollja (ROX csatorna), a negatív kontrollok nem amplifikációja és a kontrollok konzisztens átlagértékei határozták meg. A SARS-CoV-2 pozitivitást a fluoreszcein csatornában végzett amplifikáció igazolta megfelelő szigmoid görbével ≤36 CT érték mellett. Az MS2 és az MGB 3. szonda CT-értékeit megőriztük a vizsgálat minőségének és reprodukálhatóságának nyomon követése érdekében44.
Programozható RNase H vágás nanocsalihoz
A nanopórusérzékeléshez SARS-CoV-2 RNS kontrollokat, nukleinsav-kivonatokat (betegminták) vagy MS2 vírus RNS-t használtunk a nanocsalival történő kimutatáshoz. Először vezető oligot kevertünk a mintához, és 70 percig 5 °C-ra melegítettük. RNáz H-t (5,000 egység/ml; NEB) adtunk hozzá, összekevertük és 20 percig 37 °C-on melegítettük, hogy az enzim levágja az RNS-t a DNS-ben: RNS hibrid, amely hatékonyan felszabadítja a cél RNS-t. Az RNáz H-t termikusan inaktiváltuk 65 °C-on 10 percig tartó inkubálással. A vezető oligokat a NUPACK szoftverrel validáltuk, hogy nem képeznek intramolekuláris struktúrákat, homo- vagy heterodimereket45. A hiányzó célponttal végzett méréshez ugyanazt a protokollt használtuk, beleértve a vezető oligokat. A kontroll mérések nem mutatnak elmozdulást, így kizárhatunk bármilyen lényeges keresztkötést a vezető oligókból.
Vírusos célszekvencia tulajdonságai nanocsalihoz
A cél hosszát, a lábujj hosszát és a GC-tartalmat úgy választottuk meg, hogy biztosítsuk az optimális hibridizációt21. A DNS nanobait tervekhez a célszekvenciákat úgy választottuk ki, hogy a vírusgenom konzervált régióiban legyenek, és 40-60% GC tartalommal rendelkezzenek, hogy stabil 20 bp duplexet hozzanak létre. A lábujjtartó hosszát 6 nt hosszúra és 40-60% GC-tartalomra választottuk. Az összes szekvenciát teszteltük a potenciális nemkívánatos, rendkívül stabil intramolekuláris kölcsönhatások vagy homodimerek szempontjából a NUPACK szoftverrel (web alkalmazás 2020)45. Ezután keresztreaktivitás-ellenőrzést végeztünk az egyes kísérletekben alkalmazott több hely között45.
DNS virág előkészítése nanocsalihoz
A SARS-CoV-2 RNS kimutatására a betegmintákból DNS-virágot terveztünk. Három, minden SARS-CoV-2 célpontra specifikus DNS-virágot (hétirányú csomópontok, 7WJa, 7WJb és 7WJc) külön-külön készítettünk. Példaként 7WJc, 4 μM DNS szál J1, J2, J3 és J4c (kiegészítő táblázat 1) összekevertük TM pufferben (10 mM Tris-HCl, 10 mM MgCl2, pH 8.0) és 90 °C-ra melegítjük 5 percig, majd lehűtjük 65 °C-ra 15 percre, 45 °C-ra 15 percre, 37 °C-ra 20 percre, 25 °C-ra 20 percre, végül 4 °C-ra. C-on 20 percig. A J4c szálat J4b-vel helyettesítettük a 7WJb előállítására. A 7WJa esetében, hogy elkerüljük a nanocsali 43. helyén az önhajtást, a J1, J2, J3 J4a és C43 összekevertük a lágyítás előtt.
DNS nanocsali önösszeállítása
A DNS nanocsalit úgy állították össze, hogy linearizált egyszálú M13 DNS-t (M13mp18, 7,249 nt, Guild Biosciences, 100 nM) rövid, komplementer oligonukleotidokkal összekevertünk.12 (amelyek némelyike referenciastruktúrákat és befogó szálakat tartalmazott), valamint részlegesen komplementer szálak hozzáadásával, amelyek 3'-biotinileztek a toehold által közvetített száleltolódási reakcióhoz. A linearizált M13 DNS-t (7,228 nt hosszúságú) oligonukleotidokkal egészítettük ki, ezáltal egy bevágott, kétszálú nanocsalit hoztunk létre, két-terminális négy dezoxitimidin túlnyúlással, amelyek megakadályozzák a multimerizációt.12. A keverék 20 nM linearizált M13 DNS-t, 60 nM oligonukleotidot (az M13 DNS-hez képest háromszoros feleslegben), 3'-biotinilált szálakat tartalmazott 180 nM koncentrációban, 10 mM MgCl-t.2 és 1×TE (10 mM Tris-HCl, 1 mM EDTA, pH 8.0). Pipettázással összekeverjük, majd lecentrifugáljuk, majd 70 másodpercig 30 °C-ra melegítjük, majd 45 perc alatt lehűtjük környezeti hőmérsékletre. A felesleges oligonukleotidokat Amicon Ultra 0.5 ml-es centrifugális szűrőkkel távolítottuk el 100 kDa vágási határértékkel mosópufferrel (10.0 mM Tris-HCl pH 8.0, 0.5 mM MgCl).2). Ha DNS-virágokat használtunk jelölésként, az azt hordozó, részben komplementer szálakat 10 mM MgCl-ben inkubáltuk.2 2 órán át szobahőmérsékleten, majd az Amicon szűrést a fent leírtak szerint végeztük. A nanobait kialakítás aszimmetriája lehetővé teszi a kötőhelyek egyértelmű azonosítását. A nanocsalit a további kísérletekhez való felhasználásig 4-10 °C-on tároltuk 0.5 mM MgCl-ben.210.0 mM Tris-HCl, pH 8.0. A nanocsali tervezését minden mérés előtt nanopórus leolvasással ellenőriztük.
A DNS nanocsali nanopórus-leolvasása
A nanocsalit egy mintával (nukleinsav-kivonat vagy tisztított víruscélpontok tízszeres feleslegben) kevertük 10 mM MgCl-ben.2 és 100 mM NaCl. A keveréket (5 μl) szobahőmérsékleten (~10 perc) inkubáltuk a nanopórusméréshez. A célszekvencia összetételének és fizikai jellemzőinek különbsége a hibridizáció változékonyságához vezethet, és ezáltal az érzékelő helyek eltolási hatékonyságához.21. htRNS-t használtunk (100 ng μl-1; Invitrogen) háttérként, ahol jeleztük, annak bizonyítására, hogy nincsenek a humán natív RNS-ek által indukált nem specifikus jelek. A nanopórusméréshez a mintát <0.5 nM nanobaitre hígítottuk (tisztított víruscélpontok esetén), vagy 4.7 μl RNáz-H-vágott betegmintát kevertünk össze 0.3 µl monovalens sztreptavidinnel (SAe1D3).18 (1 μM), 5 μl LiCl (4.0 M) és 5.0 μl LiCl (8.0 M). 14 ± 3 nm-es (átlag ± standard deviáció) nanopórusokat állítottunk elő12 0.5 mm külső átmérőjű és 0.2 mm belső átmérőjű kvarcüveg kapillárisok (Sutter Instrument) felhasználásával lézerrel segített P-2000 lehúzóval (Sutter Instrument). A keveréket nanopórusos polidimetilsziloxán chipbe pipettáztuk, és minden mérést állandó 600 mV feszültség mellett végeztünk. A nanopórus mérés részletei a kiegészítő táblázatban láthatók 30.
Valós idejű nanopórus adatok elemzése
A nanopórus adatok elemzését a Kiegészítő részben ismertetjük részletesen 14. Röviden, a nanobait eseményeket kiszűrtük a nyers ionáram nyomaiból, majd meghatároztuk a detektálási régiót, és kinyertük a tüske jelenlétéről szóló információkat az egyes helyeken. Az ábrázolt eltolási hatékonyságot úgy számítottuk ki, mint egy mérés elmozdulási hatékonyságát, amelyből levontuk a cél nélküli kontrollt minden egyes helynél (50 nanobait esemény mindhárom nanopórusos felvételnél), hacsak nincs másképp jelezve:
$$begin{array}{l}{mathrm{Displacement}},{mathrm{efficiency}} =frac{1}{3}mathop {sum}limits_{n = 1}^3 balra{ {1 -frac{1 }{{50}}mathop {sum}limits_{n = 1}^{50} {left[ {fleft( n right) = left( {frac{{1,,mathrm{peak}}}{{0,, {mathrm{no}},{mathrm{peak}}}}} right)} right]_{{{{mathrm{target}}}}}} } right}\ – frac{1}{3}mathop {sum }limits_{n = 1}^3 {left{ {1 – frac{1}{{50}}mathop {sum }limits_{n = 1}^{50} left[ {fleft( n right) = left( { frac{{1,,mathrm{peak}}}{{0,,mathrm{no}},{mathrm{peak}}}} right)} right]_{{{{mathrm{no}}}},{ {{mathrm{target}}}}}} right}} end{array}.$$
Ellenőriztük, hogy a konvolúciós neurális hálózat QuipuNet27 képes volt a nanopórus adatok valós idejű elemzésére a leírt eljárást követve. Korábban bemutattuk, hogy körülbelül tíz eseménnyel elérjük a 99%-os megbízhatóságot a tervezett DNS-struktúráink pozitív kimutatásában.46.
AFM képalkotás
A nanocsalik AFM (Nanosurf Mobile S) leképezését levegőben, érintésmentes módban végeztük. A nanobait szerkezeteket 1 ng μl-re hígítottuk-1 1 mM MgCl-ban2 és 10 μl-t adtunk a frissen hasított csillámhoz, 1 percig inkubáltuk, szűrt Milli-Q vízzel öblítettük, majd nitrogénnel szárítottuk. A szkennelés előtt a csillámlemezt kétoldalas ragasztószalaggal az AFM mintaasztalhoz rögzítették. A képvizualizációt és -elemzést a Gwyddion (2.60-as verzió) segítségével végeztük.
Statisztikai analízis
Az összes méréshez 99.9%-os konfidenciaintervallumot számítottunk az eltolási hatékonyságra. A statisztikai szignifikanciát két célpont nélküli és célponttal rendelkező hely között kétoldalas Student-féle módszerrel teszteltük t-teszt.
Jelentési összefoglaló
A kutatástervezésről további információk a Nature Portfolio Reporting Összefoglaló ehhez a cikkhez kapcsolódik.
- SEO által támogatott tartalom és PR terjesztés. Erősödjön még ma.
- Platoblockchain. Web3 metaverzum intelligencia. Felerősített tudás. Hozzáférés itt.
- Forrás: https://www.nature.com/articles/s41565-022-01287-x
- 000
- 1
- 10
- 100
- 11
- 110
- 2014
- 2016
- 2017
- 2018
- 2020
- 2021
- 7
- 70
- 9
- a
- felett
- hiányzó
- egyetemi
- szerzett
- hozzáadott
- Után
- AIR
- Minden termék
- lehetővé teszi, hogy
- Környező
- elemzés
- Horgony
- és a
- Másik
- Alkalmazás
- megközelítés
- megfelelő
- körül
- cikkben
- összeszerelt
- elérhető
- vissza
- háttér
- előtt
- Csengő
- között
- kötés
- fúj
- BP
- tömören
- ütköző
- számított
- képes
- elfog
- visz
- Vizsgázott
- csatorna
- jellemzők
- ellenőrizze
- chen
- csip
- világos
- Klinikai
- gyűjtemény
- kombinált
- kiegészítő
- koncentráció
- bizalom
- MEGERŐSÍTETT
- következetes
- állandó
- tartalom
- ellenőrzés
- ellenőrzések
- konvolúciós neurális hálózat
- létrehozása
- Jelenlegi
- görbe
- vágás
- vágás
- ciklusok
- dátum
- adatelemzés
- igazolták
- leírt
- Design
- tervezett
- tervek
- részlet
- részletek
- Érzékelés
- Határozzuk meg
- eltökélt
- eltérés
- különbség
- digitálisan
- közvetlenül
- terjesztés
- dna
- le-
- minden
- hatékonyan
- hatékonyság
- hatékonyság
- Mérnöki
- biztosítására
- hiba
- Eter (ETH)
- Még
- események
- példa
- magyarázható
- kivonat
- kitermelés
- kivonatok
- Szűrők
- Végül
- vezetéknév
- virág
- követ
- következő
- forma
- Előre
- ból ből
- Tele
- további
- genom
- üveg
- útmutató
- nagyon
- HTTPS
- emberi
- hibrid
- Azonosítás
- kép
- Leképezés
- végrehajtás
- in
- beleértve
- Beleértve
- inkubált
- INKUBÁCIÓ
- információ
- műszer
- kölcsönhatások
- belső
- ión
- IT
- KDA kiterjesztése
- Címke
- vezet
- Hossz
- LINK
- összekapcsolt
- Hosszú
- Luna
- gép
- mester
- anyag
- mérések
- Csillámpala
- esetleg
- minimum
- vegyes
- Keverés
- keverék
- ML
- Mobil
- Mód
- MOL
- többszörös
- nano
- nanopórus
- bennszülött
- Természet
- negatív
- hálózat
- ideg-
- neurális hálózat
- nyitva
- optimálisan
- másképp
- párosítás
- beteg
- betegek
- teljesítmény
- fizikai
- kép
- műanyag
- Plató
- Platón adatintelligencia
- PlatoData
- portfolió
- pozitív
- pozitivitás
- potenciális
- Készít
- előkészített
- jelenlét
- megakadályozása
- korábban
- primer
- szonda
- folyamat
- ingatlanait
- Fehérje
- protokoll
- világítás
- Nyers
- el
- reakció
- real-time
- vidék
- régiók
- Releases
- megmaradó
- eltávolított
- Jelentő
- kutatás
- Eredmények
- fordított
- RNS
- roche
- Szoba
- rt
- szabályok
- futás
- Biztonság
- azonos
- SARS koronavírus-2
- letapogatás
- Rész
- kiválasztott
- Sorozat
- rövid
- előadás
- mutatott
- jelek
- jelentőség
- egyidejű
- egyetlen
- weboldal
- Webhely (ek)
- kicsi
- szoftver
- néhány
- különleges
- fonott
- stabil
- Színpad
- állvány
- standard
- meghatározott
- statisztikai
- Lépései
- memorizált
- Pászmák
- Később
- lényeges
- Systems
- táblázat
- bevétel
- cél
- célok
- sablon
- tíz
- A
- ezáltal
- három
- alkalommal
- TM
- nak nek
- együtt
- Végösszeg
- vágány
- Ultra
- alatt
- egységek
- Egyetemes
- érvényesített
- érték
- Értékek
- ellenőrzött
- változat
- keresztül
- vírusok
- megjelenítés
- Feszültség
- W
- Víz
- háló
- webalkalmazás
- Wells
- ami
- nélkül
- wu
- zephyrnet