24 (Nanowerk-Neuigkeiten) Das Erbmolekül DNA kann auf engstem Raum sehr viele Informationen über lange Zeiträume speichern. Seit gut zehn Jahren verfolgen Wissenschaftler daher das Ziel, DNA-Chips für die Computertechnik zu entwickeln, etwa zur Langzeitarchivierung von Daten. Solche Chips wären herkömmlichen Chips auf Siliziumbasis hinsichtlich Speicherdichte, Langlebigkeit und Nachhaltigkeit überlegen.
In einem DNA-Strang finden sich vier wiederkehrende Grundbausteine. Eine bestimmte Abfolge dieser Blöcke kann zur Verschlüsselung von Informationen verwendet werden, genau wie die Natur es tut. Um einen DNA-Chip zu bauen, muss die entsprechend kodierte DNA synthetisiert und stabilisiert werden. Funktioniert das gut, bleiben die Informationen sehr lange erhalten – Forscher gehen von mehreren tausend Jahren aus. Die Informationen können durch automatisches Auslesen und Dekodieren der Reihenfolge der vier Grundbausteine abgerufen werden.
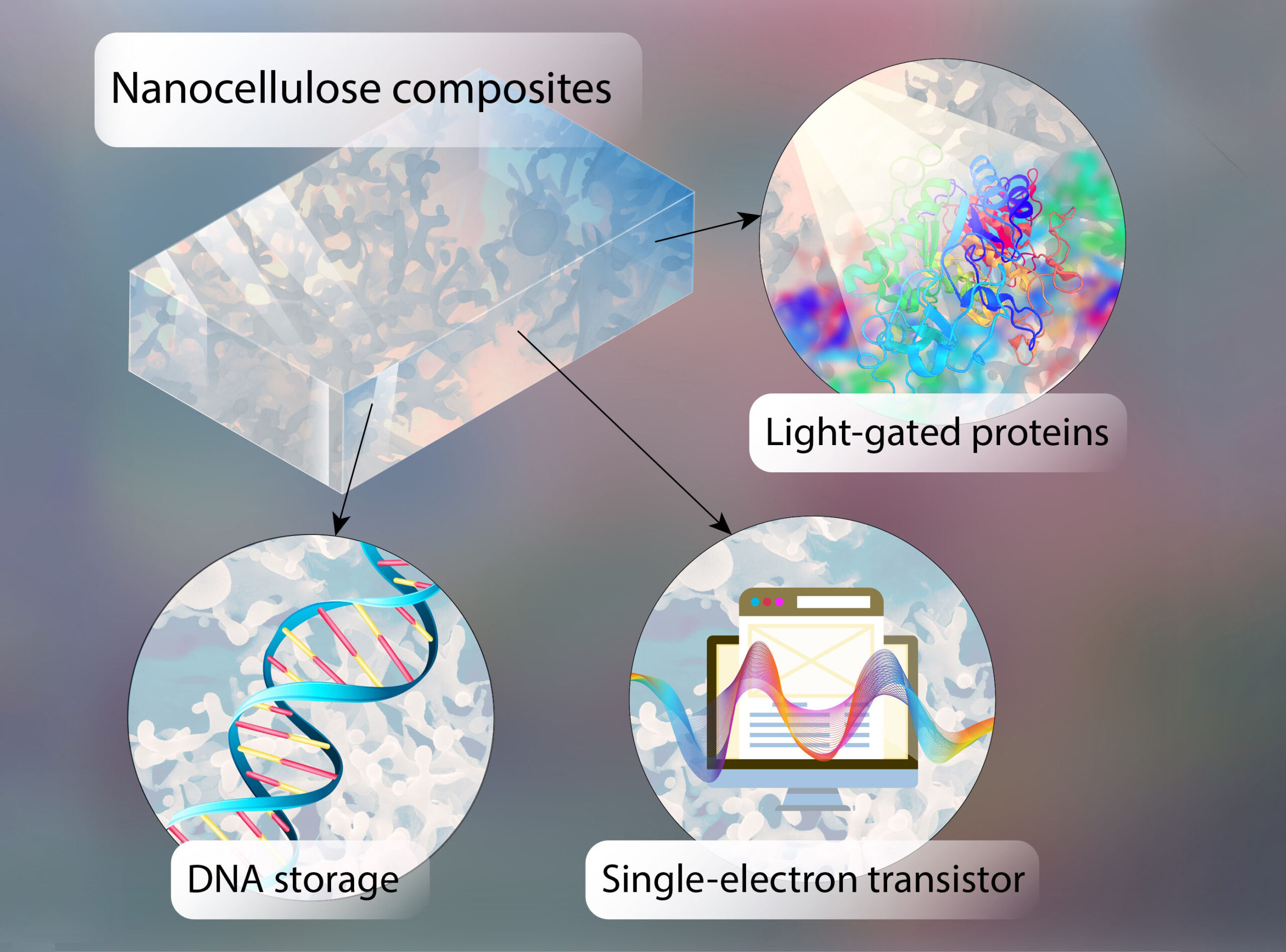
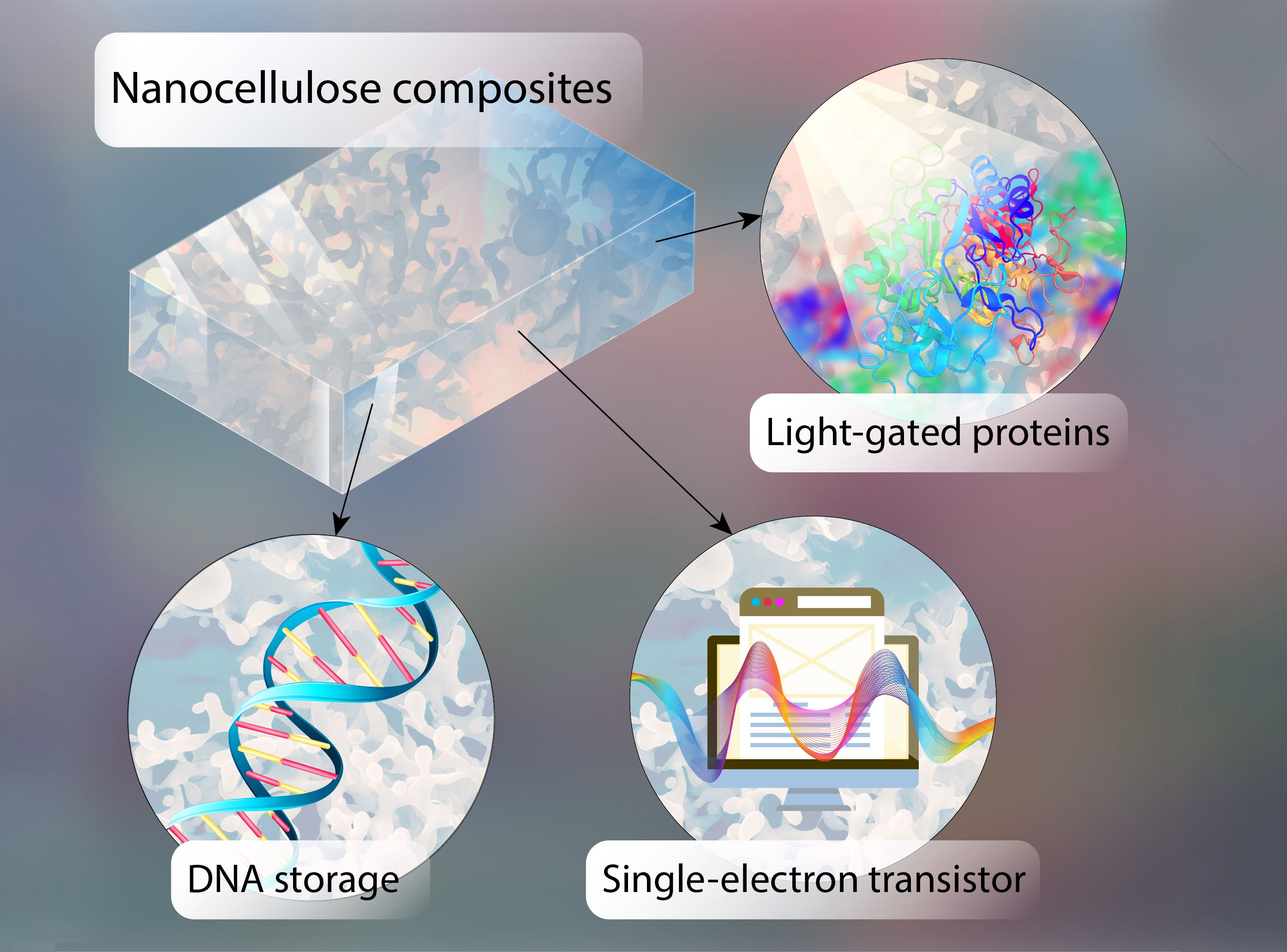 Auf Chips aus halbleitender Nanozellulose können Informationen in Form von DNA gespeichert werden. Lichtgesteuerte Proteine lesen die Informationen. (Bild: Universität Würzburg)
Auf Chips aus halbleitender Nanozellulose können Informationen in Form von DNA gespeichert werden. Lichtgesteuerte Proteine lesen die Informationen. (Bild: Universität Würzburg)
 Auf Chips aus halbleitender Nanozellulose können Informationen in Form von DNA gespeichert werden. Lichtgesteuerte Proteine lesen die Informationen. (Bild: Universität Würzburg)
Auf Chips aus halbleitender Nanozellulose können Informationen in Form von DNA gespeichert werden. Lichtgesteuerte Proteine lesen die Informationen. (Bild: Universität Würzburg)
Welche Herausforderungen müssen gemeistert werden
„Dass eine digitale DNA-Datenspeicherung mit hoher Kapazität und langer Lebensdauer möglich ist, wurde in den letzten Jahren mehrfach bewiesen“, sagt Professor Thomas Dandekar, Leiter des Lehrstuhls für Bioinformatik an der Julius-Maximilians-Universität (JMU) Würzburg. „Aber die Speicherkosten sind hoch, fast 400,000 US-Dollar pro Megabyte, und die in der DNA gespeicherten Informationen können nur langsam abgerufen werden. Je nach Datenmenge dauert es Stunden bis Tage.“ Diese Herausforderungen müssen bewältigt werden, um zu schaffen DNA-Datenspeicherung anwendbarer und marktfähiger. Geeignete Werkzeuge hierfür sind lichtgesteuerte Enzyme und Software zum Design von Proteinnetzwerken. Thomas Dandekar und seine Vorsitzendenteammitglieder Aman Akash und Elena Bencurova diskutieren dies in einer aktuellen Rezension in der Zeitschrift Trends in der Biotechnologie („Wie man die DNA-Datenspeicherung anwendbarer macht“). Dandekars Team ist überzeugt, dass DNA als Datenspeicher eine Zukunft hat. In der Zeitschrift zeigen die JMU-Forscher, wie eine Kombination aus Molekularbiologie, Nanotechnologie, neuartige Polymere, Elektronik und Automatisierung, gepaart mit systematischer Entwicklung, könnten in wenigen Jahren eine alltagstaugliche Speicherung von DNA-Daten ermöglichen.DNA-Chips aus Nanozellulose
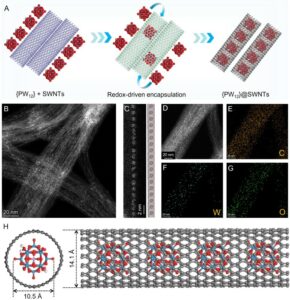
Im JMU Biocenter entwickelt Dandekars Team DNA-Chips aus halbleitenden, bakteriell hergestellten Materialien Nanocellulose. „Mit unserem Proof of Concept können wir zeigen, wie aktuelle Elektronik und Computertechnik teilweise durch molekularbiologische Komponenten ersetzt werden können“, sagt der Professor. Dadurch könnten Nachhaltigkeit, vollständige Recyclingfähigkeit und hohe Robustheit auch gegenüber elektromagnetischen Impulsen oder Stromausfällen erreicht werden, aber auch eine hohe Speicherdichte von bis zu einer Milliarde Gigabyte pro Gramm DNA. Thomas Dandekar bewertet die Entwicklung von DNA-Chips als äußerst relevant: „Wir werden als Zivilisation auf längere Sicht nur überleben, wenn wir den Sprung in diese neue Art nachhaltiger Computertechnologie schaffen, die Molekularbiologie mit Elektronik und neuer Polymertechnologie verbindet.“ Was für die Menschheit wichtig sei, sei der Übergang zu einer Kreislaufwirtschaft im Einklang mit den Grenzen des Planeten und der Umwelt, sagte er. „Das müssen wir in 20 bis 30 Jahren erreichen. Die Chiptechnologie ist dafür ein wichtiges Beispiel, doch die nachhaltigen Technologien zur Herstellung von Chips ohne Elektroschrott und Umweltverschmutzung sind noch nicht ausgereift. Unser Nanozellulose-Chip-Konzept leistet dazu einen wertvollen Beitrag. Im neuen Paper haben wir unser Konzept kritisch hinterfragt und mit aktuellen Innovationen aus der Forschung weiterentwickelt.“Weitere Verbesserung der DNA-Speichermedien
Dandekars Team arbeitet derzeit daran, die DNA-Chips aus halbleitender Nanozellulose noch besser mit den von ihnen entwickelten Designer-Enzymen zu kombinieren. Auch die Enzyme müssen weiter verbessert werden. „Damit wollen wir eine immer bessere Kontrolle über das DNA-Speichermedium erreichen und noch mehr darauf speichern können, aber auch Kosten sparen und so Schritt für Schritt eine praktische Nutzung als Speichermedium im Alltag ermöglichen.“Drei weitere Veröffentlichungen zum Thema
Bencurova E, Shityakov S, Schaack D, Kaltdorf M, Sarukhanyan E, Hilgarth A, Rath C, Montenegro S, Roth G, Lopez D, Dandekar T. Nanozellulose-Verbundwerkstoffe als intelligente Geräte mit Chassis, lichtgesteuerter DNA-Speicherung, technische elektronische Eigenschaften und Chip-Integration. Vorderseite Bioeng Biotechnol. 2022. August 8;10:869111. doi: 10.3389/fbioe.2022.869111 Salihoglu R, Srivastava M, Liang C, Schilling K, Szalay A, Bencurova E, Dandekar T. PRO-Simat: Simulations- und Designtool für Proteinnetzwerke. Comput Struct Biotechnol J. 2023;26:21-2767. doi: 2779/j.csbj.10.1016 Bencurova E, Akash A, Dobson RCJ, Dandekar T. DNA-Speicherung – von der natürlichen Biologie zur synthetischen Biologie. Comput Struct Biotechnol J. 2023 2. Februar;21:1227-1235. doi: 10.1016/j.csbj.2023.01.045- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Automobil / Elektrofahrzeuge, Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- ChartPrime. Verbessern Sie Ihr Handelsspiel mit ChartPrime. Hier zugreifen.
- BlockOffsets. Modernisierung des Eigentums an Umweltkompensationen. Hier zugreifen.
- Quelle: https://www.nanowerk.com/nanotechnology-news2/newsid=63534.php
- :hast
- :Ist
- :nicht
- $UP
- 000
- 07
- 10
- 11
- 20
- 2022
- 2023
- 24
- 26
- 30
- 7
- 8
- 9
- a
- Fähig
- Erreichen
- erreicht
- advanced
- gegen
- ebenfalls
- ein Mann
- Betrag
- an
- und
- anwendbar
- Apr
- SIND
- AS
- annehmen
- At
- August
- Im Prinzip so, wie Sie es von Google Maps kennen.
- Automation
- basic
- BE
- war
- Besser
- Milliarde
- Biologie
- Blockiert
- Grenzen
- bauen
- Building
- aber
- by
- CAN
- Kapazität
- Center
- Vorsitzende
- Herausforderungen
- Chassis
- Chip
- Pommes frites
- Kreislaufwirtschaft
- Menu
- codiert
- Kombination
- Vereinigung
- Komponenten
- Computer
- konzept
- Beitrag
- Smartgeräte App
- konventionellen
- überzeugt
- Kosten
- könnte
- gekoppelt
- Strom
- Zur Zeit
- technische Daten
- Datenspeichervorrichtung
- Datum
- Tage
- Deal
- Decoding
- Synergie
- Dichte
- Abhängig
- Design
- Designer
- entwickelt
- Entwicklung
- Entwicklung
- Geräte
- digital
- diskutieren
- Frau
- die
- Dollar
- e
- Wirtschaft
- elektronisch
- Elektronik
- ermöglichen
- Arbeitsumfeld
- Umwelt-
- Äther (ETH)
- Sogar
- jeden Tag
- Beispiel
- Tatsache
- möglich
- Februar
- wenige
- Aussichten für
- unten stehende Formular
- gefunden
- vier
- für
- voller
- weiter
- Zukunft
- Kundenziele
- gut
- Gramm
- groß
- Harmonie
- Haben
- he
- ganzer
- GUTE
- hoch
- seine
- STUNDEN
- Ultraschall
- Hilfe
- HTTPS
- Menschlichkeit
- if
- Image
- wichtig
- verbessert
- Verbesserung
- in
- Information
- Innovationen
- Integration
- in
- IT
- Zeitschrift
- jpg
- nur
- Nachname
- Springen
- Lebensdauer
- Lebensdauer
- Lang
- lange Zeit
- langfristig
- länger
- Langlebigkeit
- gemacht
- um
- MACHT
- reifen
- Medien
- mittlere
- Mitglieder
- Mitte
- Molekular-
- Molekül
- Montenegro
- mehr
- schlauer bewegen
- sollen
- Natürliche
- Natur
- Need
- Netzwerk
- Neu
- Roman
- of
- on
- EINEM
- einzige
- or
- UNSERE
- übrig
- Überwinden
- Papier
- für
- Zeiträume
- PHP
- Plato
- Datenintelligenz von Plato
- PlatoData
- Luftverschmutzung
- Polymer
- Polymere
- möglich
- Werkzeuge
- Praktisch
- produziert
- Produziert
- Professor
- Beweis
- Proof of Concept
- immobilien
- Proteine
- Proteine
- Publikationen
- R
- Honorar
- Lesen Sie mehr
- Lesebrillen
- kürzlich
- wiederkehrend
- relevant
- ersetzt
- Forschungsprojekte
- Forscher
- Überprüfen
- Robustheit
- s
- Said
- Speichern
- sagt
- Wissenschaftler
- Reihenfolge
- mehrere
- erklären
- Simulation
- Langsam
- klein
- smart
- Software
- Raumfahrt
- spezifisch
- Schritt
- Lagerung
- speichern
- gelagert
- so
- geeignet
- Oberteil
- Nachhaltigkeit
- nachhaltiger
- synthetisch
- T
- nimmt
- Team
- Teammitglieder
- Technologies
- Technologie
- zehn
- Begriff
- AGB
- zur Verbesserung der Gesundheitsgerechtigkeit
- Das
- die Informationen
- deswegen
- Diese
- vom Nutzer definierten
- fehlen uns die Worte.
- So
- Zeit
- mal
- zu
- Werkzeug
- Werkzeuge
- tippe
- Universität
- us
- US-Dollar
- -
- benutzt
- wertvoll
- sehr
- wollen
- Weg..
- we
- GUT
- Was
- Was ist
- werden wir
- mit
- ohne
- arbeiten,
- Werk
- würde
- Jahr
- noch
- Zephyrnet