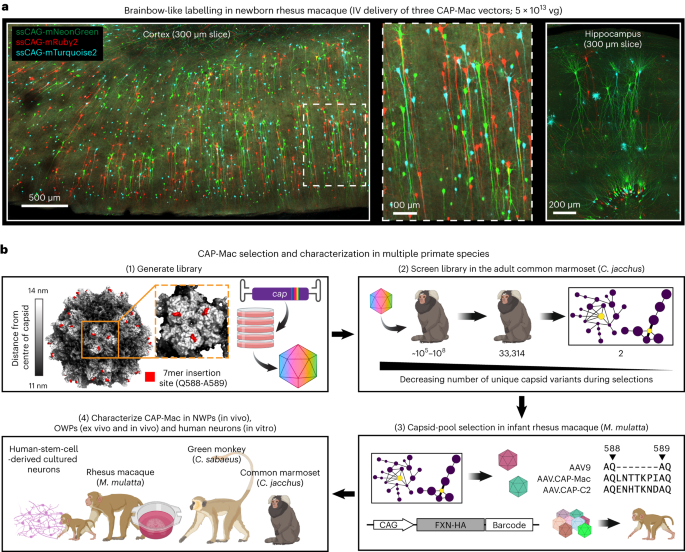
所有实验和程序均得到当地监管委员会和委员会的批准,并必须遵守研究方案。所有小鼠程序均在加州理工学院进行,并得到加州理工学院动物护理和使用委员会(IACUC;方案 1738)的批准。狨猴(方案 TGC-03)和成年猕猴(方案 LN-14)程序在 NIH 进行,并得到 NIH IACUC 的批准。狨猴程序也在加州大学圣地亚哥分校 (UCSD) 完成(方案 S09147),并且符合 UCSD IACUC 并得到其批准。婴儿猕猴手术在加州大学戴维斯分校的加州国家灵长类动物研究中心进行,并得到当地 IACUC 的批准(协议 22525)。绿猴手术在 Virscio 进行,并得到当地 IACUC 的批准。
AAV DNA 文库生成
该过程的详细信息可以在protocols.io 上找到(https://doi.org/10.17504/protocols.io.5jyl8jy89g2w/v1)。我们最初在 DNA 水平上产生多样性,然后用它来生产转染材料以产生 AAV 衣壳文库,如之前详细描述的16。对于第一轮文库,我们使用包含插入到氨基酸 588 和 589 之间的简并核苷酸的引物引入了这种遗传多样性(参考文献 XNUMX)。 12,13,16)(VP1编号;补充图。 1a)。我们使用含有 7 个简并核苷酸 ([NNK] × 7) 的反向引物随机生成聚合酶链式反应 (PCR) 片段,其中包含插入到 帽 基因。对于第二轮 DNA 文库,我们使用合成寡核苷酸库 (Twist Bioscience) 作为反向引物,仅编码选择用于进一步筛选的变体(总共 66,628 DNA 寡核苷酸;第一轮选择后恢复的 33,314 变体加上密码子修饰)每个的复制品)。所有反向引物均包含与引物互补的 20 bp 5' 突出端 帽 序列靠近 AgeI 限制性酶序列,并与含有靠近 XbaI 限制性酶序列的 20bp 5' 突出端的正向引物配对。然后,我们通过 Gibson 组装将包含多样化区域的 PCR 片段插入 rAAV-ΔCAP-in-cis-Lox 质粒中,使用 NEBuilder HiFi DNA Assembly 生成所得的 AAV DNA 文库,即 rAAV-CAP-in-cis-Lox预混液(新英格兰生物实验室,E2621)。
AAV衣壳文库制作
该过程的详细信息可以在protocols.io 上找到(https://doi.org/10.17504/protocols.io.5jyl8jyz9g2w/v1)。我们根据之前发布的协议生成了 AAV 衣壳库16,70。简而言之,我们使用转染级线性聚乙烯亚胺(PEI;Polysciences)在 293mm 组织培养板中转染 HEK3216T 细胞(ATCC,目录号 CRL-0063;RRID:CVCL_150)。在每个板中,我们转染了四个质粒:(1)组装的rAAV-Cap-in-cis-Lox AAV DNA文库,其两侧是AAV衣壳化所需的反向末端重复序列; (2) AAV2/9 REP-AAP-ΔCAP,编码 AAV 生产所需的 REP 和 AAP 补充蛋白,其 C 端为 帽 切除基因以防止与 AAV DNA 文库重组以及随后产生具有复制能力的 AAV; (3) pHelper,编码AAV生产所需的必需腺病毒蛋白; (4) pUC18 (Addgene ID: 50004; RRID: Addgene_50004),它不含哺乳动物表达载体,但用作填充 DNA,以实现最佳 PEI 转染所需的适当氮磷比。在制备 PEI-DNA 混合物的过程中,我们为每个 10mm 培养皿添加了 150ng 的 AAV DNA 文库 (rAAV-Cap-in-cis-Lox),并将 AAV2/9 REP-AAP-ΔCAP、pUC18 和 pHelper 组合在一个1:1:2 比例(每 40mm 培养皿 150μg 总 DNA)。转染后 60 小时,我们使用聚乙二醇沉淀和碘克沙醇梯度超速离心从细胞沉淀和培养基中纯化 AAV 衣壳文库。然后,我们使用定量 PCR,根据既定方案,通过相对于线性化基因组标准扩增 DNaseI 抗性病毒基因组,确定 AAV 衣壳文库的滴度70.
狨猴实验
衣壳文库选择
该过程的详细信息可以在protocols.io 上找到(https://doi.org/10.17504/protocols.io.bp2l695zklqe/v2)。所有狨猴(雅库斯)程序在国家心理健康研究所 (NIMH) 进行,并得到当地 IACUC 的批准。狨猴在 NIMH 群体中出生和长大,并在 27°C 和 50% 湿度的标准条件下以家庭群体的形式饲养。作为 NIH 灵长类动物富集计划的一部分,它们被随意喂养并接受富集。对于本研究中使用的所有狨猴,静脉输注前 1:5 血清稀释液中没有检测到中和抗体(由宾夕法尼亚大学 Penn Vector Core 测定)。然后,他们被单独安置几天,并在注射前适应新房间。四名成年男性用于文库筛选,第一轮和第二轮文库各两名。输注前一天,动物的食物被移除。在氧气中用异氟烷麻醉动物,将股静脉上的皮肤剃毛并用异丙醇擦洗和 2 × 10 消毒。12 AAV 衣壳文库的 vg 在几分钟内注入。撤除麻醉并对动物进行监测,直到它们变得活跃,然后将它们放回笼子。在接下来的 3 天内,密切监测活动和行为,此后每天进行观察。
注射后 4 周,对狨猴实施安乐死(Euthanasia,VetOne)并灌注 1× 磷酸盐缓冲盐水 (PBS)。第一轮文库后,将大脑切成四个冠状块,在2-甲基丁烷(Sigma-Aldrich,M32631)中快速冷冻,用干冰冷冻,并保存在-80℃下长期保存。第二轮文库后,将大脑切成六个冠状块,与脊髓和肝脏切片一起快速冷冻并保存在-80°C下以供长期保存。
狨猴中 AAV 的个体特征
本节中的过程的详细信息可以在protocols.io上找到(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1 和 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1)。两只成年普通狨猴(雅库斯)用于本实验:柯南(男性,2.8岁,0.386公斤)和桑迪(女性,5.8岁,0.468公斤)(补充表 3 提供更多详细信息)。它们被安置在 27°C 和 50% 湿度的标准条件下,可随意获取食物和水。所有动物均被分组饲养,实验在加州大学圣地亚哥分校的皮质系统和行为实验室进行。所有实验均得到 UCSD IACUC 的批准。输注前一天,动物的食物被移除。
用氯胺酮(Ketaset,Zoetis 043-304,20mg/kg)麻醉动物 - 1),将隐静脉上的皮肤剃毛并用异丙醇擦洗和 2 × 1013 VG kg - 1 AAV 输注时间超过 5 分钟。对这些动物进行监测,直到它们变得活跃,然后将它们放回笼子里。在接下来的 3 天内,密切监测活动和行为,此后每天进行观察。在第1、7、14、21和31天采集血样以测量血浆中的病毒浓度。
注射后 31 天,如前所述用氯胺酮麻醉狨猴,然后安乐死(Euthasol,Virbac 200-071,1mlkg - 1)并用 1× PBS 灌注。将脑和器官切成两半,一半在2-甲基丁烷(Sigma-Aldrich,M32631)中快速冷冻,用干冰冷冻并储存在-80°C。另一半在 4% 多聚甲醛 (PFA)(Thermo Scientific,J19943-K2)中固定过夜,然后在 4°C 下保存在 PBS 叠氮化物(Sigma-Aldrich,S2002-100G,0.025%)中。然后样本被运送到加州理工学院(Caltech)进行分析。对于 GLUT1 染色,我们将切片与兔抗 GLUT1(1:200;Millipore-Sigma,目录号 07-1401;RRID:AB_1587074)一起孵育,用 PBS 进行三到五次洗涤,与驴抗兔 IgG(1:200)一起孵育。 711;杰克逊免疫研究实验室,目录号 605-152-2492288;RRID:AB_0.1)并在封固前清洗三到五次。我们稀释所有抗体并使用补充有 100% Triton X-8787 (Sigma-Aldrich, T10) 和 017% 正常驴血清 (Jackson Nutrition Labs, cat # 000-121-2337258; RRID: AB_XNUMX) 的 PBS 进行过夜孵育。室温并摇动。
病毒库DNA提取和NGS样品制备
该过程的详细信息可以在protocols.io 上找到(https://doi.org/10.17504/protocols.io.bp2l695zklqe/v2)。我们之前报道过,可以使用 TRIzol 通过从水相中沉淀核酸来分离病毒文库 DNA 和内源宿主 RNA12,16。因此,为了从狨猴组织中提取病毒文库 DNA,我们使用 BeadBug(Benchmark Scientific,D100)在 TRIzol(Life Technologies,15596)中均质化 1036mg 脊髓、肝脏和大脑的每个冠状块,并从狨猴组织中分离出核酸。根据制造商推荐的方案进行水相。我们用 RNase(Invitrogen,AM2288)处理重构的沉淀物,并用 SmaI 消化,以通过 PCR 提高下游病毒 DNA 回收率。消化后,我们根据制造商推荐的方案使用 Zymo DNA Clean and Concentrator Kit (D4033) 进行纯化,并将纯化的病毒 DNA 储存在 -20°C 下。
为了在多样化区域的侧翼附加 Illumina 接头,我们首先使用提取的总病毒 DNA 的 7% 作为模板,对包含 50mer 插入的区域进行 PCR 扩增(25 个循环)。 Zymo DNA 纯化后,我们以 1:100 稀释样品,并通过十个循环的 PCR 在可变区周围进一步扩增,为下一个 PCR 反应添加结合区。最后,我们使用 NEBNext 双标签引物(New England Biolabs,E7600)通过十个以上的 PCR 循环附加 Illumina 流动池适配器和独特的标签。然后,我们使用 2% 低熔点琼脂糖凝胶(Thermo Fisher Scientific,16520050)对最终 PCR 产物进行凝胶纯化,并恢复 210bp 条带。
仅对于第二轮文库,我们还分离了封装的 AAV 文库 ssDNA 用于 NGS 来计算文库富集分数,这是我们用来标准化文库中各种变体滴度差异的定量指标(参见参考文献 XNUMX)。 16 以及“NGS 读取对齐、分析和网络图生成”部分)。为了分离衣壳化的病毒基因组,我们用 DNaseI 处理 AAV 衣壳文库,并使用蛋白酶 K 消化衣壳。然后,我们使用苯酚-氯仿纯化 ssDNA,通过两个 PCR 扩增步骤扩增病毒转基因,以添加 Illumina NGS 的接头和索引,并纯化使用凝胶电泳。该病毒文库 DNA 以及从组织中提取的病毒 DNA 被送去使用 Illumina HiSeq 2500 系统(加州理工学院 Millard 和 Muriel Jacobs 遗传学和基因组实验室)进行深度测序。
NGS 读取对齐、分析和生成网络图
NGS 运行的原始 FASTQ 文件使用定制脚本进行处理(https://github.com/GradinaruLab/protfarm 和 https://github.com/GradinaruLab/mCREATE)16。对于第一轮文库,处理这些数据集的流程涉及过滤以删除低质量的读数、利用每个序列的质量评分以及消除 PCR 诱导的突变或高 GC 含量带来的偏差。然后通过完美的字符串匹配算法对过滤后的数据集进行对齐并进行修剪以提高对齐质量。然后,我们显示了每个组织内测序运行期间每个变体的绝对读数计数,并且在大脑中发现的所有 33,314 个变体都被选择用于第二轮选择。
第二轮选择后,我们进行了相同的分析,以显示注射病毒库和每个组织内每个变体的变体绝对读数计数。此外,我们还计算了文库富集度16 对于每个组织内的每个变体:
$${上划线{{rm{RC}}}}_{x,{rm{注入}},{rm{库}}}=,压裂{{{rm{RC}}}_{x,{rm{注入}},{rm{库}}}}{mathop{sum }nolimits_{i=1}^{{N}_{{rm{注入}},{rm{库}}}}{{rm{RC }}}_{i,{rm{注入},{库}}}},$$
(1)
$${上划线{{rm{RC}}}}_{x,{rm{组织}}}=,frac{{{rm{RC}}}_{x,{rm{病毒}}}}{mathop {sum }nolimits_{i=1}^{{N}_{{rm{组织}}}}{{rm{RC}}}_{i,{rm{组织}}}},$$
(2)
$${rm{库},{丰富}}=,{log }_{10}left(frac{{overline{{rm{RC}}}}_{x,rm{{注入},{库}} }}{{上划线{{rm{RC}}}}_{x,{rm{组织}}}}右),$$
(3)
这样对于给定的样本 y (例如,注射的病毒库或组织样本),RCx,y 是变体的绝对读取计数 x, Ny 是恢复的变体总数, ({上划线{{rm{RC}}}}_{x,{y}}) 是标准化的读取计数。
为了构建 CAP-Mac 序列聚类图,我们根据以下标准过滤了第二轮 NGS 数据:(1) 注入文库样本中的读取计数≥100(24,186/33,314 变体),(2) ≥0.7 文库富集度在两个以上的大脑样本(415 变体)中得分,并且(3)与库富集度低于-0.7的大脑样本(0.7 变体)相比,至少多两个具有≥323库富集的大脑样本。为了构建 CAP-C2 序列图,我们根据以下标准过滤了第二轮 NGS 数据:(1) 注入文库样本中的读取计数≥100;(2) 至少两个脑样本中存在两个密码子复制≥0.7 文库富集(95 个变体)。然后独立处理这些变体以确定成对的反向汉明距离(https://github.com/GradinaruLab/mCREATE)并使用 Cytoscape(v. 3.9.0;RRID:SCR_003032)进行聚类,如前面详细描述的16。如果成对反向汉明距离≥3,则所呈现的网络显示由边缘连接的衣壳变体(节点)。
克隆单个 AAV 衣壳变体
该过程的详细信息可以在protocols.io 上找到(https://doi.org/10.17504/protocols.io.n2bvj87ebgk5/v1)。对于单变体表征,我们通过使用 MscI 和 AgeI 消化 pUCmini-iCAP-PHP.eB(Addgene ID:103005;RRID:Addgene_103005)骨架的修改版本来克隆新的变体质粒。我们设计了一个 100bp 引物,其中包含每个衣壳变体所需的 21bp 插入以及与 AAV9 模板互补的区域,其中约 20bp 与消化的主链重叠。然后,我们使用 NEBuilder HiFi DNA Assembly Master Mix 组装变体质粒,将 5μl 200nM 引物与反应混合物中的 30ng 消化骨架混合。用于生产 AAV.CAP-Mac 的衣壳质粒可在 Addgene 上获得(Addgene ID:200658;RRID:Addgene_200658)。
个体AAV的生产和纯化
该过程的详细信息可以在protocols.io 上找到(https://doi.org/10.17504/protocols.io.14egn2dqzg5d/v1)。为了生成用于池测试的变体,我们遵循之前发布的协议70 使用150mm的组织培养皿。对于体内和体外的个体 AAV.CAP-Mac 和 AAV9 表征,我们采用了我们发布的方案,利用十层 CellSTACK(康宁,3320)以高滴度高效生产病毒,以对恒河猴和绿猴进行给药。具体来说,我们在转染前 150 小时将 70 个 24 mm 培养皿以大约 150% 的汇合度传代到 72 层 CellSTACK 中。转染当天,我们为 150 个 120mm 培养皿准备了 PEI-DNA 转染混合物,并将转染混合物与培养基混合,并对 CellSTACK 进行了完整的培养基更换。我们在转染后 15575020 小时收集并更换培养基,类似于 10 毫米培养皿中的生产。转染后 37 小时,我们添加乙二胺四乙酸(Invitrogen,20)至终浓度为 XNUMX mM,并在 XNUMX°C 下孵育 XNUMX 分钟,偶尔旋转并轻敲 CellSTACK 的侧面以分离细胞。然后我们除去培养基和细胞混合物并继续进行 AAV 纯化方案70。值得注意的是,在超速离心后的缓冲液交换步骤中,我们使用带有聚醚砜膜的离心蛋白浓缩器(Thermo Scientific,88533)代替 Amicon 过滤装置,并使用补充有 0.001% Pluronic F-68(Gibco,24040032)的 Dulbecco PBS。
啮齿动物实验
所有啮齿动物手术均在加州理工学院进行,并得到当地 IACUC 的批准。我们购买了 C57BL/6J(品系号:000664;RRID:IMSR_JAX:000664)、BALB/cJ(品系号:000651;RRID:IMSR_JAX:000651)和 DBA/2J(品系号:000671;RRID:IMSR_JAX:000671)小鼠(所有雄性,6-8 周大)来自杰克逊实验室。对于小鼠静脉注射,我们输送了 5 × 1011 病毒通过眼眶后窦的VG70,71 使用 31 号胰岛素注射器(BD,328438)。有关小鼠眼眶后注射 AAV 的更多详细信息,请参阅protocols.io(https://doi.org/10.17504/protocols.io.3byl4joy8lo5/v1)。对于小鼠脑室内(ICV)给药,我们注射了 5.0 × 1010 或 1.5 × 1011 vg进入侧脑室。简而言之,我们使用异氟烷(5% 用于诱导,1-3% 用于维持)和 95% OXNUMX 麻醉小鼠2/ 5%CO2 (1 l 分钟 - 1)并将小鼠头部固定在立体定位框架中。剃光头并用氯己定消毒该区域后,我们皮下注射 0.05mg ml 2.5ml - 1 布比卡因,进行中线切口,清除颅骨上的血液和结缔组织。头部调平后,在侧脑室上方(距前囟后 0.60mm,距中线 1.15mm)两侧钻钻孔。使用10号显微注射针将病毒载体吸入33μl NanoFil注射器(World Precision Instruments)中,并将针缓慢降低到侧脑室(距离软脑膜表面1.6毫米)。允许针就位约 5 分钟,并使用微注射泵(World Precision Instruments,UMP3)和泵控制器(World Precision Instruments,Mircro5)以 3 nl 分钟的速率注射 3-300 µl 病毒载体 - 1。所有小鼠术中给予1 mg kg - 1 丁丙诺啡 SR 和 5 mg kg - 1 皮下注射酮洛芬 30 mg kg - 1 布洛芬和 60 mg kg - 1 术后 5 天服用甲氧苄啶/磺胺甲恶唑。有关在小鼠中 ICV 注射 AAV 的更多详细信息,请参阅protocols.io(https://doi.org/10.17504/protocols.io.5qpvorm4dv4o/v1)。表达3周后,所有小鼠均灌注PBS并固定在4%PFA中。提取所有器官,在4.00%PFA中孵育过夜,转移至补充有0.01%叠氮化钠的PBS中,并在4℃下保存以长期保存。我们使用振动切片机(Leica Biosystems,VT100S)将大脑切成 1200μm 的切片,安装在 Prolong Diamond Antifade(Invitrogen,P36970)中,并使用 ZEN(黑色版)使用共焦显微镜(Zeiss,LSM 880)进行成像。有关组织处理的更多详细信息,请参阅protocols.io(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1 和 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1).
恒河猴实验
本节中的过程的详细信息可以在protocols.io上找到(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1)。新生猕猴(0.45-1.40kg)在出生时断奶。在第一个月内,猕猴通过静脉或鞘内注射 AAV 载体。本研究中的所有成年猕猴(8-17 岁;4.65-11 公斤)仅通过静脉注射注射 AAV。对于IV注射,用氯胺酮(0.10ml)麻醉动物,并将隐静脉上的皮肤剃毛并消毒。 AAV(2 × 10之间13 和 1 × 1014 VG kg - 1)在约1分钟内缓慢注入隐静脉中<0.75ml的PBS中。对于ICM注射,给动物肌肉注射镇静剂,并将颈部皮肤区域剃毛并进行无菌准备。将一根针插入小脑延髓池,取出与注入液体量成比例的少量脑脊液。然后,使用装有 AAV 无菌制剂的无菌注射器(1.5 × 1012 或 2.5 × 1013 VG kg - 1)与收集的液体量成比例,无菌连接并缓慢注射。所有动物在镇静恢复期间全天进行监测,然后每天监测是否有任何不良发现。所有的猴子都被单独安置在同种动物的视线和声音范围内。注射后4-11周收集组织。根据加州国家灵长类动物研究中心的动物人道安乐死指南,对这些动物进行深度麻醉并接受戊巴比妥钠治疗。注射到恒河猴体内的所有材料均不含内毒素(<0.1 EU ml - 1),并通过十二烷基硫酸钠-聚丙烯酰胺凝胶电泳证实蛋白质纯度。补充表 4 和 5 列出每个实验的给药途径、AAV 变体、病毒剂量、遗传货物和表达持续时间。
恒河猴池测试
本节中的过程的详细信息可以在protocols.io上找到(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1, https://doi.org/10.17504/protocols.io.3byl4jo68lo5/v1 和 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1)。新生猕猴池实验(RM-001 至 RM-004)在加州大学戴维斯分校的 CNPRC 进行,并得到当地 IACUC 的批准。成年猕猴池实验(RMN-001 和 RMN-002)在 NIMH 进行,并得到当地 IACUC 的批准。用冰冷的无 RNase PBS 灌注猕猴。灌注时,大脑的一个半球被快速冷冻,另一个半球被切成4毫米的冠状块,并在4% PFA中后固定48小时,然后转移到加州理工学院进行进一步处理。对于 HA 染色,我们将切片与兔抗 HA(1:200;Cell Signaling Technology,目录号 3724;RRID:AB_1549585)一起孵育,用 PBS 进行三到五次洗涤,与驴抗兔 IgG(1:200;RRID:AB_711)一起孵育。 Jackson ImmunoResearch Labs,目录号 605-152-2492288;RRID:AB_0.1)并在封固前清洗三到五次。我们稀释了所有抗体,并使用补充有 100% Triton X-8787(Sigma-Aldrich,T10)和 017% 正常驴血清(Jackson Nutrition Labs,目录号 000-121-2337258;RRID:AB_XNUMX)的 PBS 进行所有孵育。室温下摇晃过夜。
为了分离病毒 DNA 和全 RNA,使用 BeadBug(Benchmark Scientific,D100)在 TRIzol(Life Technologies,15596)中均质化来自大脑和肝脏的 1036mg 切片,并根据制造商推荐的方案回收总 DNA 和 RNA 。回收的 DNA 用 RNase 处理,用 SmaI 限制性消化,并用 Zymo DNA Clean and Concentrator 试剂盒 (D4033) 纯化。回收的 RNA 用 DNase 处理,并根据制造商推荐的方案,使用 SuperScript III (Thermo Fisher Scientific, 18080093) 和oligo(dT) 引物从 mRNA 生成 cDNA。我们以 50 ng 病毒 DNA 或 cDNA 作为模板,使用 PCR 扩增条形码区域。 Zymo DNA 纯化后,我们以 1:100 稀释样品,并使用引物进一步扩增条形码区域,以附加用于 Illumina NGS 的适配器。净化后,使用用于 Illumina 测序的 NEBNext 双标签引物(New England Biolabs,E7600)进一步扩增这些产物十个循环。然后,我们使用 2% 低熔点琼脂糖凝胶(Thermo Fisher Scientific,16520050)对最终 PCR 产物进行凝胶纯化。池测试富集的计算与文库富集相同,但如图 XNUMX 所示。 2b,c 在线性范围内。
恒河猴 CAP-Mac 的个体特征
本节中的过程的详细信息可以在protocols.io上找到(https://doi.org/10.17504/protocols.io.5qpvormddv4o/v1 和 https://doi.org/10.17504/protocols.io.j8nlkwxxwl5r/v1)。新生猕猴用 PBS 和 4% PFA 进行灌注。将大脑切成4mm的冠状块,所有组织在4%PFA中后固定3天,然后储存在PBS中。用于体内、个体表征的单个成年猕猴(RM-020;17岁,11公斤)用无RNase的PBS灌注,并将一个半球快速冷冻,另一个半球切成4毫米的冠状块,并后固定在 4% PFA 中。所有组织都被转移到加州理工学院进行进一步处理。使用振动切片机将脑和肝脏切成100μm的切片。此外,将脑和脊髓切片在 30% 蔗糖中孵育过夜并包埋在 O.C.T. 中。化合物(Scigen,4586)并使用低温恒温器(Leica Biosystems,CM50)切成1950μm的切片。所有切片均使用 Prolong Diamond Antifade 封片,并使用共焦显微镜成像。对于鞘内给药的猕猴脊髓和脑切片的 GFP 染色,我们将切片与鸡抗 GFP(1:500;Aves Labs,目录号 GFP-1020;RRID:AB_10000240)一起孵育,用 PBS 进行三到五次洗涤,与驴抗鸡 IgY(1:200;杰克逊免疫研究实验室,目录号 703-605-155;RRID:AB_2340379)一起孵育,并在封固前清洗三到五次。我们稀释所有抗体并使用补充有 0.1% Triton X-100 (Sigma-Aldrich, T8787) 和 10% 正常驴血清 (Jackson ImmunoResearch, 017-000-121) 的 PBS 在室温下摇动过夜进行所有孵育。
为了进行形态重建,我们将大脑切成 300μm 的切片,并将它们在折射率匹配溶液中孵育72 72 小时,然后安装在浸入折射率匹配溶液中的载玻片上。我们使用共焦显微镜和 ×25 物镜(LD LCI Plan-Apochromat ×25/0.8 Imm Corr DIC)并使用 100% 甘油作为浸没液进行成像。我们捕获了平铺的 Z 堆栈(使用建议的捕获设置,每帧为 1,024 × 1,024)围绕感兴趣的细胞,并裁剪适当的视野以进行跟踪。追踪是在 Imaris(Oxford Instruments;RRID:SCR_007370)中使用半自动和自动化方法完成的。
对于神经元 (NeuN) 和星形胶质细胞 (S100β) 定量,使用抗 NeuN (EPR12763) 抗体(1:200;Abcam,目录号 ab177487;RRID:AB_2532109)或抗 S100β 抗体(1:200;Abcam)对切片进行染色,目录号 ab52642;RRID:AB_882426)在补充有 0.1% Triton X-100 和 10% 正常驴血清的 PBS 中过夜。用 PBS 洗涤切片 647 至 1 次,并在 PBS+200% 中与 Alexa Fluor 711(605:152;JacksonImmunoResearch Labs,目录号 2492288-0.1-100;RRID:AB_10)缀合的抗兔 IgG 抗体孵育过夜。 Triton X-XNUMX + XNUMX% 正常驴血清。经过三到五次洗涤并使用 Prolong Diamond Antifade 镶嵌后,我们获得了 Z 使用共焦显微镜和×25物镜进行堆叠。我们使用 Python 中的自定义脚本 (RRID: SCR_008394) 和 Cellpose (https://www.cellpose.org/; RRID:SCR_021716)73.
离体双光子成像
使用振动切片机从较大的切片中制备厚度为 400μm 的适合成像的脑切片,并在双光子成像之前储存在用卡波金气体鼓泡的人工脑脊液中,如先前在已发布的协议中所述74,75。为了测试 GCaMP8s 反应,使用距离成像神经元 4-5μm 的细胞外单极电极进行指定脉冲数的电刺激(80-0.3V、100Hz、200s 持续时间)。成像帧率为30Hz。分段感兴趣区域的迹线绘制为 ΔF/F0 = (F(t) − F0)/F0,其中 F0 定义为电刺激前所有荧光值的平均值。上升时间定义为信号的上升阶段从峰值的10%达到峰值的90%所需的时间。通过将指数之和拟合到信号的衰减相位来获得衰减时间常数。通过将信号的峰值幅度除以电刺激前荧光迹线的标准偏差来获得信噪比。
成年恒河猴切片的表征
华盛顿国家灵长类研究中心的一只成年恒河猴(14 岁零 1 个月大;10.83 公斤)计划进行常规安乐死,并作为该设施组织分配计划的一部分收集大脑。将颞上回块切成 300μm 的切片并回收切片74 并在空气-液体膜界面上培养,如前所述76。切片铺板后约 30 分钟,我们施用 1–2μl AAV(5 × 1013 VG ml - 1 AAV9 或 AAV.CAP-Mac 包装 ssCAG-FXN-HA 或 ssCAG-eGFP)。对于每种条件,以生物学一式三份进行实验,并且每48小时更新一次培养基,直到转导后8天收集组织。在组织采集当天,对切片进行成像以确认转导,将切片切成两半,并将每半片在干冰-乙醇浴中快速冷冻。将样品储存在-20℃下直至进一步处理。
每半片均进行处理(DNA 和 RNA 回收各一个)。使用 Qiagen DNeasy 血液和组织试剂盒(Qiagen,目录号 69504)分离 DNA,并使用 TRIzol(Thermo Fisher Scientific,目录号 15596026)和 PureLink RNA Mini 试剂盒(Thermo Fisher Scientific,目录号 12183018A)回收 RNA。通过如下修改 PureLink RNA Mini 试剂盒的第一次洗涤,从 RNA 样品中去除 DNA:用 350 µl 洗涤缓冲液 1 洗涤,然后在 RDD 缓冲液(Qiagen 目录号 # 80)中添加 79254 µl 不含 RNase 的 DNaseI 并孵育室温下过柱15分钟;然后,在继续操作之前再次用 350μl 洗涤缓冲液 1 洗涤。我们使用 Promega GoScript 逆转录试剂盒(Promega,目录号 A400)在 20 µl 反应中从 5000 ng 总 RNA 合成第一链 cDNA。
然后,我们在 Roche Lightcycler II 上使用定量 PCR 评估了每个样本中发现的载体基因组和病毒转录本。这里,使用 Thermo Fisher Scientific 的 TaqMan 探针(EGFP-FAM 探针,测定 ID Mr100_mr,目录号 #20;定制基因组参考探针 CN04097229-4331182-VIC,测定 ID ARH2386DUK,目录号 #2)在 6 µl 扩增反应中使用 4448512 ng DNA。 XNUMX,旨在针对两者 穆拉塔 和 猕猴nemestrina).
绿猴实验
所有的绿猴(鲫鱼) 程序在 Virscio 进行并经 IACUC 批准。对所有猴子进行中和抗体筛查,并确认滴度<1:5。在大约 7-8 个月大(1.0-1.3 公斤)时,对猴子进行静脉注射(补充表 6)。给药前,使剂量制剂平衡至大约室温至少10分钟,但不超过60分钟。 IV剂量体积基于第0天的体重。用氯胺酮(8.0mg/kg)使动物镇静 - 1)和甲苯噻嗪(1.6 mg kg - 1)。将注射区域剃毛并用氯己定和 70% 异丙醇进行准备,并在插入 IV 导管之前进行手术擦洗。通过单次静脉输注 AAV 进行给药(7.5 × 1013 或 7.6 × 1013 VG kg - 1)在第 0 天通过隐静脉使用手持式输液装置以 1 ml min 的目标速率给药 - 1。从给药前 1 周开始,通过笼旁观察每天两次确认总体健康状况。在预定的安乐死时间,用氯胺酮(8-10毫克公斤)对猴子进行镇静。 - 1 肌肉注射)并用戊巴比妥钠(100 mg kg)安乐死 - 1 IV 生效)。角膜反射消失后,使用蠕动泵以约 100ml/min 的速率用冷冻 PBS 进行经心灌注(左心室) - 1 直至流出的液体清澈为止,然后收集组织。从左脑半球和各种其他器官收集组织块,并冷冻在液氮气相中,以便进一步处理以进行生物分布。取出右脑半球并切成约4毫米的冠状切片,并在室温下用约20体积的10%中性缓冲福尔马林完整地后固定约24小时。
使用 Thermo Fisher MagMax DNA Ultra 2.0 提取试剂盒(目录号#A36570)从 CNS 和外周组织中提取基因组 DNA。通过 Qubit dsDNA 测定的荧光定量来评估 DNA 的产量。每个 20μl 反应中加载大约 20ng DNA,并将板在 BioRad CFX Connect 实时 PCR 检测系统(目录号#1855201)上运行。通过检测单个扩增产物来验证病毒拷贝数测定的特异性;灵敏度,通过评估每个反应的检测下限大于十个拷贝;和线性,通过确保标准曲线 R2 >0.95。反应在 FastStart Universal SYBR Green Master (Rox)(目录号 4913850001)中组装。引物的序列为ACGACTTCTTCAAGTCCGCC(正向)和TCTTGTAGTTGCCGTCGTCC(反向)。 PCR 方案使用 95℃ 180 秒的初始变性步骤,然后是 40℃ 95 秒和 15℃ 60 秒的 60 个循环,每个 60℃ 循环后有一个成像步骤。使用包含 1 × 10 病毒中存在的 GFP 模板序列的线性化质粒生成标准曲线8 至 1 × 1010 拷贝,稀释在未经处理的幼猴 DNA 样本中,该样本使用与本研究中的样本相同的试剂盒制备,以控制基质效应。使用最佳拟合线方程从标准曲线计算病毒 DNA 的拷贝数。根据每次反应测量的宿主细胞 DNA 的总基因组重量计算感染多重值。
固定后,将组织放入10%>20%>30%蔗糖中,24℃各4小时,然后包埋在O.C.T中。化合物并储存在-80°C直至冷冻切片。将组织块在低温恒温器中升至-20°C,然后切成30μm的切片,并在冷冻切片后干燥安装到载玻片上。切片后,将载玻片在室温下放置过夜以干燥。为了协助神经元定量,我们用以下抗体和浓度对切片进行染色:兔抗 GFP(1:100;Millipore-Sigma,目录号 AB3080;RRID:AB_91337)和小鼠抗 NeuN (A60)(1:500;RRID:AB_377)和小鼠抗 NeuN (A2298772)(488:1;RRID:AB_500)。 Millipore-Sigma,目录号 MAB21206;RRID:AB_2535792)。对于二抗染色,使用以下二抗和浓度:驴抗兔 Alexa Fluor 647(1:500;Thermo Fisher Scientific,目录号 A-31571;RRID:AB_162542)和驴抗小鼠 Alexa Fluor 1(0.25 :100;Thermo Fisher Scientific,目录号 A-5.00;RRID:AB_2)。所有抗体均用补充有 1% Triton X-10,000 (PBST) 和 1306% 正常驴血清的 5× PBS 稀释。一抗孵育在室温下过夜。然后用PBST洗涤切片。二抗在室温下孵育XNUMX小时。将切片在 PBST 中洗涤三次。将切片在DAPI溶液(XNUMX:XNUMX;Invitrogen,DXNUMX)中室温孵育XNUMX分钟,然后洗涤。使用 Prolong Diamond Antifade 将切片盖玻片。
对每只动物的三个切片进行染色和成像。每个切片一式三份成像,每个感兴趣区域总共有九张图像。使用 Keyence BZ-X800 对感兴趣的组织区域进行成像,采集参数如下:GFP (1/500 s)、Cy5 (1 s)、DAPI (1/12 s)、高分辨率 Z 以 1.2μm 间距堆叠。对以下大脑分区进行成像:额叶、顶叶、颞叶、枕叶皮质、小脑、尾状核、壳核和丘脑(内侧核、腹外侧核和腹后核)。使用 ImageJ (RRID: SCR_003070) 进行半自动细胞计数方法进行定量。使用阈值和颗粒分析,我们量化了 NeuN 阳性和 DAPI 阳性细胞。使用 ImageJ 的细胞计数器,我们手动计数了 GFP 阳性细胞以及 GFP 和 NeuN 双阳性细胞。
iPSC 实验
根据制造商的方案,使用 Stemdiff 前脑分化和成熟试剂盒(分别为 StemCell # 08600 和 # 08605)分化和成熟 iPSC 衍生的神经祖细胞,从而产生神经元培养物。通过使用 Stemdiff SMADi 神经诱导试剂盒 (StemCell l#1019) 分化包皮成纤维细胞衍生的 iPSC 系:ACS-0100 (ATCC# DYS-499;RRID:CVCL_X08581),用 Stemdiff Neural Rosette Selection 进行选择,产生神经祖细胞根据其制造商的方案,使用试剂 (StemCell l#05832) 并在 Stemdiff 神经祖细胞培养基 (StemCell l#05833) 中进行扩增。神经元在重新铺板进行转导之前至少成熟 8 天。
将成熟神经元培养物每孔接种 15,000 个细胞于聚鸟氨酸和层粘连蛋白包被的黑壁 96 孔光学板中,在转导前再培养 4 天。复制孔用在 90% 成熟培养基和 10% OptiPRO SFM 中连续稀释六个数量级的病毒转导。转导后四天,用 4% PFA 固定培养物并用 1 μg ml 复染 - 1 Hoechst 33322。转导细胞的鉴定通过在 CellInsight CX60 HCS 平台上使用双通道荧光检测(Hoechst,ex386/em440;eGFP,ex485/em521)对每孔 5 个视野进行成像来确定。通过 Hoechst 检测细胞核并对每个细胞应用尺寸和接触约束的环形掩模来识别单个细胞。通过测量单个环形掩模内高于阈值水平的 eGFP 荧光来确定细胞转导。对于每个群体,将转导细胞的百分比与所施加的剂量作图。曲线拟合和 EC50 使用 Prism GraphPad (RRID: SCR_002798) 激动剂与响应(三参数)回归方法确定值。为了报告每个细胞的 eGFP 表达效率,对每孔至少 5,000 个细胞的每个环形掩模的 eGFP 点荧光强度进行平均。使用 Prism GraphPad Biphasic X 作为浓度回归方法获得曲线拟合。
统计和再现性
对于代表性图像,每个样品至少安装三个单独的切片进行成像。在单个动物的每个大脑区域内,至少获取三个不同的视野(平铺后的最小视野,2.38 mm × 2.38 mm;切片厚度,50 μm),相当于三个脑切片的九个独立视野,以确保成像样本的一致性。
报告摘要
有关研究设计的更多信息,请参阅 自然投资组合报告摘要 链接到这篇文章。
- :是
- :在哪里
- $UP
- 000
- 1
- 10
- 100
- 11
- 12
- 13
- 14
- 15%
- 16
- 17
- 180
- 2%
- 20
- 200
- 2011
- 2015
- 2016
- 2017
- 2018
- 2019
- 2020
- 2021
- 24
- 25
- 27
- 30
- 300
- 31
- 33
- 39
- 40
- 50
- 500
- 60
- 66
- 7
- 70
- 72
- 75
- 8
- 80
- 9
- 95%
- a
- 以上
- 绝对
- ACCESS
- 按照
- 根据
- 获得
- 横过
- 要积极。
- 活动
- Ad
- 加
- 添加
- 额外
- 另外
- 管理
- 管理
- 采用
- 成人
- 高级
- 不利的
- 后
- 再次
- 年龄
- AL
- Alexa的
- 算法
- 对齐的
- 所有类型
- 允许
- 沿
- 还
- 量
- 放大
- 放大
- 放大
- an
- 分析
- 锚
- 和
- 动物
- 动物
- 抗体
- 任何
- 应用的
- 应用
- 适当
- 批准
- 约
- 国家 / 地区
- 围绕
- 刊文
- 人造的
- AS
- 组装
- 组装
- 评估
- 评估
- 协助
- At
- 自动化
- 可使用
- 远离
- 骨干
- BAND
- 基于
- BD
- BE
- 成为
- before
- 开始
- 作为
- 基准
- 最佳
- 之间
- 偏见
- 捆绑
- 黑色
- 阻止
- 吹氣梢
- 血液
- 身体
- 天生的
- 都
- BP
- 大脑
- 大脑
- 简要地
- 带
- 缓冲
- 但是
- by
- 笼子
- 计算
- 计算
- 加州
- CAN
- 捕获
- 捕获
- 关心
- 货物
- 喵星人
- 细胞
- 细胞
- 细胞的
- Center
- 中央
- 链
- 陈
- 更改
- 变
- 选择
- 清除
- 点击
- 密切
- 集群
- 采集
- 柱
- 结合
- 结合
- 委员会
- 相当常见
- 补充
- 完成
- 完成
- 符合
- 执行
- 复合肥产线
- 浓度
- 流程条件
- 条件
- 确认
- CONFIRMED
- 分享链接
- 已联繫
- 常数
- 建设
- 包含
- 包含
- 控制
- 调节器
- 副本
- 核心
- 冠
- Counter
- 标准
- 文化塑造
- 曲线
- 习俗
- 量身定制
- 切
- 周期
- 周期
- 每天
- data
- 数据集
- 戴维斯
- 天
- 一年中的
- 深
- 定义
- 提升
- 交货
- 描述
- 设计
- 设计
- 期望
- 详情
- 检测
- 确定
- 决心
- 偏差
- 设备
- 设备
- 钻石
- 迭戈
- 差异
- 不同
- 稀释
- 屏 显:
- 显示
- 距离
- 不同
- 分配
- 多元化
- 多元化
- 的DNA
- 完成
- 剂量
- 加药
- 驾驶
- 干
- 为期
- ,我们将参加
- e
- Ë&T
- 每
- 此前
- 版
- 效果
- 影响
- 效率
- 高效
- 有效
- 或
- 分子
- 消除
- 嵌入式
- 英国
- 确保
- 保证
- 成熟
- 醚(ETH)
- EU
- 评估
- 所有的
- 例子
- 交换
- 扩张
- 实验
- 实验
- 表达
- 提取
- 萃取
- 家庭
- 美联储
- 女
- 部分
- 字段
- 无花果
- 数字
- 档
- 过滤
- 最后
- 终于
- 发现
- (名字)
- 适合
- 配件
- 五
- 固定
- Flash
- 流
- 流体
- 其次
- 以下
- 如下
- 食品
- 针对
- 为了产量
- 向前
- 发现
- 四
- FRAME
- 自由的
- 止
- 冻结
- 实用
- 进一步
- 天然气
- 测量
- 其他咨询
- 生成
- 产生
- 代
- 基因
- 基因组
- 基因组学
- 特定
- 图形
- 更大的
- 绿色
- 团队
- 组的
- 方针
- 半
- 处理
- 有
- 有
- 头
- 健康管理
- 相关信息
- 高
- 高分辨率
- 孔
- 匀浆
- 主持人
- HTTPS
- 人
- ICE
- ID
- 相同
- 鉴定
- 确定
- if
- ii
- 三
- 图片
- 同步成像
- 沉浸
- 浸没
- 改善
- in
- 包括
- 孵化
- 独立地
- 指数
- 表示
- 指数
- 个人
- 个别地
- 感应
- 感染
- 信息
- 输注
- 注入
- 初始
- 原来
- 代替
- 研究所
- 机构
- 文书
- 兴趣
- 接口
- 成
- 静脉
- 介绍
- 参与
- 孤立
- 杰克逊
- 试剂盒
- 实验室
- 实验室
- 实验室
- 大
- ld
- 最少
- 左
- 减
- Level
- 库
- 自学资料库
- 生活
- 极限
- Line
- 友情链接
- 链接
- 液体肥产线
- 清单
- 肝
- 本地
- 长期
- 离
- 降低
- 降低
- 制成
- 保养
- 手动
- 制图
- 面膜
- 面膜
- 主
- 匹配
- 匹配
- 材料
- 矩阵
- 衡量
- 测量
- 测量
- 媒体
- 中等
- 心理
- 心理健康
- 方法
- 方法
- 公
- 小鼠
- 显微镜
- 分钟
- 最低限度
- 分钟
- 混合
- 混合物
- ML
- 改性
- 监控
- 月
- 个月
- 更多
- 基因
- MSCI
- 亦即
- 纳米技术
- National
- 自然
- 近
- 必要
- 网络
- 网络
- 神经
- 神经元
- 神经元
- 全新
- 下页
- 美国国立卫生研究院
- 没有
- 节点
- 正常
- 数
- 目标
- 获得
- 发生
- of
- 老
- on
- 一
- 仅由
- 最佳
- 优化
- or
- 订单
- 其他名称
- 我们的
- 超过
- 过夜
- 牛津
- 氧
- 包装
- 配对
- 参数
- 部分
- 粒子
- PBS
- PCR
- 高峰
- 佩恩
- 宾夕法尼亚
- 为
- 百分比
- 执行
- 外围设备
- 相
- 管道
- 沥青
- 地方
- 计划
- 血浆
- 平台
- 柏拉图
- 柏拉图数据智能
- 柏拉图数据
- 加
- 池
- 人口
- 个人档案
- 平台精度
- 准备
- 准备
- 当下
- 呈现
- 防止
- 先前
- 小学
- 底漆
- 探测器
- 程序
- 程序
- 过程
- 处理
- 处理
- 生产
- 生成
- 产品
- 生产
- 核心产品
- 祖细胞
- 曲目
- 保护
- 蛋白质
- 蛋白质
- 协议
- 协议
- 提供
- 出版
- 泵
- 购买
- 蟒蛇
- 质量
- 量
- 量子比特
- 兔
- 凸
- 率
- 比
- 达到
- 反应
- 反应
- 阅读
- 实时的
- 收到
- 建议
- 恢复
- 地区
- 地区
- 回归
- 监管
- 相对的
- 去掉
- 去除
- 重复
- 报告
- 报道
- 报告
- 代表
- 代表
- 必须
- 研究
- 分别
- 响应
- 回复
- 限制
- 导致
- 反转
- 右
- 戒指
- 上升
- 上升
- RNA
- 健壮
- 罗氏
- Room
- 路线
- 运行
- 运行
- s
- 同
- 圣
- San Diego
- 沙
- 鳞片
- 预定
- SCI
- .
- 得分了
- 分数
- 筛查
- 脚本
- 次
- 部分
- 部分
- 看到
- 分割
- 选
- 选择
- 可选择的
- 灵敏度
- 发送
- 分开
- 序列
- 测序
- 精華
- 集
- 设置
- 几个
- 运
- 显示
- 双方
- 视力
- 信号
- 类似
- 单
- 坐
- SIX
- 尺寸
- 皮肤
- 切片
- 滑
- 幻灯片
- 慢慢地
- 小
- 钠
- 方案,
- 听起来
- 特别是
- 特异性
- Spot
- 堆
- 堆栈
- 标准
- 步
- 步骤
- 存储
- 存储
- 串
- 学习
- 随后
- 合适的
- 优于
- 磁化面
- 合成的
- 系统
- 系统的
- 产品
- 表
- 拍摄
- 窃听
- 目标
- 针对
- 瞄准
- 技术
- 专业技术
- 模板
- 十
- 终端
- 测试
- 比
- 这
- 区域
- 其
- 他们
- 然后
- 那里。
- 因此
- 博曼
- 他们
- Free Introduction
- 三
- 门槛
- 通过
- 始终
- 次
- 时
- 组织
- 至
- 了
- 合计
- 追踪
- 追踪
- 转让
- 转移
- 治疗
- 海卫一
- 两次
- 捻
- 二
- 类型
- 超级
- 下
- 独特
- 普遍
- 大学
- 美国加州大学
- 宾夕法尼亚大学
- 直到
- 使用
- 用过的
- 运用
- 利用
- 利用
- 验证
- 价值观
- 变种
- 各个
- 版本
- 与
- 通过
- 查看
- 病毒
- 病毒
- 病毒
- 体内
- 卷
- 是
- 华盛顿
- 水
- we
- 周
- 周
- 重量
- 井
- 井
- 为
- 这
- 全
- 广泛
- 中
- 世界
- X
- 年
- 产量
- 产量
- , Zen Thermosat
- 和风网