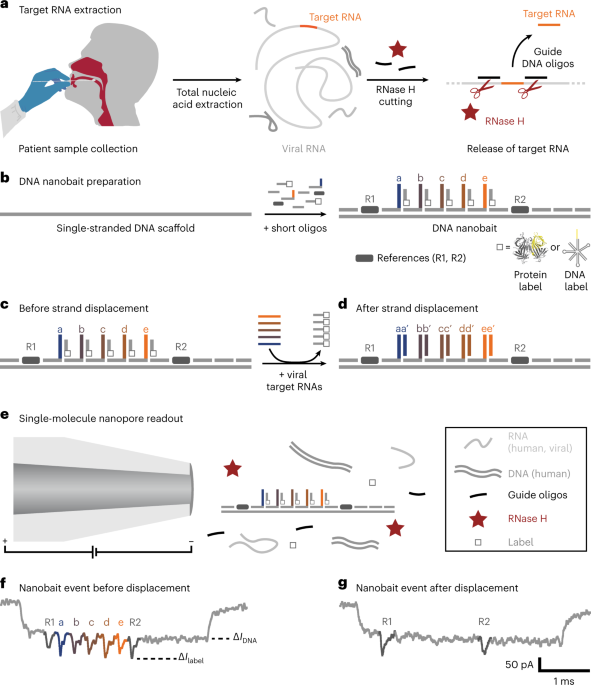
การเก็บตัวอย่างผู้ป่วย
เก็บตัวอย่างผู้ป่วยโดยการเช็ดหลังคอ (oropharyngeal swab) ของผู้ป่วยตามที่อธิบายไว้ก่อนหน้า26. ตัวอย่างถูกรวบรวมจากผู้ป่วยที่มีภาพทางคลินิกคล้าย COVID-19 และทดสอบด้วย qRT-PCR หลังจากการสกัดกรดนิวคลีอิก โดยสังเขป หลังจากการเก็บรวบรวม ไม้กวาดถูกใส่ลงในหลอดตัวอย่างที่มีฉลากซึ่งมีบัฟเฟอร์สลาย (4 M guanidine thiocyanate, 25 mM Tris–HCl, 0.5% β-mercaptoethanol และ MS2 RNA (200 ng µl-1; โรช)). หลอดถูกกวนเบา ๆ เพื่อให้แน่ใจว่ามีการกระจายบัฟเฟอร์การสลายอย่างสม่ำเสมอ ขั้นตอนด้านความปลอดภัยได้รับการอธิบายไว้ก่อนหน้านี้และดำเนินการในห้องปฏิบัติการ CL2 ที่ได้รับการรับรอง26.
การสกัดกรดนิวคลีอิก
กรดนิวคลีอิกทั้งหมดถูกสกัดโดยใช้ระบบที่มีแกนหมุนและใช้โดยการทดสอบ qRT-PCR ที่เป็นมาตรฐาน26. การควบคุมการขยายเสียงภายใน (MS2 (~6 × 104 พีเอฟยู มล-1) ต่อ 10 มล. ของบัฟเฟอร์การสลายตัว) ถูกเติมในบัฟเฟอร์การสลายตัวแบบเติม (25 ไมโครลิตรต่อ 10 มล. ของบัฟเฟอร์การสลายตัว) ตัวอย่างถูกชะในน้ำที่ปราศจากนิวคลีเอส 100 ไมโครลิตร (nfH2O; Invitrogen) และปล่อยทิ้งไว้ 1 นาทีก่อนปั่นแยกเป็นเวลา 1 นาทีที่ 21,130×g (15,000 รอบต่อนาที) ในไมโครฟิวจ์แบบตั้งโต๊ะ ตัวอย่างที่ชะออกนั้นอยู่ภายใต้ qRT-PCR โดยตรง สารสกัดกรดนิวคลีอิกที่เหลือถูกเก็บไว้ที่ −80 °C และใช้สำหรับการตรวจจับนาโนเบต-นาโนพอร์ต่อไป
qRT-PCR สำหรับ SARS-CoV-2
การตรวจจับ SARS-CoV-2 ดำเนินการตามที่อธิบายไว้ก่อนหน้านี้26. ต่อปฏิกิริยา ส่วนผสมหลักประกอบด้วย 12.5 µl ของ 2× Luna Universal Probe One-Step reaction mix, 0.5 µl ของ 20 µM Wu ไพรเมอร์ไปข้างหน้า (5′-ATGGGTTGGGGATTATCCTAAATGTGA-3′), 0.5 µl ของ 20 µM Wu ไพรเมอร์ย้อนกลับ (5′ -GCAGTTGTGGCATCTCCTGATGAG-3′), 0.3 µl ของ 10 µM MGB Probe 3 fluorescein (5′-ATGCTTAGAATTATGGCCTCAC-3′), 0.5 µl ของ 10 µM ของไพรเมอร์การควบคุมภายในไปข้างหน้าสำหรับ MS2 RNA, 0.5 µl ของ 10 µM ไพรเมอร์ย้อนกลับการควบคุมภายในสำหรับ MS2 RNA, 0.3 µl ของโพรบภายใน 10 µM (MS2 ROX), 1 µl ของ Luna WarmStart RT Enzyme Mix และ 3.9 µl ของ nfH2O. จากนั้น 20 µl ของส่วนผสมหลักถูกแบ่งส่วนลงในแต่ละหลุมของเพลต 96 หลุม และจากนั้นรวมกับ 5 µl ของสารสกัดแต่ละชนิด การควบคุมการสกัดและการขยายภายในของ MS2 ที่อยู่ภายใต้โปรโตคอลการสกัดแบบสมบูรณ์ถูกรวมไว้เป็นการควบคุมการสกัดเชิงลบในอย่างน้อยสองหลุมบนเพลต qRT-PCR เพื่อระบุการปนเปื้อนที่อาจเกิดขึ้นในกระบวนการ qRT-PCR, 5 µl nfH2O ถูกรวมเป็นตัวควบคุมเชิงลบ qRT-PCR จากนั้น พลาสมิดแม่แบบ SARS-CoV-5 ที่ขัดขวาง 2 µl ถูกรวมอยู่ในหลุมเดียวพร้อมกับการควบคุมเชิงบวก qRT-PCR หลังจากเพิ่ม 5 µl ของตัวอย่างแต่ละตัวอย่างลงในหลุมที่กำหนด จานจะถูกปิดผนึกด้วยซีลพลาสติกใส จานถูกหมุนเหวี่ยงเป็นเวลา 1 นาทีที่ 2,000×g (1,000 รอบต่อนาที) ที่อุณหภูมิ 4 °C แล้วใส่ในเครื่อง qRT-PCR (QuantStudio, Thermo Fisher Scientific) และการทำงานเป็นแบบพาราเมตริก ได้รับสัญญาณสำหรับ fluorescein (FAM) และ carboxyrhodamine (ROX) ROX ใช้เพื่อตรวจจับการควบคุม MS2 ภายใน และใช้ fluorescein เพื่อตรวจจับ SARS-CoV-2 RNA การทดสอบดำเนินการเป็นเวลา 2 นาทีที่ 25 °C, 15 นาทีที่ 50 °C (สำหรับรีเวิร์สทรานสคริปเทส), 2 นาทีที่ 90 °C ก่อน 45 รอบที่ 95 °C เป็นเวลา 3 วินาทีตามด้วย 60 °C เป็นเวลา 30 วินาที . ผลลัพธ์ถูกกำหนดโดยการยืนยันการควบคุมเชิงบวกที่ถูกต้อง (การขยายของพลาสมิด) การสกัดและการควบคุมการขยายของตัวอย่างทั้งหมด (ช่อง ROX) ไม่มีการขยายในกลุ่มควบคุมเชิงลบและค่าเฉลี่ยที่สอดคล้องกันของการควบคุม ความเป็นบวกของ SARS-CoV-2 ได้รับการยืนยันโดยการขยายสัญญาณในช่องสัญญาณฟลูออเรสซีนด้วยเส้นโค้ง sigmoidal ที่เหมาะสมโดยมีค่า CT เท่ากับ ≤36 ค่า CT ของโพรบ MS2 และ MGB 3 ได้รับการบำรุงรักษาเพื่อติดตามคุณภาพและความสามารถในการทำซ้ำของการทดสอบ44.
การตัด RNase H ที่ตั้งโปรแกรมได้สำหรับนาโนเบต
สำหรับการตรวจหารูพรุนระดับนาโน การควบคุม SARS-CoV-2 RNA, สารสกัดกรดนิวคลีอิก (ตัวอย่างผู้ป่วย) หรือ RNA ของไวรัส MS2 ถูกนำมาใช้เพิ่มเติมเพื่อตรวจหาด้วยเหยื่อนาโน ขั้นแรก เราผสมโอลิโกไกด์กับตัวอย่างและทำให้ร้อนถึง 70 °C เป็นเวลา 5 นาที RNase H (5,000 หน่วยต่อมิลลิลิตร NEB) ถูกเติม ผสมและให้ความร้อนเป็นเวลา 20 นาทีที่ 37 °C เพื่อให้เอนไซม์ตัด RNA ใน DNA: RNA ไฮบริดที่ปล่อย RNA เป้าหมายอย่างมีประสิทธิภาพ RNase H ถูกปิดใช้งานด้วยความร้อนโดยการบ่มที่ 65 °C เป็นเวลา 10 นาที คู่มือ oligos ได้รับการตรวจสอบแล้วว่าไม่สร้างโครงสร้างภายในโมเลกุล, โฮโม- หรือเฮเทอโรไดเมอร์โดยใช้ซอฟต์แวร์ NUPACK45. สำหรับการวัดที่ไม่มีเป้าหมาย จะใช้โปรโตคอลเดียวกันรวมถึงไกด์โอลิโกส การวัดการควบคุมไม่แสดงการกระจัด และด้วยเหตุนี้ เราสามารถแยกการโยงข้ามที่สำคัญออกจากโอลิโกไกด์ได้
คุณสมบัติลำดับเป้าหมายของไวรัสสำหรับนาโนเบต
ความยาวของเป้าหมาย ความยาวส่วนปลาย และเนื้อหา GC ถูกเลือกเพื่อให้แน่ใจว่ามีการผสมที่เหมาะสมที่สุด21. สำหรับการออกแบบ DNA nanobait ลำดับเป้าหมายถูกเลือกให้อยู่ในบริเวณอนุรักษ์ของจีโนมไวรัส และมีปริมาณ GC 40–60% เพื่อสร้างดูเพล็กซ์ 20 bp ที่เสถียร ความยาวส่วนปลายถูกเลือกให้มีความยาว 6 nt และมีเนื้อหา GC 40–60% เราทดสอบลำดับทั้งหมดสำหรับการโต้ตอบภายในโมเลกุลที่มีความเสถียรสูงหรือโฮโมไดเมอร์ที่ไม่พึงประสงค์โดยใช้ซอฟต์แวร์ NUPACK (เว็บแอปพลิเคชัน 2020)45. จากนั้น เราทำการตรวจสอบปฏิกิริยาข้ามระหว่างไซต์หลายแห่งที่ใช้ในการทดลองแต่ละครั้ง45.
การเตรียมดอกไม้ DNA สำหรับเหยื่อนาโน
เราได้ออกแบบดอกไม้ DNA เพื่อเป็นอีกหนึ่งฉลากสำหรับการตรวจจับ SARS-CoV-2 RNA จากตัวอย่างผู้ป่วย ดอกไม้ DNA สามดอกที่เฉพาะเจาะจงสำหรับแต่ละเป้าหมายของ SARS-CoV-2 (ทางแยกเจ็ดทาง, 7WJa, 7WJb และ 7WJc) ถูกเตรียมแยกกัน ยกตัวอย่าง 7WJc, 4 μM DNA strand J1, J2, J3 และ J4c (ตารางเสริม 1) ถูกผสมเข้าด้วยกันใน TM บัฟเฟอร์ (10 mM Tris–HCl, 10 mM MgCl2, pH 8.0) และทำให้ร้อนถึง 90 °C เป็นเวลา 5 นาที จากนั้นทำให้เย็นลงถึง 65 °C เป็นเวลา 15 นาที 45 °C เป็นเวลา 15 นาที 37 °C เป็นเวลา 20 นาที 25 °C เป็นเวลา 20 นาที และในที่สุดก็ถึง 4 ° C เป็นเวลา 20 นาที Strand J4c ถูกแทนที่ด้วย J4b เพื่อเตรียม 7WJb สำหรับ 7WJa เพื่อหลีกเลี่ยงการพับตัวเองที่ไซต์ 43 บนนาโนเบต จึงผสม J1, J2, J3 J4a และ C43 เข้าด้วยกันก่อนหลอม
การประกอบตัวเองของ DNA nanobait
DNA nanobait ถูกรวบรวมโดยการผสม M13 DNA เส้นเดี่ยวที่เป็นเส้นตรง (M13mp18, 7,249 nt, Guild Biosciences, 100 nM) กับ oligonucleotides สั้น ๆ12 (บางส่วนมีโครงสร้างอ้างอิงและจับเส้นใย) และโดยการเพิ่มเส้นใยเสริมบางส่วนที่มี 3′-biotinylated สำหรับปฏิกิริยาการกระจัดของเส้นใยที่ปลายเท้า DNA M13 ที่ทำให้เป็นเส้นตรง (ความยาว 7,228 nt) ถูกเสริมด้วยโอลิโกนิวคลีโอไทด์ ด้วยเหตุนี้จึงสร้างนาโนเบตสองเส้นที่มีเกลียวสองขั้วซึ่งมีดีออกซีไทมิดีนสี่ขั้วสองขั้วที่ป้องกันการมัลติเมอไรเซชัน12. ส่วนผสมประกอบด้วย DNA M20 เชิงเส้น 13 nM, 60 nM ของ oligonucleotides (มากกว่าสามเท่าของ DNA M13), 3′-biotinylated strands ในความเข้มข้น 180 nM, 10 mM MgCl2 และ 1× TE (10 มิลลิโมลาร์ ทริส–HCl, 1 มิลลิโมลาร์ EDTA, pH 8.0) มันถูกผสมโดยการปิเปตและหมุนก่อนที่จะให้ความร้อนถึง 70 °C เป็นเวลา 30 วินาที และทำให้เย็นลงกว่า 45 นาทีจนถึงอุณหภูมิแวดล้อม โอลิโกนิวคลีโอไทด์ส่วนเกินถูกกำจัดออกโดยใช้ตัวกรองแบบแรงเหวี่ยง Amicon Ultra 0.5 มล. พร้อมการตัด 100 kDa พร้อมบัฟเฟอร์การล้าง (10.0 mM Tris–HCl pH 8.0, 0.5 mM MgCl2). หากใช้ดอกไม้ DNA เป็นฉลาก สายเสริมบางส่วนที่มีมันจะถูกบ่มใน 10 mM MgCl2 เป็นเวลา 2 ชั่วโมงที่อุณหภูมิแวดล้อม และต่อมา การกรอง Amicon ถูกดำเนินการตามที่อธิบายไว้ข้างต้น ความไม่สมมาตรของการออกแบบเหยื่อนาโนทำให้สามารถระบุตำแหน่งที่มีผลผูกพันได้อย่างชัดเจน นาโนเหยื่อถูกเก็บไว้จนกว่าจะใช้สำหรับการทดลองเพิ่มเติมภายใต้ 4–10 °C ใน 0.5 mM MgCl2, 10.0 มิลลิโมลาร์ ทริส–HCl, pH 8.0 การออกแบบเหยื่อนาโนได้รับการตรวจสอบโดยการอ่านค่านาโนพอร์ก่อนการวัดแต่ละครั้ง
การอ่านค่า Nanopore ของ DNA nanobait
เหยื่อนาโนผสมกับตัวอย่าง (สารสกัดจากกรดนิวคลีอิกหรือเป้าหมายของไวรัสที่บริสุทธิ์เกิน 10 เท่า) ใน XNUMX mM MgCl2 และ 100 mM NaCl ของผสม (5 ไมโครลิตร) ถูกบ่มที่อุณหภูมิห้อง (~10 นาที) จนกระทั่งเตรียมพร้อมสำหรับการวัดนาโนพอร์ ความแตกต่างในองค์ประกอบของลำดับเป้าหมายและลักษณะทางกายภาพอาจนำไปสู่ความแปรปรวนในการผสมข้ามพันธุ์ และด้วยเหตุนี้ประสิทธิภาพการแทนที่ของไซต์ตรวจจับ21. เราใช้ htRNA (100 ng μl-1; Invitrogen) เป็นพื้นหลังเมื่อระบุ เพื่อแสดงว่าไม่มีสัญญาณที่ไม่เฉพาะเจาะจงที่เหนี่ยวนำโดย RNA ดั้งเดิมของมนุษย์ สำหรับการวัดขนาดนาโนพอร์ ตัวอย่างถูกเจือจางเป็น <0.5 nM nanobait (สำหรับเป้าหมายไวรัสที่บริสุทธิ์) หรือ 4.7 ไมโครลิตรของตัวอย่างผู้ป่วย RNase-H-cut ผสมกับ 0.3 μlของ monovalent streptavidin (SAe1D3)18 (1 ไมโครโมลาร์), LiCl 5 ไมโครลิตร (4.0 โมลาร์) และ LiCl 5.0 ไมโครลิตร (8.0 โมลาร์) เราได้ประดิษฐ์รูพรุนขนาดนาโน 14 ± 3 นาโนเมตร (ค่าเฉลี่ย ± ส่วนเบี่ยงเบนมาตรฐาน)12 ใช้หลอดแก้วควอทซ์ที่มีเส้นผ่านศูนย์กลางภายนอก 0.5 มม. และเส้นผ่านศูนย์กลางภายใน 0.2 มม. (เครื่องมือซัตเตอร์) โดยใช้ตัวดึงที่ใช้เลเซอร์ช่วย P-2000 (เครื่องมือซัตเตอร์) ปิเปตของผสมถูกปิเปตในชิปนาโนโพลิไดเมทิลไซลอกเซน และการวัดทั้งหมดดำเนินการที่แรงดันคงที่ 600 มิลลิโวลต์ รายละเอียดการวัดขนาดนาโนพอร์แสดงในตารางเสริม 30.
การวิเคราะห์ข้อมูล nanopore ตามเวลาจริง
การวิเคราะห์ข้อมูล Nanopore มีคำอธิบายโดยละเอียดในส่วนเสริม 14. โดยสังเขป เหตุการณ์ของนาโนเบตถูกกรองออกจากร่องรอยของกระแสไอออนิกดิบ จากนั้นจึงกำหนดขอบเขตการตรวจจับ และข้อมูลของการมีอยู่ของสไปค์ที่ไซต์เฉพาะแต่ละแห่งถูกดึงออกมา ประสิทธิภาพการกระจัดที่วางแผนไว้คำนวณเป็นประสิทธิภาพการกระจัดสำหรับการวัดที่ลบออกไปยังการควบคุมที่ไม่มีเป้าหมายสำหรับแต่ละไซต์ (50 เหตุการณ์นาโนเบตสำหรับแต่ละการบันทึกของนาโนพอร์สามรายการ) เว้นแต่จะระบุไว้เป็นอย่างอื่น:
$$begin{array}{l}{mathrm{Displacement}},{mathrm{efficiency}} =frac{1}{3}mathop {sum}limits_{n = 1}^3 เหลือ{ {1 -frac{1 }{{50}}mathop {sum}limits_{n = 1}^{50} {left[ {fleft( n right) = left( {frac{{1,,mathrm{peak}}}{{0,, {mathrm{no}},{mathrm{peak}}}}} right)} right]_{{{{mathrm{target}}}}}} } right}\ – frac{1}{3}mathop {ผลรวม }limits_{n = 1}^3 {left{ {1 – frac{1}{{50}}mathop {sum }limits_{n = 1}^{50} left[ {fleft( n right) = left( { frac{{1,,mathrm{peak}}}{{0,,mathrm{no}},{mathrm{peak}}}} right)} right]_{{{mathrm{no}}}},{ {{mathrm{target}}}}}} right}} end{array}.$$
เราตรวจสอบว่า QuipuNet เครือข่ายประสาท convolutional27 มีความสามารถในการวิเคราะห์แบบเรียลไทม์ของข้อมูลนาโนพอร์ตามขั้นตอนที่อธิบายไว้ ก่อนหน้านี้ เราแสดงให้เห็นว่าด้วยเหตุการณ์ประมาณ 99 เหตุการณ์ เรามีความมั่นใจถึง XNUMX% ในการตรวจพบโครงสร้าง DNA ที่เราออกแบบในเชิงบวก46.
การถ่ายภาพ AFM
การถ่ายภาพ AFM (Nanosurf Mobile S) ของเหยื่อนาโนดำเนินการในอากาศในโหมดไม่สัมผัส โครงสร้างนาโนเบตถูกเจือจางเป็น 1 ng μl-1 ใน 1 mM MgCl2 และเติม 10 ไมโครลิตรลงในไมกาที่ตัดใหม่ บ่มเป็นเวลา 1 นาที ล้างด้วยน้ำ Milli-Q ที่กรองแล้วเป่าให้แห้งด้วยไนโตรเจน ก่อนการสแกน แผ่นไมกาถูกติดไว้ที่แท่นวางตัวอย่าง AFM โดยใช้เทปกาวสองหน้า การสร้างภาพและการวิเคราะห์ภาพดำเนินการโดยใช้ Gwyddion (เวอร์ชัน 2.60)
การวิเคราะห์ทางสถิติ
สำหรับการวัดทั้งหมด มีการคำนวณช่วงความเชื่อมั่น 99.9% สำหรับประสิทธิภาพการเคลื่อนที่ นัยสำคัญทางสถิติระหว่างสองไซต์ที่ไม่มีและมีเป้าหมายได้รับการทดสอบโดยใช้นักเรียนสองด้าน t-ทดสอบ.
สรุปการรายงาน
ข้อมูลเพิ่มเติมเกี่ยวกับการออกแบบการวิจัยมีอยู่ใน สรุปการรายงานผลงาน Nature เชื่อมโยงกับบทความนี้
- เนื้อหาที่ขับเคลื่อนด้วย SEO และการเผยแพร่ประชาสัมพันธ์ รับการขยายวันนี้
- เพลโตบล็อคเชน Web3 Metaverse ข่าวกรอง ขยายความรู้. เข้าถึงได้ที่นี่.
- ที่มา: https://www.nature.com/articles/s41565-022-01287-x
- 000
- 1
- 10
- 100
- 11
- 110
- 2014
- 2016
- 2017
- 2018
- 2020
- 2021
- 7
- 70
- 9
- a
- ข้างบน
- ไม่อยู่
- นักวิชาการ
- ที่ได้มา
- ที่เพิ่ม
- หลังจาก
- AIR
- ทั้งหมด
- ช่วยให้
- ล้อมรอบ
- การวิเคราะห์
- สมอ
- และ
- อื่น
- การใช้งาน
- เข้าใกล้
- เหมาะสม
- รอบ
- บทความ
- ลอม
- ใช้ได้
- กลับ
- พื้นหลัง
- ก่อน
- ระฆัง
- ระหว่าง
- ผูกพัน
- ระเบิด
- BP
- สั้น
- กันชน
- คำนวณ
- สามารถ
- จับ
- พกพา
- มีมาตรฐาน
- ช่อง
- ลักษณะ
- ตรวจสอบ
- เฉิน
- ชิป
- ชัดเจน
- คลินิก
- ชุด
- รวม
- ประกอบ
- สมาธิ
- ความมั่นใจ
- ยืนยัน
- คงเส้นคงวา
- คงที่
- เนื้อหา
- ควบคุม
- การควบคุม
- เครือข่ายประสาทเทียม
- การสร้าง
- ปัจจุบัน
- เส้นโค้ง
- ตัด
- ตัด
- รอบ
- ข้อมูล
- การวิเคราะห์ข้อมูล
- แสดงให้เห็นถึง
- อธิบาย
- ออกแบบ
- ได้รับการออกแบบ
- การออกแบบ
- รายละเอียด
- รายละเอียด
- การตรวจพบ
- กำหนด
- แน่นอน
- การเบี่ยงเบน
- ความแตกต่าง
- ดิจิทัล
- โดยตรง
- การกระจาย
- ดีเอ็นเอ
- ลง
- แต่ละ
- มีประสิทธิภาพ
- ประสิทธิภาพ
- อย่างมีประสิทธิภาพ
- ชั้นเยี่ยม
- ทำให้มั่นใจ
- ความผิดพลาด
- อีเธอร์ (ETH)
- แม้
- เหตุการณ์
- ตัวอย่าง
- อธิบาย
- สารสกัด
- การสกัด
- สารสกัดจาก
- ฟิลเตอร์
- ในที่สุด
- ชื่อจริง
- ดอกไม้
- ตาม
- ดังต่อไปนี้
- ฟอร์ม
- ข้างหน้า
- ราคาเริ่มต้นที่
- เต็ม
- ต่อไป
- จีโนม
- กระจก
- ให้คำแนะนำ
- อย่างสูง
- HTTPS
- เป็นมนุษย์
- เป็นลูกผสม
- ประจำตัว
- ภาพ
- การถ่ายภาพ
- การดำเนินงาน
- in
- รวม
- รวมทั้ง
- ฟักไข่
- ฟักไข่
- ข้อมูล
- ตราสาร
- ปฏิสัมพันธ์
- ภายใน
- อิออน
- IT
- KDA
- ฉลาก
- นำ
- ความยาว
- LINK
- ที่เชื่อมโยง
- นาน
- Luna
- เครื่อง
- เจ้านาย
- วัสดุ
- วัด
- ไมกา
- อาจ
- ขั้นต่ำ
- ผสม
- การผสม
- สารผสม
- ML
- โทรศัพท์มือถือ
- โหมด
- MOL
- หลาย
- นาโน
- นาโนพอร์
- พื้นเมือง
- ธรรมชาติ
- เชิงลบ
- เครือข่าย
- ประสาท
- เครือข่ายประสาท
- เปิด
- ดีที่สุด
- มิฉะนั้น
- การจับคู่
- ผู้ป่วย
- ผู้ป่วย
- การปฏิบัติ
- กายภาพ
- ภาพ
- พลาสติก
- เพลโต
- เพลโตดาต้าอินเทลลิเจนซ์
- เพลโตดาต้า
- ผลงาน
- บวก
- positivity
- ที่มีศักยภาพ
- เตรียมการ
- เตรียม
- การมี
- ป้องกัน
- ก่อนหน้านี้
- เชื้อปะทุ
- การสอบสวน
- กระบวนการ
- คุณสมบัติ
- โปรตีน
- โปรโตคอล
- คุณภาพ
- ดิบ
- มาถึง
- ปฏิกิริยา
- เรียลไทม์
- ภูมิภาค
- ภูมิภาค
- สัมพันธ์
- ที่เหลืออยู่
- ลบออก
- การรายงาน
- การวิจัย
- ผลสอบ
- ย้อนกลับ
- อาร์เอ็นเอ
- หิน
- ห้อง
- rt
- กฎระเบียบ
- วิ่ง
- ความปลอดภัย
- เดียวกัน
- โรคซาร์ส COV-2
- การสแกน
- Section
- เลือก
- ลำดับ
- สั้น
- โชว์
- แสดง
- สัญญาณ
- ความสำคัญ
- พร้อมกัน
- เดียว
- เว็บไซต์
- สถานที่ทำวิจัย
- เล็ก
- ซอฟต์แวร์
- บาง
- โดยเฉพาะ
- ปั่น
- มั่นคง
- ระยะ
- ยืน
- มาตรฐาน
- ระบุ
- ทางสถิติ
- ขั้นตอน
- เก็บไว้
- เส้น
- ต่อจากนั้น
- เป็นกอบเป็นกำ
- ระบบ
- ตาราง
- การ
- เป้า
- เป้าหมาย
- เทมเพลต
- สิบ
- พื้นที่
- ดังนั้น
- สาม
- ครั้ง
- TM
- ไปยัง
- ร่วมกัน
- รวม
- ลู่
- รุนแรง
- ภายใต้
- หน่วย
- สากล
- การตรวจสอบ
- ความคุ้มค่า
- ความคุ้มค่า
- การตรวจสอบแล้ว
- รุ่น
- ผ่านทาง
- ไวรัส
- การสร้างภาพ
- แรงดันไฟฟ้า
- W
- น้ำดื่ม
- เว็บ
- โปรแกรมประยุกต์บนเว็บ
- เวลส์
- ที่
- ไม่มี
- wu
- ลมทะเล