Síntese de nanobots
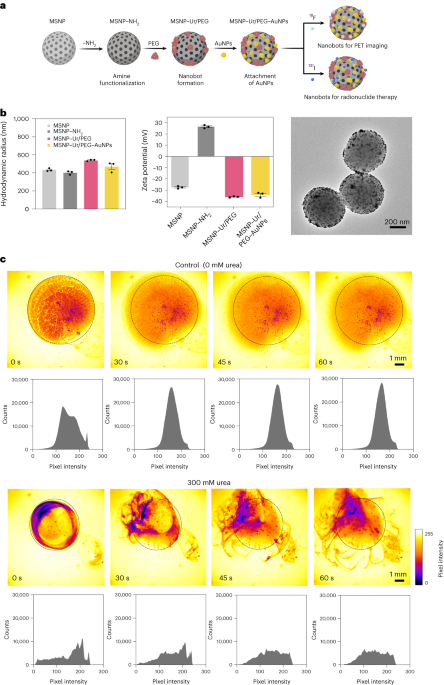
Nanobots foram preparados conforme descrito anteriormente33. Resumidamente, as MSNPs foram sintetizadas usando um método de Stöber modificado41, reagindo trietanolamina (35 g), água ultrapura (20 ml) e brometo de hexadeciltrimetilamônio (CTAB; 570 mg) a 95 °C por 30 min enquanto se agita. Ortossilicato de tetraetila (1.5 ml) foi subsequentemente adicionado gota a gota; a mistura foi deixada reagir por 2 horas a 95°C e as MSNPs resultantes coletadas por centrifugação e lavadas em etanol (três vezes, 2,500g, 5 min). Para remover o modelo CTAB, os MSNPs foram colocados sob refluxo em metanol ácido (1.8 ml de HCl, 30 ml de metanol) por 24 horas. Em seguida, as MSNPs foram coletadas por centrifugação e lavadas três vezes em etanol (2,500g, 5 min) antes de incorporar a modificação de amina adicionando APTES (6 µl por mg de MSNP) a MSNPs (1 mg ml-1) em solução etanólica a 70% a 70 °C, agitando vigorosamente por 1 h. MSNPs-NH2 foram coletados e lavados três vezes em etanol e três vezes em água por centrifugação (três vezes, 1,150g, 5 min). MSNPs-NH2 foram ressuspensos em PBS na concentração de 1 mg ml-1 e volume total de 900 µl, e ativado com glutaraldeído (100 µl) por 2.5 h em temperatura ambiente. Os MSNPs-NH ativados2 foram coletados e lavados em PBS três vezes por centrifugação (1,150g, 5 min), ressuspenso em solução de urease (3 mg ml-1) e PEG heterobifuncional (1 μg PEG por mg de 5 kDa HS-MSNPs-NH2) em PBS e reagiu por 24 horas à temperatura ambiente. Os nanobots resultantes foram então coletados e lavados três vezes em PBS por centrifugação (1,150g, 5 min) antes de ressuspendê-los em uma dispersão de AuNPs, preparada conforme descrito anteriormente51, deixando-os reagir por 10 minutos e lavando completamente por centrifugação (três vezes, 1,150g, 5 minutos).
Distribuição de tamanho hidrodinâmico e carga superficial dos MSNPs, MSNPs-NH2, nanobots e nanobots decorados com AuNP foram determinados usando um sistema de dispersão de luz dinâmica Wyatt Mobius e um Malvern Zetasizer, respectivamente. Em todos os casos, a concentração foi de 20 µg ml-1 e tempo de aquisição 5 s, utilizando três execuções por experimento. Foram realizadas três medições por tipo de partícula.
Síntese de MSNPs FITC
Uma mistura de FITC (2 mg), etanol (5 ml) e APTES (400 µl) foi preparada e agitada durante 30 min. Em seguida, foi seguido o protocolo descrito anteriormente para síntese de MSNP, exceto que adicionamos ortossilicato de tetraetila (1.25 ml) gota a gota em combinação com a mistura FITC-APTES (250 µl). As etapas de funcionalização para obter nanobots marcados com FITC foram as mencionadas acima.
Síntese de AuNPs
AuNPs foram sintetizados usando um método relatado33. Resumidamente, todos os materiais foram limpos com água régia recém-preparada, enxaguados abundantemente com água e secos ao ar. Posteriormente, um AuCl 1 mM4 A solução foi aquecida até o ponto de ebulição enquanto se agitava em um balão de fundo redondo integrado a um sistema de refluxo. Em seguida, foram adicionados 10 ml de solução de citrato de sódio (30.8 mM) e a solução foi fervida por 20 min, resultando em uma cor vermelha. A solução foi então deixada arrefecer até à temperatura ambiente enquanto se agitava durante 1 h. Os AuNPs resultantes foram armazenados no escuro e a caracterização foi realizada utilizando microscopia eletrônica de transmissão.
Atividade enzimática
Atividade enzimática de nanobots, 18F-nanobots e 131Os I-nanobots foram medidos usando vermelho de fenol. Para fazer isso, 2 µl de nanobots (1 mg ml-1) foram adicionados a uma placa de 96 poços e misturados com 200 µl de diferentes soluções de ureia (0, 50, 100, 200 mM) em vermelho de fenol 1.1 mM. A absorvância a 560 nm foi medida ao longo do tempo a 37 °C.
Dinâmica de movimento de nanobots através de microscopia óptica
Vídeos ópticos de nanorrobôs foram adquiridos usando um microscópio Leica Thunder, acoplado a uma câmera CCD de alta velocidade Hamamatsu e uma objetiva ×1.25. Para isso, os nanorrobôs foram centrifugados e ressuspensos em 50 µl de PBS (concentração final de 20 mg ml-1). Em seguida, uma placa de Petri foi preenchida com 3 ml de PBS ou uma solução de uréia 300 mM (em PBS) e observada ao microscópio. Uma gota de 5 µl com nanobots (20 mg ml-1) foi então adicionado à placa de Petri cheia de líquido e os vídeos foram gravados a 25 quadros por segundo. As distribuições de intensidade de pixel de vídeo em ROIs foram analisadas em intervalos de 15 segundos usando o software ImageJ.
Radiomarcação de nanobots com [18F]F-PyTFP
Síntese de [18F]F-PyTFP
[18F] F-PyTFP foi sintetizado em um módulo Neptis xSeed (Optimized Radiochemical Applications), seguindo um método relatado anteriormente33.
Síntese de 18Nanorrobôs marcados com F
Nanobots foram rotulados com [18F]F-PyTFP, com base em procedimento previamente estabelecido com pequenas modificações33. Em resumo, 200 µl de solução nanobot (1 mg ml-1) foi centrifugado (10 min, 13,853g), ressuspensos em 10 µl de PBS (1 mM, pH 8) e incubados com 4 µl de [18F]F-PyTFP em acetonitrila (cerca de 37 MBq) por 35 min à temperatura ambiente. Após a incubação, a mistura reacional foi diluída com água (200 µl) e purificada por centrifugação (5 min, 13,853g). O sedimento resultante foi então enxaguado três vezes com água antes de ser medido num calibrador de dose (CPCRC-25R, Capintec). O rendimento radioquímico foi calculado como a razão entre a quantidade de radioatividade presente nos nanobots após a lavagem e a quantidade inicial de radioatividade. A pureza radioquímica após purificação foi ≥99%, conforme determinado por cromatografia em camada fina de rádio (rádio-TLC) usando papel de cromatografia iTLC-SG (Agilent Technologies) e diclorometano e metanol (2:1) como fases estacionária e móvel, respectivamente. As placas de TLC foram analisadas utilizando um leitor de TLC (MiniGITA, Raytest).
Estabilidade de 18F-nanobots
A estabilidade de 18Os nanorrobôs marcados com F foram determinados usando os seguintes meios: (1) uréia 300 mM, (2) água e (3) urina de animais portadores de tumor. 18Nanobots marcados com F (10 µl) foram incubados com a solução correspondente (100 µl) por 1 h em temperatura ambiente. Em seguida, os nanobots e o sobrenadante foram separados por centrifugação e coletados, e a radioatividade medida em um calibrador de dose (CPCRC-25R).
Radiomarcação de nanobots com 131I
A radioiodação de nanobots de urease foi realizada incubando nanobots com injetáveis [131Solução I]NaI (925 MBq ml-1; GE Saúde). Em resumo, 400 µl de solução nanobot de urease (1 mg ml-1) foi centrifugado (13,853g, 5 min), ressuspenso em 100 µl de PBS (10 mM, pH 7.4) e incubado com 25 µl ou 185 µl de injetável [131I]NaI (cerca de 42.55 ou 277.5 MBq, respectivamente) por 30 min, dependendo da atividade final desejada. Após a incubação, a mistura reacional foi purificada por centrifugação (13,853g, 5 min). O precipitado resultante foi lavado três vezes com água (100 µl). A radioatividade do sobrenadante e do precipitado foi determinada utilizando um calibrador de dose (CPCRC-25R), e ambas as frações foram analisadas por radio-TLC, assim como para 18F-nanobots.
Desenvolvimento de modelo animal
Os ratos foram mantidos e manuseados de acordo com a Diretiva do Conselho Europeu 2010/63/UE e diretrizes internas. Todos os procedimentos experimentais foram aprovados pelo comitê de ética do CIC biomaGUNE e pelas autoridades locais (Diputación Foral de Guipuzcoa, PRO-AE-SS-276). A análise de imagens (PET e RM) foi cegada em relação à distribuição dos grupos dos animais.
O modelo murino ortotópico de câncer de bexiga foi gerado por administração intravesical de células MB49 (linha celular de carcinoma murino de bexiga) a camundongos fêmeas C57BL/6JRj (8 semanas de idade, Janvier). Para experimentos destinados a determinar o acúmulo de tumor (quatro grupos; detalhes abaixo), seis animais foram inoculados por grupo, conforme determinado por análise de precisão, com as seguintes suposições: precisão necessária, 20%; dp esperado, ± 20%; confiança, 95%; perda de animais, 20%. Para experimentos de eficácia terapêutica (seis grupos; detalhes abaixo), dez animais foram incluídos por grupo, conforme calculado usando um Student unilateral t-teste, diferença entre duas médias independentes, com os seguintes pressupostos: hipótese nula, o tratamento não afeta o crescimento do tumor; α, 0.05; 1 − β, 0.95; dp, ± 50%; diferenças esperadas entre grupos, 50%; perda de animais, 20%. Como o experimento foi conduzido em dois lotes por questões operacionais, um grupo controle foi incluído em ambos os lotes (Tabela 2), e então todos os animais foram agrupados. Para o estabelecimento do tumor, os camundongos foram anestesiados por inalação de 3% de isoflurano em O puro2 e mantido por 1.0–1.5% de isoflurano em 100% de O2. Em seguida, a bexiga foi esvaziada e lesões químicas induzidas no urotélio pela instilação intravesical de 50 µl de poli-l-lisina (Sigma-Aldrich) através de um cateter de calibre 24 por 15 min. Posteriormente, a bexiga foi esvaziada novamente e células MB49 (105 células) em DMEM com alto teor de glicose (100 µl) foram instiladas por 1 hora antes de remover o cateter e esvaziar a bexiga por meio de massagem abdominal. Ao longo dos experimentos, os ratos foram monitorados e pesados para monitoramento de saúde e bem-estar. Um endpoint humano foi aplicado se a perda de peso excedesse 20% ou com base em sintomas clínicos, a critério do veterinário responsável.
Rastreamento do tamanho do tumor
Os estudos de ressonância magnética foram realizados 7 e 14 dias após a indução do tumor, usando um scanner 7 T Bruker BioSpec USR 70/30 (Bruker BioSpin) equipado com uma inserção de gradiente BGA-12S de 440 mT m-1 e um ressonador QSN 112/086 (T12053V3) para radiofrequência14 transmissão e uma bobina de superfície cerebral de rato (T11205V3) para recepção de RF (ambas operando a 300 MHz). Os animais foram anestesiados com isoflurano (4% para indução e 1.5% para manutenção em ambiente com 50% de O2/50%N2 mistura) e colocado em um suporte compatível com RM. A temperatura corporal e a frequência respiratória foram monitoradas continuamente usando um dispositivo de monitoramento compatível com MR (modelo 1030 SA, Small Animal Instruments), conectado a um sistema de aquecedor de ar para pequenos roedores para manter a temperatura corporal. Após a aquisição de imagens de referência, uma sequência de imagem ponderada por difusão baseada em spin-eco foi usada para visualizar tumores, usando os seguintes parâmetros: tempo de eco (TE) = 22.3 ms, tempo de repetição (TR) = 2,500 ms, n = 2 médias, uma imagem A0 (imagem basal com b = 0smm-2) e uma imagem DW adquirida usando gradientes de difusão na direção (1, 0, 0) com duração de gradiente δ = 4.5 ms e uma separação de gradiente Δ = 10.6 ms, dando b = 650smm-2, um 16 × 16 mm2 campo de visão, tamanho da matriz de imagem de 160 × 160 pontos, 20 fatias consecutivas de 0.5 mm de espessura (sem lacuna, adquirida em modo intercalado) e largura de banda de 192.9 Hz por pixel. Para visualizar os tumores, as imagens foram pós-processadas com o software ImageJ, dividindo as imagens adquiridas com gradiente de difusão (b = 650smm-2) por aqueles adquiridos sem (b = 0smm-2) e aplicando um filtro gaussiano 3D (σx = σy = σz = 0.7) ao resultado. Os tumores foram delineados manualmente para determinar seu volume.
Biodistribuição in vivo
No dia 15 após a indução do tumor, os ratos foram randomizados em quatro grupos para obter distribuições médias homogêneas do volume tumoral entre os grupos. Os exames PET-CT (scanners MOLECUBES β e X-CUBE) foram adquiridos 3 h após a administração intravesical de 100 µl de 18F-BSA (grupos 1 e 2) ou 18Nanorrobôs de F-urease (grupos 3 e 4) na concentração de 200 µg ml-1, usando água (grupos 1 e 3) ou 300 mM de uréia em água (grupos 2 e 4) como veículo (Tabela 1). Para aquisição das imagens, os animais foram induzidos com anestesia (isoflurano 5% em oxigênio puro) e colocados em posição supina antes de massagear a região abdominal para evacuação da bexiga. Imediatamente depois, o correspondente 18Nanorrobôs marcados com F (18F-BSA/18F-urease em água/ureia) foram instilados na bexiga através de um cateter de calibre 24 e incubados por 1 hora, antes de remover o cateter, esvaziar a bexiga e deixar os ratos se recuperarem da anestesia. No t = 3 h após a administração, os animais foram anestesiados novamente e 10 min de imagens PET estáticas de corpo inteiro foram adquiridas, seguidas de tomografias computadorizadas. As imagens PET foram reconstruídas usando o algoritmo de reconstrução de maximização de expectativa de subconjunto ordenado em 3D com correções aleatórias, de dispersão e de atenuação. Imagens PET-CT do mesmo camundongo foram co-registradas e analisadas utilizando a ferramenta de processamento de imagens PMOD. Gráficos de concentração de radioatividade versus tempo foram obtidos criando um volume de interesse na região superior da bexiga usando uma ferramenta de contorno 3D e medindo a atividade (decaimento corrigido) em quilobecquerels por órgão. Os resultados foram corrigidos aplicando um fator de calibração e depois normalizados pelo volume do tumor derivado da ressonância magnética.
Estudos ex vivo
Análises histopatológicas
Depois de concluir todas as imagens, as bexigas selecionadas (n = 3 por grupo) de animais saudáveis e portadores de tumor foram removidos em condições assépticas e imediatamente fixados em formaldeído a 4%. Em seguida, as bexigas foram embebidas em parafina antes de serem retiradas seções de 2–3 μm para coloração com hematoxilina-eosina. Imagens representativas foram obtidas de todas as condições para exame histopatológico.
Análise ICP-MS
As medições foram realizadas em um Thermo iCAP Q ICP-MS (Thermo Fisher Scientific) acoplado a um amostrador automático ASX-560 (CETAC Tech). Depois de completar todas as imagens, os animais foram mortos e as bexigas selecionadas (n = 2 por grupo; quatro grupos) coletados e digeridos em 1 ml de HNO3:HCl (mistura 4:1). A dispersão foi fervida até que os órgãos estivessem completamente dissolvidos. Em seguida, a solução foi resfriada à temperatura ambiente e analisada por ICP-MS para determinar a concentração de Au em cada amostra, transformando os resultados em porcentagens de dose injetada por grama de tecido (%ID g-1).
Imuno-histoquímica e microscopia confocal
Para análises imuno-histoquímicas, os animais portadores de tumor receberam nanorrobôs marcados com FITC em água ou uréia 300 mM (n = 4 por grupo), conforme descrito acima, para estudos PET-CT. Além disso, animais portadores de tumores sem nanobots serviram como grupo de controle (n = 2). Em todos os casos, as bexigas foram coletadas, congeladas e cortadas em seções de 10 µm que foram imediatamente fixadas em formaldeído a 10% por 15 min, lavadas com PBS 10 mM e depois incubadas em NH 50 mM.4Cl em PBS por 5 minutos antes de enxaguar novamente com PBS. A permeabilização foi realizada com metanol:acetona (1:1) por 5 minutos em temperatura ambiente e Triton 0.1% em PBS por 5 minutos. Após a lavagem com PBS, as amostras foram saturadas com uma solução de 5% de BSA – 0.5% de Tween em PBS por 15 minutos em temperatura ambiente e incubadas por 1 hora em temperatura ambiente com anti-FITC de camundongo (1:100, Abcam) em 5% de BSA. –0.5% entre. As seções foram lavadas três vezes com PBS 10 mM por 5 min e incubadas por 30 min em temperatura ambiente com anticorpo secundário Alex Fluor 647 IgG anti-camundongo de burro (Molecular Probes, Life Technologies, 1: 1,000) em 5% BSA – 0.5% Tween em PBS, lavado novamente em PBS (3 × 5 min) e montado com um kit antifade ProLong com 4,6-diamidino-2-fenilindole (DAPI; Molecular Probes, Life Technologies). As imagens foram adquiridas com um microscópio confocal Leica STELLARIS 5 (UPV/EHU Scientific Park) com configurações idênticas para todas as seções: ampliação de × 10 com imagem de ladrilho e costura (normalmente campo de visão 4 × 5). A linha do laser e as janelas de detecção foram 405 nm e 440–503 nm para DAPI, 489 nm e 494–602 nm para o laser branco FITC e 653 nm e 660–836 nm para o laser branco Alexa647.
Limpeza óptica
Após perfusão com paraformaldeído a 4% e PBS, as amostras de bexiga foram removidas e posteriormente fixadas em paraformaldeído a 4% durante a noite a 4 °C, depois incorporadas em uma seringa de 5 ml com agarose de baixo ponto de fusão a 0.8% para formar um bloco cilíndrico e permitir fácil montagem na cubeta de quartzo. Todo o bloco foi progressivamente desidratado com metanol:H2O a 4 °C (30%:70% por 1 h, 50%:50% por 1 h, 70%:30% por 1 h, 100%:0% por 1 h, depois 100% de metanol durante a noite e novamente por 4 h) e finalmente imerso em álcool benzílico-benzoato de benzila (BABB) como solução de correspondência de índice de refração para geração de imagens. Para comparações in vitro de nanobots FITC verdes com partículas vermelhas comerciais, usamos nanopartículas de sílica fluorescentes vermelhas DiagNano (Creative Diagnostics), com 1 µm de diâmetro, resistentes à compensação BABB.
Autofluorescência e imagem sLS polarizada
A imagem light-sheet foi realizada no MacroSPIM, um sistema personalizado para imagens limpas de órgãos inteiros desenvolvido no IRB Barcelona44,45. Em resumo, as amostras são incorporadas em um bloco de agarose, limpas junto com a amostra e fotografadas dentro de uma cubeta de quartzo. A imagem de autofluorescência utilizou lasers de 488, 561 ou 638 nm, fornecendo iluminação através de uma lente cilíndrica dupla acromática de 50 mm (ACY254-050-A, Thorlabs). Para reduzir artefatos de listras, a folha de luz é articulada com um scanner ressonante SC-10 (EOPC) ao longo de um telescópio 4f com lentes duplas acromáticas G322288322 100mm (QI Optic Photonics). A autofluorescência tecidual é coletada através de filtros de fluorescência de banda ou passagem longa e gravada com uma câmera ORCA Flash v2 (Hamamatsu Photonics). A imagem foi realizada em × 9.6 com zoom × 8, lente × 2 e lente de tubo × 0.6. A folha de luz foi achatada em todo o campo de visão, produzindo 5–6 µm de resolução axial. A imagem 3D foi feita em passos de 2.5 µm. A imagem da bexiga inteira foi realizada em 2 × 3 ou 3 × 4 XY telhas, dependendo do tamanho do órgão.
A imagem sLS foi obtida removendo o filtro de fluorescência ou usando qualquer filtro que transmita o laser. A rotação da folha de luz reduziu o ruído de manchas do laser, resultando na média temporal da coerência do laser, conforme mostrado anteriormente52. A orientação da polarização linear da folha de luz na iluminação foi controlada girando uma placa de meia onda (AHWP05M-600, Thorlabs) antes do scanner pivô. O sinal detectado foi selecionado na polarização usando um polarizador linear rotativo (LPVISC100, Thorlabs) antes da roda de filtros na detecção, introduzindo perda de intensidade> 50% na detecção de fluorescência. Embora a distribuição do sinal sLS em geral mude com a orientação do polarizador, o sinal de autofluorescência tecidual permanece não afetado pela rotação do polarizador. sLS produz uma resolução espacial de 2.4 ± 0.3 µm em BABB, que é comparável à resolução em imagens de folha de luz de fluorescência (confirmada pelo ajuste de uma função gaussiana ao XY resposta de imagem de uma única partícula, Fig. Complementar. 8l–m) e próximo da resolução teórica no ar (1.53 µm com abertura numérica (NA) = 0.2 com zoom macro máximo ×8).
Processamento de imagens e análise 3D
O processamento de imagens, segmentação e análise de conjuntos de dados de lightsheets foram feitos com ImageJ/Fiji, enquanto as Figs. 3 e 4 foram gerados com Imaris Viewer 9.9 (https://imaris.oxinst.com/imaris-viewer) e vídeo complementar 3 foi gerado com Imaris 9 (https://imaris.oxinst.com/) (Bitplane, Oxford Instruments). Conjuntos de dados de lightsheet lado a lado foram costurados com MosaicExplorerJ53. A segmentação 3D do tecido da bexiga foi realizada usando macros ImageJ/Fiji personalizadas para anotação 3D semiautomática de grandes volumes no modo virtual. Resumidamente, um primeiro script, ‘Macro1’, carrega pilhas de imagens 3D, permite ao usuário anotar ROIs em vários planos e interpola automaticamente as ROIs para gerar e exportar máscaras 3D. Os ROIs foram desenhados a cada 15 planos (a cada 37.5 µm) para facilitar a boa continuidade da segmentação, mantendo as anotações em um mínimo razoável. Um segundo script, ‘Macro2’, realiza as operações matemáticas ou booleanas, plano por plano, sem carregar todas as pilhas na memória, seja entre máscaras 3D ou entre uma máscara 3D e os dados originais, salvando o resultado como uma nova pilha. Todas as máscaras foram geradas anotando imagens de autofluorescência.
Ambas as camadas superficiais do tumor e do tecido saudável (Fig. 3) foram delineados usando a varinha de Fiji e ferramentas de laço na cavidade da bexiga em uma máscara. Chamando esta primeira iteração de BC1, as execuções subsequentes de Macro1 dilatam automaticamente esse contorno 3D em uma quantidade de pixels definida para produzir novas iterações de máscara, BC2, BC3 e assim por diante, com dilatações crescentes. A primeira camada contendo tumor e tecido saudável, máscara L1, é obtida subtraindo a máscara BC1 de BC2 e assim por diante, produzindo L2 e L3 como camadas concêntricas. O volume do tumor mais próximo da cavidade foi obtido anotando o tumor com ferramentas de varinha e laço para criar uma máscara T1, enquanto a camada 3D de urotélio saudável foi detectada separadamente na máscara U1. Subtrair U1 de L1 produz a camada superficial do tumor e assim por diante: L2 − U1, L3 − U1. Por outro lado, a primeira camada do urotélio é obtida subtraindo T1 de L1. Todas as camadas na Fig. 3 foram definidos para ter 33 µm de espessura.
O mesmo conjunto de macros e procedimentos (ferramenta de varinha ImageJ, erosão digital de 500 µm e assim por diante) foi usado para delinear e segmentar a parte interna do tecido da bexiga e depois estimar o volume do tecido interno da bexiga (Fig. 4, veja detalhes acima). Histogramas da intensidade do sinal disperso foram criados em Fiji combinando o sinal disperso e a máscara.
RNT usando 131I-nanobots
Entre os dias 8 e 15 após a implantação do tumor, os animais foram divididos em seis grupos (grupos 1 a 6), tentando atingir volumes tumorais médios semelhantes entre os grupos (Tabela 2). Para os experimentos, os animais foram induzidos com anestesia (5% de isoflurano em O puro2) e posicionado em decúbito dorsal antes de esvaziar a bexiga massageando a região abdominal. Imediatamente a seguir, 100 µl do tratamento adequado na concentração de 400 µg ml-1 (Mesa 2) foi instilado na bexiga usando um cateter de calibre 24. O tratamento e o veículo (água ou uréia) permaneceram na bexiga por 1 hora antes da remoção do cateter. A bexiga foi esvaziada novamente por massagem abdominal e os camundongos se recuperaram da anestesia em suas gaiolas, substituindo a serragem da gaiola dos animais 24 horas após o tratamento para remover a contaminação radioativa.
Eficácia terapêutica determinada por ressonância magnética
Dois estudos de ressonância magnética foram realizados em cada camundongo: (1) entre os dias 7 e 14 após a inoculação do tumor para randomizar os animais entre os grupos e medir os volumes tumorais iniciais (pré-tratamento); (2) entre os dias 16 e 21 após a inoculação do tumor (pós-tratamento) para avaliar a eficácia terapêutica. A ressonância magnética foi realizada usando scanners 7 T Bruker BioSpec e 11.7 T Bruker BioSpec (ambos com software ParaVision 7), dependendo da disponibilidade. Isto não afetou os resultados, uma vez que o campo externo não é crítico para imagens anatômicas14. Os experimentos de imagem foram conduzidos usando os mesmos parâmetros de imagem e processamento explicados acima (Rastreamento do tamanho do tumor). No caso do scanner de 11.7 T, a configuração consistia em uma bobina de superfície cardíaca de rato para recepção e uma bobina volumétrica para transmissão. Os volumes tumorais em cada fatia foram determinados a partir de volumes de interesse desenhados manualmente, cobrindo a área do tumor.
Análise estatística
Em estudos de imagem PET, porcentagens de dose injetada (% ID) e dose injetada por volume tumoral (% ID cm-3) foram comparados usando ANOVA unidirecional. As diferenças entre os grupos foram determinadas pelo teste de comparações múltiplas de Tukey. O NTV na seção RNT foi obtido de um t-teste de valores não pareados. A distribuição dos dados foi considerada normal, mas isso não foi formalmente testado. As análises estatísticas foram realizadas com GraphPad Prism v.8.
Resumo de relatórios
Mais informações sobre projeto de pesquisa estão disponíveis no site Resumo do Relatório do Portfólio da Natureza vinculado a este artigo.
- Conteúdo com tecnologia de SEO e distribuição de relações públicas. Seja amplificado hoje.
- PlatoData.Network Gerativa Vertical Ai. Capacite-se. Acesse aqui.
- PlatoAiStream. Inteligência Web3. Conhecimento Amplificado. Acesse aqui.
- PlatãoESG. Carbono Tecnologia Limpa, Energia, Ambiente, Solar, Gestão de resíduos. Acesse aqui.
- PlatoHealth. Inteligência em Biotecnologia e Ensaios Clínicos. Acesse aqui.
- Fonte: https://www.nature.com/articles/s41565-023-01577-y
- :é
- :não
- ][p
- 000
- 1
- 10
- 100
- 11
- 13
- 14
- 15%
- 16
- 160
- 20
- 200
- 2015
- 2017
- 2018
- 2019
- 2020
- 2021
- 22
- 23
- 24
- 25
- 250
- 30
- 300
- 33
- 35%
- 3d
- 400
- 41
- 50
- 500
- 51
- 52
- 53
- 7
- 70
- 8
- 9
- 95%
- a
- Sobre
- acima
- conformidade
- acumulação
- Alcançar
- alcançado
- adquirido
- aquisição de
- aquisição
- em
- ativado
- ativo
- atividade
- adicionado
- acrescentando
- Adicionalmente
- administração
- afetar
- Depois de
- depois
- novamente
- Destinado
- AR
- AL
- alex
- algoritmo
- Todos os Produtos
- permitidas
- juntamente
- A doença de Alzheimer
- entre
- quantidade
- amilóide
- an
- analisa
- análise
- Apresentadora
- e
- animal
- animais
- anticorpo
- qualquer
- aplicações
- aplicado
- Aplicando
- apropriado
- aprovou
- água
- SOMOS
- ÁREA
- artigo
- AS
- assumiu
- suposições
- At
- Autoridades
- automaticamente
- disponibilidade
- disponível
- média
- média
- b
- BABB
- Largura de Banda
- base
- BE
- antes
- comportamento
- ser
- abaixo
- entre
- Bloquear
- corpo
- fervido
- ambos
- Cérebro
- carga
- mas a
- by
- gaiolas
- calculado
- chamada
- Câmera
- Câncer
- casas
- casos
- CCD
- célula
- Células
- Alterações
- carregar
- químico
- Desobstrução
- clique
- Clínico
- Fechar
- bobina
- combinação
- combinando
- comercial
- comitê
- comparável
- comparado
- comparações
- completamente
- completando
- concentração
- condição
- condições
- conduzido
- confiança
- CONFIRMADO
- consecutivo
- continuidade
- continuamente
- ao controle
- controlado
- Por outro lado
- Legal
- corrigida
- Correções
- Correspondente
- Conselho
- acoplado
- cobertura
- crio
- criado
- Criar
- Criatividade
- critérios
- crítico
- Tomografia computadorizada
- personalizadas
- Cortar
- Escuro
- dados,
- conjuntos de dados
- dia
- dias
- de
- definido
- entregando
- Dependendo
- descrito
- Design
- desejado
- detalhes
- detectou
- Detecção
- Determinar
- determinado
- determinando
- desenvolvido
- dispositivo
- diagnósticos
- DID
- diferença
- diferenças
- diferente
- Distribuição
- digital
- diluído
- direção
- Doença
- prato
- Dispersão
- distribuição
- distribuições
- dividido
- do
- parece
- feito
- dosar
- desenhado
- Cair
- duração
- dinâmico
- dinâmica
- e
- E & T
- cada
- fácil
- eco
- eficácia
- ou
- incorporado
- permitir
- permite
- Ponto final
- aprimorada
- Todo
- enzimático
- equipado
- estabelecido
- estabelecimento
- estimativa
- Éter (ETH)
- ética
- Europa
- avaliar
- Cada
- exame
- excedido
- Exceto
- expectativa
- esperado
- experimentar
- experimental
- experimentos
- explicado
- exportar
- externo
- facilitar
- fator
- feminina
- campo
- Figo
- Figura
- preenchida
- filtro
- filtros
- final
- Finalmente
- Primeiro nome
- apropriado
- fixado
- Flash
- seguido
- seguinte
- Escolha
- formulário
- formaldeído
- Formalmente
- adiante
- quatro
- da
- congelado
- função
- mais distante
- lacuna
- ge
- GE Healthcare
- Geral
- gerar
- gerado
- Dando
- Bom estado, com sinais de uso
- gradientes
- Grama
- Verde
- Grupo
- Do grupo
- Growth
- orientações
- Ter
- he
- Saúde
- saúde
- saudável
- Coração
- HTTPS
- Huang
- humano
- icap
- ID
- idêntico
- if
- imagem
- análise de imagem
- imagens
- Imagiologia
- imediatamente
- imerso
- melhora
- in
- incluído
- incorporando
- aumentando
- incubado
- Incubando
- incubação
- de treinadores em Entrevista Motivacional
- índice
- indução
- INFORMAÇÕES
- do estado inicial,
- interior
- dentro
- instrumentos
- integrado
- interativo
- interesse
- interno
- para dentro
- introduzindo
- iteração
- iterações
- ESTÁ
- KDA
- manutenção
- de emergência
- l2
- grande
- laser
- lasers
- camada
- camadas
- partida
- esquerda
- Lente
- lentes
- vida
- leve
- Line
- LINK
- ligado
- carregamento
- cargas
- local
- fora
- Macro
- macros
- a manter
- manutenção
- manualmente
- máscara
- Máscaras
- correspondente
- material
- materiais
- matemático
- Matriz
- máximo
- significa
- a medida
- medido
- medições
- medição
- Mídia
- Memória
- merino
- Metanol
- métodos
- camundongos
- Microscópio
- Microscopia
- minutos
- mínimo
- menor
- misto
- mistura
- ML
- Móvel Esteira
- Moda
- modelo
- modelos
- modificada
- módulo
- molecular
- monitorados
- monitoração
- movimento
- mouse
- MRI
- MS
- MT
- múltiplo
- nanotecnologia
- Natureza
- redes
- Neutro
- Novo
- não
- Ruído
- normal
- objetivo
- observado
- obter
- obtido
- of
- Velho
- on
- ONE
- operando
- operacional
- Operações
- otimizado
- or
- orca
- original
- Acima de
- durante a noite
- Oxford
- Oxygen
- Papel
- parâmetros
- Park
- parte
- partícula
- PBS
- Cavilha
- penetração
- para
- realizada
- executa
- animal de estimação
- Petri
- fases
- articulação
- pixels
- colocado
- avião
- Planos
- platão
- Inteligência de Dados Platão
- PlatãoData
- ponto
- pontos
- pasta
- posição
- posicionado
- Precisão
- preparado
- presente
- anteriormente
- procedimentos
- procedimentos
- em processamento
- progressivamente
- protocolo
- Qi
- quantitativo
- Rádio
- acaso
- Randomizado
- RAT
- Taxa
- relação
- Reagir
- reação
- Leitor
- razoável
- razões
- recebido
- recepção
- gravado
- Recuperar
- Vermelho
- reduzir
- Reduzido
- reduz
- referência
- região
- permaneceu
- permanece
- remover
- Removido
- removendo
- Informou
- Relatórios
- representante
- requeridos
- pesquisa
- resistente
- Resolução
- respectivamente
- resposta
- resultar
- resultando
- Resultados
- robô
- Quarto
- é executado
- s
- SA
- mesmo
- poupança
- digitaliza
- disperso
- SCI
- científico
- escrita
- Segundo
- secundário
- Seção
- seções
- Vejo
- segmento
- segmentação
- selecionado
- Seqüência
- servido
- Configurações
- vários
- folha
- mostrando
- Signal
- Distribuição de sinal
- semelhante
- desde
- solteiro
- SIX
- Tamanho
- Fatia
- pequeno
- So
- sódio
- Software
- solução
- Soluções
- Espacial
- Estabilidade
- pilha
- Pilhas
- estatístico
- stellaris
- Passos
- armazenadas
- listra
- estudante
- caso
- subseqüente
- Subseqüentemente
- suíte
- superfície
- Sintomas
- síntese
- .
- T
- T1
- mesa
- tomar
- tecnologia
- Tecnologias
- telescópio
- modelo
- dez
- teste
- testado
- que
- A
- deles
- Eles
- então
- teórico
- Terapêutico
- terapia
- isto
- completamente
- aqueles
- três
- tridimensional
- Através da
- todo
- tempo
- vezes
- tecido
- para
- juntos
- ferramenta
- ferramentas
- Total
- para
- para
- transformando
- tratamento
- Tritão
- tentando
- tumores
- dois
- tipo
- tipicamente
- não afetado
- para
- até
- sobre
- usava
- Utilizador
- utilização
- Valores
- veículo
- Contra
- via
- Vídeo
- VÍDEOS
- Ver
- Virtual
- visualizar
- vivo
- volume
- volumes
- volumétrico
- andador
- varinha
- foi
- lavagem
- Água
- we
- semanas
- peso
- Bem-estar
- foram
- Roda
- qual
- enquanto
- branco
- Windows
- de
- dentro
- sem
- X
- Produção
- produzindo
- rendimentos
- zefirnet
- zoom