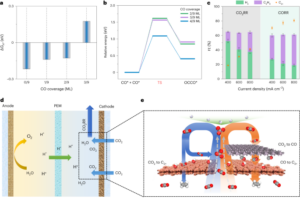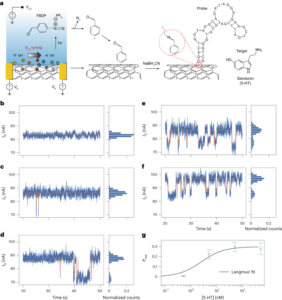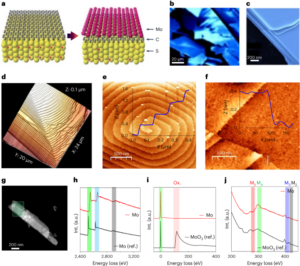
Projekt badania
To badanie zostało zatwierdzone przez McGill Research Ethics Office (Institutional Review Board, A03-M24-21B). Miał on na celu opracowanie uniwersalnej platformy do molekularnego wykrywania infekcji dróg oddechowych w miejscu opieki. Użyliśmy H1N1 grypy A, RNA SARS-CoV-2 i inaktywowanych termicznie cząsteczek wirusa SARS-CoV-2 do szybkiej diagnostyki opartej na wzmocnionych plazmonicznie testach RT-LAMP. RNA MERS-CoV i RNA HCoV-229E zastosowano jako kontrole negatywne w testach RT-LAMP. Ponadto do swoistego wykrywania wariantów SARS-CoV-2, które budzą obawy, zastosowano test RCA. Na koniec użyliśmy 33 niezidentyfikowanych próbek ludzkich SARS-CoV-2-dodatnich (zweryfikowanych RT-PCR) uzyskanych z PRESERVE Pandemic Response Biobank University Health Network i 15 próbek SARS-CoV-2-ujemnych (zweryfikowanych qPCR) jako kontroli w próbce pacjenta badanie. Jako dowód zasady, profilowanie DNA E. coli a MRSA przeprowadzono stosując testy LAMP. Pseudomonas aeruginosa zastosowano jako kontrolę negatywną w testach LAMP. Badanie to przeprowadzono w Montrealu w Kanadzie od kwietnia 2020 r. do grudnia 2022 r.
Materiały
Materiały pozyskano w następujący sposób: nanokulki polistyrenowe (cząsteczki polistyrenu (PS-R); mikrocząstki); syntetyczny SARS-CoV-2 RNA (kolekcja amerykańskich kultur typu (ATCC); Cedarlane); SARS-CoV-2 B.1.1.7 Wariant alfa RNA (ATCC VR-3326D; Cedarlane); MERS-CoV (ATCC VR-3248SD; Cedarlane); inaktywowany termicznie SARS-CoV-2 (ATCC VR-1986HK; Cedarlane); inaktywowany termicznie H1N1 grypy A, szczep NY/01/09 (0810248CFHI; Cedarlane); I E. coli (nr 211540, Merlan Scientific). kolorymetryczna RT-LAMP master mix i enzym HiFi Taq DNA Ligase (New England Biolabs); PLP, startery RCA i syntetyczne cele cDNA SARS-CoV-2, MgClXNUMX2, KCl, dinukleotyd nikotynamidoadeninowy (NAD) i Triton X-100 (Sigma Aldrich); i zastosowano startery LAMP i ditiotreitol (DTT) (Thermo Fisher Scientific). Zdrową ludzką ślinę zebraną (IRHUSL50ML) i zdrową ludzką ślinę od pojedynczego dawcy (IRHUSLS5ML) zakupiono od firmy Innovative Research i przechowywano po przybyciu w temperaturze –80°C. Próbki RNA wariantu SARS-CoV-2 (Omicron, Delta, Eta i Gamma) uzyskano ze współpracującego laboratorium na Uniwersytecie McGill (Vidal Lab). RNA HCoV-229E uzyskano z laboratorium współpracującego w Lady Davis Institute for Medical Research w Jewish General Hospital (laboratorium C. Liang). DNA MRSA i P. aeruginosa DNA uzyskano z laboratorium D. Nguyen w instytucie badawczym McGill University Health Centers (MUHC). Wszystkie testy przygotowano przy użyciu wody destylowanej UltraPure wolnej od DNazy/RNazy (Thermo Fisher Scientific).
Startery i sondy
Startery SARS-CoV-2 RT-LAMP użyte w tym badaniu zostały zaprojektowane przez Yu i in.32 celować w RdRp gen w ORF1ab genomu (rekomendowany przez WHO jako gen docelowy do wykrywania SARS-CoV-2)31. Selektywność startera potwierdzono w eksperymencie elektroforezy żelowej (Uzupełniająca Fig. 16). Składy i stężenia starterów są dostępne w tabeli dodatkowej 6.
Startery H1N1 RT-LAMP zostały zaprojektowane za pomocą narzędzia New England Biolabs LAMP Primer Design Tool ukierunkowanego na wysoce konserwatywne sekwencje genu hemaglutyniny (HA) wirusa H1N1 IAV. Część rozpoznawania LAMP została oceniona przez narzędzie do wyszukiwania podstawowego wyrównania lokalnego50i nie zaobserwowano żadnych zmian sekwencji w 100 trafieniach dostarczonych przez stronę internetową National Center for Biotechnology Information. Nominowane startery LAMP zostały wybrane zgodnie ze zoptymalizowanymi parametrami oferowanymi przez protokół Primer Explorer V5. Selektywność starterów została dodatkowo potwierdzona w eksperymencie elektroforezy żelowej (Uzupełniająca Fig. 16b). Składy i stężenia starterów są dostępne w tabeli dodatkowej 7.
Miejsca mutacji P681H i L452R określono zgodnie z narzędziami CoV-GLUE-Viz i GISAID36,51, a części rozpoznające PLP zostały zaprojektowane tak, aby były specyficzne dla mutacji36. Ponadto miejscem docelowym PLP do wykrywania WT SARS-CoV-2 był RdRp gen, który jest taki sam jak miejsce docelowe dla starterów SARS-CoV-2 LAMP. PLP są zaprojektowane w taki sposób, że SNP wyróżnia się przez górną część PLP, która zapewnia grupę 3′-hydroksylową na złączu ligacyjnym sparowaną zasadą obok fosforylowanego końca 5′ nici docelowej. Prawdziwość cyrkulacji PLP po wykryciu SNP została określona przy użyciu kalkulatora temperatury reakcji termostabilnej ligazy dostarczonego przez New England Biolabs. Następnie PLP oceniano przy użyciu serwera sieciowego Mfold, aby uniknąć niepożądanej struktury drugorzędowej, zwłaszcza w miejscu rozpoznawania PLP. Startery do przodu i do tyłu RCA zaprojektowano do hybrydyzacji z częścią rozdzielającą PLP, która łączy razem dwa określone ramiona PLP. Selektywność PLP potwierdzono za pomocą eksperymentu elektroforezy żelowej (Uzupełniająca Fig. 21). Kompozycje i stężenia PLP, a także kompozycje i stężenia starterów RCA oraz skład cDNA są dostępne w tabelach uzupełniających 8-10.
Bakteryjne startery LAMP zastosowane w tym badaniu zostały zaprojektowane przez Hilla i in.52 za celowanie w E. coli malB gen i Chen i in.53 do zwalczania MRSA mecA gen. Składy i stężenia starterów są dostępne w tabelach uzupełniających 11 i 12.
Przygotowanie testu
Do testów RT-LAMP zastosowano standardową objętość reakcyjną 20 ul. Objętość ta składała się z 2 µl x 10 starterów, 10 µl x 2 master mix, 7 µl wody wolnej od RNaz i 1 µl próbki RNA. Próbki wirusa inaktywowane termicznie najpierw poddano lizie termicznej w temperaturze 95 ° C przez 3 minuty, a następnie zmieszano z testem. Następnie inkubowano w temperaturze 65°C przez różne okresy w celu wizualizacji zmiany koloru w funkcji czasu dla różnych próbek.
Reakcję ligacji PLP przeprowadzono w końcowej objętości 10 μl, w tym 1 μl syntetycznego cDNA genomu RNA SARS-CoV-2, 1 μl 1 μM PLP, 2 μl wody destylowanej UltraPure i 5 μl × 2 no-Tris-HCl HiFi Roztwór do ligacji Taq DNA Ligase (20 mM MgClXNUMX2, 20 mM KCl, 2 mM NAD, 0.1% Triton X-100, 20 mM DTT, pH 8.50) i 1 μl ligazy DNA HiFi Taq. Mieszaninę ligacyjną najpierw inkubowano w 95 ° C przez 5 minut w celu denaturacji DNA, a następnie schłodzono do temperatur hybrydyzacji PLP (które wynoszą odpowiednio 60, 58 i 55 ° C dla PLP P681H, L452R i WT), aby pozwolić PLP na hybrydyzację z cDNA i ligat za pomocą enzymu HiFi w termocyklerze (Analytik Jena). Następnie w reakcji ligacji wykorzystano 12 μl WarmStart Colorimetric LAMP × 2 Master Mix i 1.6 μM RCA startery do tyłu i do przodu w końcowej objętości 24 μl. Reakcję amplifikacji RCA przeprowadzono w temperaturze 65°C przez różne okresy, aby zwizualizować zmianę koloru w funkcji czasu dla różnych próbek.
Do ekstrakcji bakteryjnego DNA, E. coli próbki hodowano przez noc w 37°C w pożywce Luria Broth. Stężenie bakterii określono za pomocą spektrofotometru Spectronic 21D. Porcje o różnych stężeniach po 107 CFU ml-1, 105 CFU ml-1, 104 CFU ml-1, 103 CFU ml-1, 102 CFU ml-1 i 10 CFU ml-1 przygotowano przez zawieszenie E. coli kultury w pożywce Luria Broth. E. coli DNA ekstrahowano przez gotowanie kultur w temperaturze 95°C przez 10 minut. DNA MRSA uzyskano z Centrum Zdrowia Uniwersytetu McGill przy użyciu metody lizy chemicznej. Stężenia wszystkich próbek DNA zmierzono za pomocą spektrofotometru Nanodrop 2000 i zawieszono w buforze Universal Buffer 48 (Bio Basic) w celu uzyskania pożądanych stężeń.
Test QolorEX dla SARS-CoV-2 przetestował roztwory wzbogacone (woda wolna od RNaz i zdrowa ślina) 8 × 105 kopie RNA μl-1 do 5 kopii RNA µl-1 RNA SARS-CoV-2 i 90 cząsteczek wirusa μl-1 do 0.01 cząstek wirusa μl-1 aby inaktywowany termicznie SARS-CoV-2 mieścił się w biologicznie istotnym zakresie. Podobnie przeprowadzono badanie dla Delta B.1.617.2, Omicron B.1.1.529, Omicron BA.4, Omicron BA.2.21, Omicron BA.5.2, Omicron BA.5.1.1, Eta B.1.525 i Gamma P .1 z 8 × 105 kopie RNA μl-1 do 104 kopie RNA μl-1.
Test QolorEX dla H1N1 badał wzbogacone roztwory (woda wolna od RNaz i zdrowa ślina) 8 × 105 kopie RNA μl-1 do 5 kopii RNA µl-1 RNA grypy A H1N1. Do badań selektywności użyto RNA z wielu wirusów (SARS-CoV-2, MERS-CoV i HCoV-229E) przy 8 × 105 kopie RNA μl-1.
Test QolorEX dla E. coli badali wzbogacone roztwory (woda wolna od RNaz) 7.2 × 106 kopie gDNA ml-1 do 7.2 kopii gDNA ml-1, co odpowiada 0.0343 ng μl-1 do 3.43 × 10-8 ng μl-1 of E. coli DNA. Do badań selektywności DNA z wielu bakterii (E. coli, MRSA i P. aeruginosa) badano w stężeniu 102 kopie gDNA μl-1.
Test QolorEX dla MRSA badał wzbogacone roztwory (woda wolna od RNaz) 105 kopie gDNA ml-1 do 1 kopii gDNA ml-1, co odpowiada 3.05 × 10-4 ng μl-1 do 3.05 × 10-9 ng μl-1 DNA MRSA. Do badań selektywności DNA wielu bakterii (MRSA, E. coli i P. aeruginosa) badano w stężeniu 102 kopie gDNA μl-1.
Test QolorEX RCA przeprowadzono w roztworach wzbogaconych (woda wolna od RNaz i zdrowa ślina) 8 × 105 cDNA kopiuje ul-1 do 5 kopii cDNA ul-1 syntetycznego cDNA sekwencji P681H, L452R i WT SARS-CoV-2. Pod względem selektywności PLP P681H oceniano w obecności cDNA P681H, WT-P681H i L452R. Tę samą strategię zastosowano do testowania selektywności PLP L452R przy użyciu docelowych cDNA L452R, WT-L452R i P681H, jak również PLP WT w obecności sekwencji cDNA WT, P681H i L452R. Wszystkie cele cDNA oceniono w stężeniach 105 cDNA kopiuje ul-1.
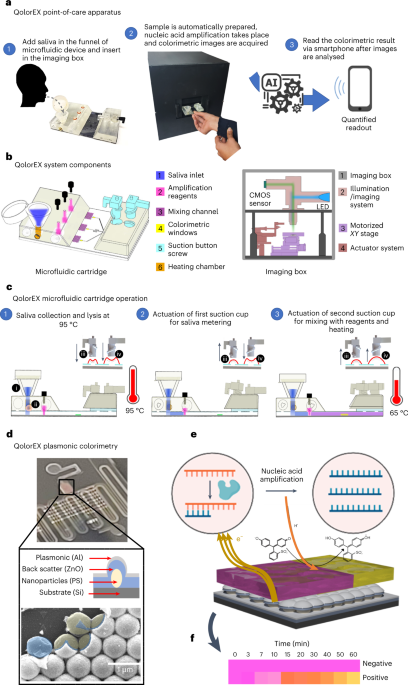
Zautomatyzowane pudełko do obsługi próbek i obrazowania QolorEX
Przenośna konfiguracja obrazowania składa się z trzech głównych elementów: konfiguracji obrazowania epi-iluminacyjnego, modułu obsługi płynów i elementów automatyki (rys. 1). Zewnętrzna obudowa zestawu jest w całości wykonana przez drukowanie 3D metodą osadzania topionego (Prusa I3 Mk3, Prusa) z rozdzielczością warstwy 0.3 mm.
Konfiguracja obrazowania epiluminacyjnego została zaprojektowana w celu uchwycenia zmiany kolorymetrycznej roztworu testowego. Układ obrazowania obejmuje moduł oświetlenia oraz moduł przechwytywania i przetwarzania obrazu. Obiektyw ×20 (TU Plan Fluor EPI ×20, Nikon) został użyty jako soczewka kondensująca do przechwytywania obrazu. Wszystkie zastosowane komponenty optyczne zostały zakupione w firmie Thorlabs. Ostateczny obraz jest wyświetlany na matrycy CMOS (Sony IMX477R, 12.3 MP, Raspberry Pi.) i dalej przetwarzany przez Raspberry Pi 4 (Raspberry Pi).
Do automatyzacji procesów zastosowaliśmy pięć siłowników liniowych (Actuonix), które wykonują wiele etapów obsługi płynów (rysunki uzupełniające). 1-3). Zestaw wykorzystuje moduł grzewczy, którym jest przenośna lutownica (TS-100), do lizy próbek. Jakiś XY etap translacji został wykorzystany do skanowania różnych obszarów wewnątrz i między obszarami komór detekcyjnych. Zastosowaliśmy sterowany liniowo, skomputeryzowany stopień sterowania numerycznego, który jest napędzany silnikiem krokowym (FUYU). Sterowanie zarówno układem wykonawczym, jak i stolikiem odbywa się za pomocą mikrokontrolera Arduino UNO (Arduino). Dwa elementy grzejne są sterowane za pomocą dwukanałowego modułu przekaźnikowego (Yizhet). Zautomatyzowanym przesyłaniem i analizą danych w naszym systemie steruje mikrokontroler Raspberry Pi 4 (Raspberry Pi). Wyniki są wyświetlane w aplikacji zaprojektowanej przez MIT App Inventor (rys. 2).
Produkcja wkładu mikroprzepływowego i platformy plazmonicznej
Proces produkcji przebiegał zgodnie z ustalonym protokołem27,54,55 i jest szczegółowo omówiony w Dodatkowa informacja (Uzupełniająca ryc. 5). W skrócie, wkład mikroprzepływowy oparty jest na sześciocalowej płytce krzemowej. Trzyetapowy proces litograficzny jest stosowany do modelowania utrudnionego elementu grzejnego i cech mikroprzepływowych. Najpierw przeprowadza się etap litografii w celu przeniesienia elementu grzejnego (szerokość 400 µm) i podkładki (długość 5 mm; szerokość 2 mm) na warstwę fotorezystu przez fotomaskę z pożądanymi wzorami. Następnie następuje wytrawianie buforowanym tlenkiem w celu usunięcia rodzimego tlenku i wytrawianie wodorotlenkiem potasu w przypadku wytrawiania krzemem 200 nm. Następnie przeprowadzany jest proces podnoszenia w celu selektywnego osadzania elementów grzejnych w wytrawionych rowkach. Rozpoczyna się to drugim etapem litografii, po którym następuje osadzanie Al 240 nm wiązką elektronów przy użyciu Temescal BJD 1800. W związku z tym odrywanie jest zakończone przez zanurzenie w odpowiednim zmywaczu. Następnie przeprowadzany jest ostatni etap litografii w celu ukształtowania cech urządzenia płynowego, w tym portów wlotowych/wylotowych (średnica 2 mm), komorę lizy (długość 1.74 mm; szerokość 1.5 mm; głębokość 50 urn), kanały mieszania (szerokość 200 urn; wysokość 50 urn) i okienko plazmoniczne (długość 1.94 mm; szerokość 1.5 mm; wysokość, 50 µm) w warstwę SU-8 (SU-8 2050). Następnie sześciocalowy wafel kroi się w kostkę na pojedyncze czipsy (długość 26.5 mm; szerokość 35 mm) za pomocą piły do kostkowania Disco DAD 3240.
Następnie następuje integracja wrażliwych na kolor platform we wkładzie mikroprzepływowym przy użyciu bezbłędnej techniki nanowzorców. Zastosowano ogólne podejście do opracowania koloidalnej samoorganizującej się monowarstwy nanocząstek na granicy faz woda-powietrze56. Następnie powstałe struktury plastra miodu są przenoszone do komory wykrywania koloru we wkładzie mikroprzepływowym. Następnie osadza się cienką warstwę ZnO (120 nm), a następnie cienką warstwę Al (10 nm), aby zapewnić przestrajalny zlokalizowany powierzchniowy rezonans plazmonowy z białym tłem.
Polidimetylosiloksan (PDMS) jest przygotowywany przy użyciu stosunku 10:1 elastomeru do środka sieciującego, odgazowywany w eksykatorze i inkubowany w temperaturze 65°C przez noc. Utwardzony PDMS jest cięty do rozmiaru wkładu mikroprzepływowego, a porty wlotowe/wylotowe są dziurkowane za pomocą dziurkacza PDMS (Thermo Fisher Scientific) i używane do uszczelniania urządzeń. Proces zamykania obejmuje 50-sekundową obróbkę plazmą, po której następuje całonocna inkubacja w temperaturze 105°C. Następnie wytworzono warstwę przyssawek opartą na PDMS przy użyciu form stereolitograficznych (SLA) wydrukowanych w 3D (Form 3, Formlabs). Przyssawki zostały następnie połączone z płynną warstwą PDMS przez wiązanie aktywowane plazmą.
Przykładowe kartridże do przetwarzania (wyprodukowane przy użyciu drukarki 3D SLA w rozdzielczości 50 µm na z oś) są łączone z chipem mikroprzepływowym pokrytym PDMS za pomocą dwustronnej taśmy sprzyjającej wiązaniu aktywowanemu plazmą i umieszczane w piecu w temperaturze 95 ° C na 90 minut. W kartridżu wydrukowanym w 3D znajdują się dwie mosiężne metalowe wkładki (McMaster Carr) do lizy próbki.
Przetwarzanie obrazu
Przetwarzanie obrazu rozpoczyna się od zestawu danych składającego się z trzech powtórzeń obrazów z każdego badanego stanu, które zostaną podzielone na miniobrazy w celu wygenerowania łącznie 40 i 60 punktów danych na warunek odpowiednio dla czułości i selektywności. Specjalnie dla próbek klinicznych, każdą badano w trzech równoległych komorach zbiorczych chipa mikroprzepływowego; ponieważ wszystkie zdjęcia wykonano w trzech powtórzeniach, a następnie podzielono, dla każdej próbki pacjenta zbadano łącznie 90 punktów danych. Całe przetwarzanie obrazu polega na przycięciu zewnętrznych 20% oryginalnego obrazu RGB w celu usunięcia efektu pierścienia kawy57, po czym następuje zastosowanie filtra niebieskiego, w którym piksele o wartości odcienia między 85 a 140 (zakres koloru niebieskiego) są usuwane i zastępowane średnią wartością pozostałej części obrazu57. Obraz z filtrem niebieskim jest następnie progowany przez zastąpienie o 25% mniej nasyconych pikseli średnią wartością pozostałej części filtrowanego obrazu. Na koniec przetworzony obraz jest cięty na podobrazy, z których wyodrębnia się kilka cech wynikających z implementacji formuł wyszczególnionych w Sergyan58. Modyfikacja formuł polega na zamianie intensywności skali szarości z intensywnością każdego kanału RGB58. Z każdego podobrazu wyodrębnianych jest łącznie 18 wartości odpowiadających średniej wartości koloru, odchyleniu standardowemu, trybowi, pochyleniu, energii i entropii dla każdego z kanałów RGB.
W celu automatyzacji prawdziwych ludzkich próbek zaimplementowaliśmy nadzorowany algorytm uczenia maszynowego, aby sklasyfikować obrazy na dwie klasy, niezainfekowane i zainfekowane (rys. 14). Utworzono SVM z jądrem radialnej funkcji bazowej (RBF) z jej hiperparametrami C i gamma oceniane przez przeszukiwanie Bayesa59 oraz brak nadmiernego dopasowania potwierdzony pięciokrotną walidacją krzyżową. Baza danych dla tego badania jest zintegrowana z 33 zainfekowanymi i 15 niezainfekowanymi kontrolami; dla każdej próbki badania przeprowadzono w trzech powtórzeniach, uzyskując łącznie dziewięć obrazów na punkt czasowy. Zbiory danych są podzielone na dwie klasy: niezainfekowane (negatywne) i zainfekowane (pozytywne). Następnie są one dzielone na odrębne zestawy treningowe i testowe. Zestaw treningowy składa się z dwóch trzecich wektorów od pacjentów 1, 7, 9, 11, 15, 19, 21, 23, 25, 29 i 31 oraz negatywów 2, 3, 4, 5, 6, 7, 8, 9, 11, 13 i 15; zbiór testowy jest całkowany przez pozostałe wektory. SVM tworzy prognozę dla każdego wektora zestawu testowego jako niezainfekowanego lub zainfekowanego.
Próbki kliniczne i oświadczenie etyczne
Pobraliśmy próbki śliny z University Health Network's PRESERVE Pandemic Response Biobank (UHN REB 20-5364 i McGill IRB nr A03-M24-21B). 33 próbki kliniczne SARS-CoV-2 pobrano od dorosłych pacjentów z objawami COVID-19, takimi jak gorączka, zmęczenie i suchy kaszel. Wszystkie próbki zostały zdeidentyfikowane i uzyskały pozytywny wynik testu na obecność SARS-CoV-2 przy użyciu RT-PCR. Ponadto miano wirusa oceniono metodą qPCR (QuantStudio 12K Flex, Thermo Fisher Scientific). Sekwencje starterów qPCR podano w tabeli dodatkowej 13. Próbki zostały ocenione w placówce poziomu 2+ znajdującej się w Instytucie Lady Davis w Żydowskim Szpitalu Ogólnym.
Pomiary elektrochemiczne
Pomiary elektrochemiczne przeprowadzono w konwencjonalnym ogniwie trójelektrodowym przy użyciu potencjostatu/galwanostatu Autolab PGSTAT204. Platformy plazmoniczne zostały użyte jako elektrody pracujące, podczas gdy Ag/AgCl i drut platynowy służyły odpowiednio jako elektrody odniesienia i przeciwelektrody. Potencjał cyklicznych testów woltamperometrii wahał się od −1 do 1 V w porównaniu z elektrodą odniesienia przy szybkości skanowania 50 mV s-1. Pomiar fotoreakcji przeprowadzono stosując technikę chronoamperometrii w przerywanym świetle widzialnym otoczenia (cykle włączania i wyłączania światła, 5 s) przy potencjale polaryzacji 1 V względem Ag/AgCl w wodnym roztworze testowym do amplifikacji kwasów nukleinowych (10 części LAMP Master Wymieszać, 2 części × 10 części startera, 7 części wody wolnej od RNaz i 1 część próbki docelowego RNA).
Charakteryzacja
Do badań optymalizacyjnych platform plazmonicznych wykorzystano techniki charakteryzacji fizycznej, w tym mikroskopię sił atomowych (AFM) i SEM. AFM przeprowadzono za pomocą sprzętu Bruker MultiMode8, podczas gdy SEM przeprowadzono za pomocą środowiskowego skaningowego mikroskopu elektronowego FEI Quanta 450.
Charakterystykę optyczną przeprowadzono za pomocą sprzętu widzialnego Lambda750 bliskiej podczerwieni-UV, w którym pomiary absorpcji światła białego podczas 60 minut amplifikacji zbierano od 200 nm do 850 nm. Ustawienie badania obejmowało akumulację rozwiązań punktu czasowego na platformie, w której padające i zebrane wiązki światła miały normalny wpływ na platformę. Podstawowy rozkład pola elektrycznego platformy został zbadany przy użyciu modułu różnic skończonych w dziedzinie czasu (wersja 8.21.1781, Lumerical Solutions). Podstawowe właściwości przepływu płynu i wymiany ciepła wkładu mikroprzepływowego zbadano za pomocą COMSOL Multiphysics (wersja 5.6).
Analiza statystyczna
Wyniki są nadawane jako wartość średnia ± błąd standardowy średniej dla potrójnych pomiarów, jak wyjaśniono w części dotyczącej przetwarzania obrazu. Do analizy statystycznej wykorzystano pakiet oprogramowania OriginPro (OriginLab, 2021). Granice wykrywalności i zakresy liniowe obliczono przy użyciu metod regresji liniowej, w tym nachylenia linii i błędu standardowego punktu przecięcia. Istotność statystyczną oceniono stosując jednokierunkową analizę wariancji (ANOVA) z testem post hoc Tukeya dla porównania średnich. Różnicę w zbiorze danych uznano za istotną statystycznie dla P < 0.001. Aplikacja do tworzenia wykresów Paired Comparison Plot (v.3.60, OriginLab) została wykorzystana do wygenerowania danych przy użyciu P wartości.
Podsumowanie raportowania
Dalsze informacje na temat projektu badań są dostępne w Podsumowanie sprawozdawczości dotyczącej portfela przyrody powiązane z tym artykułem.
- Dystrybucja treści i PR oparta na SEO. Uzyskaj wzmocnienie już dziś.
- PlatoAiStream. Analiza danych Web3. Wiedza wzmocniona. Dostęp tutaj.
- Wybijanie przyszłości w Adryenn Ashley. Dostęp tutaj.
- Kupuj i sprzedawaj akcje spółek PRE-IPO z PREIPO®. Dostęp tutaj.
- Źródło: https://www.nature.com/articles/s41565-023-01384-5
- :ma
- :Jest
- :Gdzie
- ][P
- 1
- 10
- 100
- 11
- 12
- 13
- 14
- 15%
- 20
- 200
- 2008
- 2011
- 2016
- 2018
- 2019
- 2020
- 2021
- 2022
- 2023
- 2050
- 23
- 24
- 26
- 27
- 31
- 3d
- 3D druku
- 40
- 50
- 60
- 66
- 7
- 8
- 9
- a
- AC
- przyśpieszony
- Stosownie
- odpowiednio
- akumulacja
- Osiągać
- nabywanie
- dodatek
- Dorosły
- AL
- algorytm
- Wszystkie kategorie
- Alfa
- całkowicie
- Rozproszone
- amerykański
- Wzmocnienie
- an
- analiza
- Kotwica
- i
- Aplikacja
- Zastosowanie
- stosowany
- podejście
- zatwierdzony
- kwiecień
- Arduino
- SĄ
- Ramiona
- przylot
- artykuł
- AS
- oceniać
- At
- zautomatyzowane
- Automatyzacja
- dostępny
- uniknąć
- Oś
- tło
- Bakteria
- na podstawie
- podstawowy
- podstawa
- Bayesian
- BE
- być
- pomiędzy
- stronniczość
- biotechnologia
- krew
- Niebieski
- deska
- obie
- kupiony
- mosiądz
- krótko
- bufor
- by
- obliczony
- Kanada
- Rak
- Komórki nowotworowe
- zdobyć
- który
- prowadzone
- nieść
- Komórki
- Centrum
- Centra
- centrum
- CGI
- Izba
- zmiana
- kanały
- Charakterystyka
- chemiczny
- chen
- żeton
- Frytki
- Klasy
- Klasyfikuj
- kliknij
- Kliniczne
- Kawa
- kolekcja
- wspólny
- w porównaniu
- porównanie
- Zakończony
- całkowicie
- składniki
- w składzie
- Składa się
- stężenie
- Troska
- warunek
- przeprowadzone
- przyznane
- ZATWARDZIAŁY
- łączy
- wynikający
- konserwatywny
- za
- składa się
- kontrola
- kontrolowanych
- kontroli
- Konwencjonalny
- kopie
- Koronawirus
- Odpowiedni
- Przeciwdziałać
- COVID-19
- kultura
- Kubek
- Ciąć
- Cykle
- Tatuś
- dane
- punkty danych
- Baza danych
- zbiory danych
- Davis
- grudzień
- Delta
- zdeponowany
- głębokość
- Wnętrze
- zaprojektowany
- życzenia
- szczegółowe
- Wykrywanie
- ustalona
- rozwijać
- odchylenie
- urządzenie
- urządzenia
- różnica
- różne
- odrębny
- Wybitny
- 分配
- podzielony
- DNA
- zrobić
- napędzany
- suchy
- podczas
- E i T
- każdy
- bądź
- element
- Elementy
- zatrudniony
- zatrudnia
- Umożliwia
- zakończenia
- energia
- Anglia
- wzmocnione
- środowiskowy
- EPI
- sprzęt
- Równoważny
- błąd
- szczególnie
- ustanowiony
- Eter (ETH)
- etyka
- oceniane
- Każdy
- eksperyment
- wyjaśnione
- badacz
- ekstrakcja
- Łatwość
- zmęczenie
- Korzyści
- fei
- Gorączka
- Figa
- Postacie
- Film
- filmy
- filtrować
- finał
- W końcu
- i terminów, a
- dopasować
- pływ
- płyn
- następnie
- następujący sposób
- W razie zamówieenia projektu
- wytrzymałość
- Nasz formularz
- Naprzód
- od
- funkcjonować
- fundamentalny
- dalej
- Ogólne
- Generować
- Genom
- Zarządzanie
- miał
- Prowadzenie
- Zdrowie
- zdrowy
- wysokość
- wysoko
- Odsłon
- Szpital
- HTTPS
- człowiek
- Uwodornienia
- i
- i3
- IEEE
- obraz
- zdjęcia
- Obrazowanie
- realizacja
- realizowane
- in
- incydent
- obejmuje
- Włącznie z
- inkubowane
- inkubacja
- indywidualny
- zakażenia
- Grypa
- Informacja
- Innowacyjny
- Wkłady
- Instytut
- instytucjonalny
- zintegrowany
- integracja
- Inteligencja
- zamierzony
- na świecie
- najnowszych
- IT
- JEGO
- laboratorium
- laboratorium
- dama
- Nazwisko
- warstwa
- nauka
- Długość
- Obiektyw
- mniej
- poziom
- lekki
- Limity
- Linia
- LINK
- powiązany
- załadować
- miejscowy
- maszyna
- uczenie maszynowe
- zrobiony
- poważny
- zrobiony fabrycznie
- mistrz
- Mata
- materiał
- oznaczać
- pomiary
- Pomiary
- Media
- medyczny
- badania medyczne
- metal
- metoda
- metody
- Mikroskop
- Mikroskopia
- min
- MIT
- mieszany
- Mieszanie
- mieszanina
- Moda
- modelowanie
- moduł
- Cząsteczkowa
- monitorowanie
- Montreal
- Ponadto
- Silnik
- MRSA
- wielokrotność
- Mutacja
- nanotechnologia
- narodowy
- rodzimy
- Natura
- ujemny
- Nowości
- Następny
- Nguyen
- NIH
- Nie
- normalna
- cel
- uzyskane
- of
- oferowany
- Biurowe
- on
- Elementy optyczne
- optymalizacja
- zoptymalizowane
- or
- organizacja
- oryginalny
- ludzkiej,
- na zewnątrz
- w ciągu nocy
- pakiet
- ścieżka
- sparowany
- pandemiczny
- Parallel
- parametry
- część
- cząstka
- strony
- pacjent
- pacjenci
- Wzór
- wzory
- wykonywane
- okresy
- fizyczny
- krok po kroku
- Plazma
- Platforma
- Platformy
- platyna
- plato
- Analiza danych Platona
- PlatoDane
- punkt
- zwrotnica
- teczka
- porty
- pozytywny
- Post
- potencjał
- przepowiednia
- przygotowany
- obecność
- elementarz
- zasada
- druk
- PROC
- wygląda tak
- Automatyzacja procesów
- obrobiony
- przetwarzanie
- Profilowanie
- Przewiduje
- dowód
- protokół
- zapewniać
- pod warunkiem,
- zapewnia
- ilościowy
- zasięg
- szybki
- Malina
- Raspberry Pi
- Kurs
- stosunek
- Rbf
- reakcja
- real
- w czasie rzeczywistym
- uznanie
- Zalecana
- regresja
- Przekaźnik
- wydany
- pozostały
- usunąć
- Usunięto
- otrzymuje
- Raportowanie
- Badania naukowe
- Rozkład
- rezonans
- odpowiednio
- odpowiedź
- Odpowiedzi
- REST
- wynikły
- Efekt
- rewers
- przeglądu
- RGB
- Pierścień
- RNA
- s
- Ślina
- taki sam
- SARS CoV-2
- waga
- skanować
- skanowanie
- SCI
- Szukaj
- druga
- wtórny
- Sekcja
- widziany
- wybrany
- selektywny
- SEM
- wrażliwy
- Wrażliwość
- Sekwencja
- zestaw
- Zestawy
- ustawienie
- kilka
- shared
- Sigma
- znaczenie
- znaczący
- Krzem
- Podobnie
- ponieważ
- witryna internetowa
- Witryny
- Rozmiar
- ukośnie
- Nachylenie
- mały
- Tworzenie
- rozwiązanie
- Rozwiązania
- Sony
- pozyskiwany
- specyficzny
- swoiście
- STAGE
- standard
- rozpocznie
- statystyczny
- Ewolucja krok po kroku
- Cel
- stany magazynowe
- przechowywany
- Odmiany Konopi
- Strategia
- Struktura
- Studiował
- badania naukowe
- Badanie
- Następnie
- taki
- odpowiedni
- Powierzchnia
- zawieszony
- Sympozjum
- objawy
- syntetyczny
- system
- stół
- Zadania
- biorąc
- cel
- kierowania
- cele
- Techniki
- test
- Testowanie
- Testy
- że
- Połączenia
- następnie
- Tam.
- one
- to
- trzy
- trzystopniowy
- Przez
- czas
- do
- narzędzie
- Kwota produktów:
- Trening
- przenieść
- przeniesione
- Tłumaczenie
- leczenie
- Tryton
- drugiej
- dwie trzecie
- rodzaj
- dla
- uniwersalny
- uniwersytet
- ONE
- na
- używany
- za pomocą
- zatwierdzony
- wartość
- Wartości
- Wariant
- zweryfikowana
- wszechstronny
- Przeciw
- przez
- wirusowy
- Wirusy
- widoczny
- Tom
- była
- Uzdatnianie wody
- Droga..
- we
- sieć
- serwer wWW
- Strona internetowa
- DOBRZE
- były
- który
- Podczas
- biały
- KIM
- będzie
- Drut
- w
- pracujący
- świat
- Światowa Organizacja Zdrowia
- X
- zefirnet