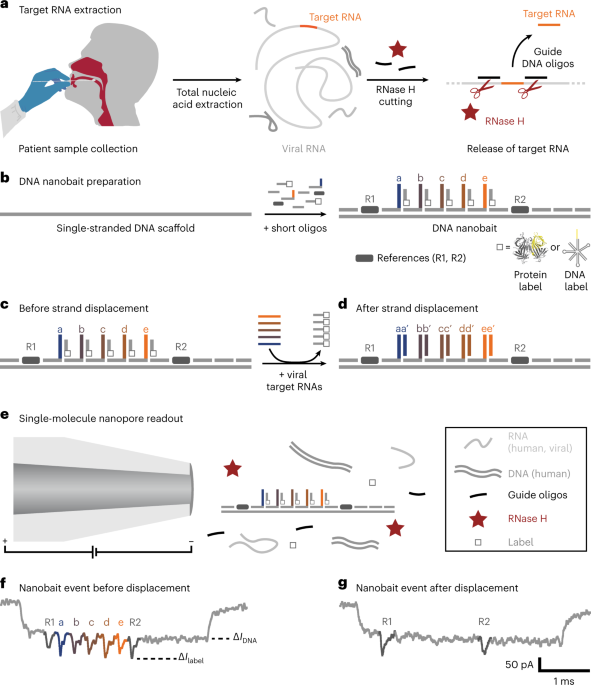
איסוף דגימות של מטופל
דגימות מטופלים נאספו על ידי ניקוי החלק האחורי של הגרון (ספוגית אורופ-לוע) של מטופלים, כפי שתואר קודם לכן26. הדגימות נאספו מחולים עם תמונה קלינית דמוית COVID-19 ונבדקו עם qRT-PCR לאחר מיצוי חומצת גרעין. בקצרה, לאחר האיסוף, ספוגיות הונחו לתוך צינור דגימה מסומן המכילה חיץ תמוגה (4 M guanidine thiocyanate, 25 mM Tris-HCl, 0.5% β-mercaptoethanol ו-MS2 RNA (200 ng μl)-1; רוש)). הצינור היה נסער בעדינות כדי להבטיח חלוקה שווה של חיץ תמוגה. שלבי הבטיחות תוארו בעבר ובוצעו במעבדת CL2 מוסמכת26.
מיצוי חומצה גרעינית
חומצת הגרעין הכוללת הופקה באמצעות מערכות מבוססות עמודות ספין וכפי שהופעלו על ידי בדיקת qRT-PCR סטנדרטית26. בקרת ההגברה הפנימית (MS2 (~6 × 104 PFU ml-1) לכל 10 מ"ל של חיץ תמוגה) נוספה במאגר התמוגג המטען (25 μl לכל 10 מ"ל של חיץ תמוגה). הדגימה נפלטה ב-100 μl של מים נטולי נוקלאז (nfH2O; Invitrogen) ונשאר לעמוד דקה אחת לפני צנטריפוגה למשך דקה אחת ב-1×g (15,000 סל"ד) במיקרופוג' מטופח. הדגימות שנפלטו עברו ישירות ל-qRT-PCR. תמציות חומצת הגרעין הנותרות אוחסנו ב-80 מעלות צלזיוס ושימשו עוד יותר עבור חישת ננובייט-ננופור.
qRT-PCR עבור SARS-CoV-2
זיהוי SARS-CoV-2 בוצע כפי שתואר קודם לכן26. לכל תגובה, תערובת המאסטר הכילה 12.5 μl של 2x Luna Universal Probe One-Step תערובת תגובה, 0.5 μl של 20 μM Wu קדימה פריימר (5′-ATGGGTTGGGATTATCCTAAATGTGA-3′), 0.5 μl של 20 μM Wu5 primer הפוך. -GCAGTTGTGGCATCTCCTGATGAG-3′), 0.3 µl של 10 µM MGB Probe 3 פלואורססאין (5′-ATGCTTAGAATTATGGCCTCAC-3′), 0.5 µl של 10 µM של 2 µM של 0.5 µM של MGB Probe 10 פלואורסצין עבור 2 µl reverse primer בקרה פנימית של MS0.3 µl עבור 10. MS2 RNA, 1 µl של בדיקה פנימית של 3.9 µM (MSXNUMX ROX), XNUMX µl של Luna WarmStart RT מיקס אנזים ו-XNUMX µl של nfH2O. לאחר מכן, 20 μl של תערובת המאסטר חולקו לתוך כל באר של צלחת 96 באר ולאחר מכן בשילוב עם 5 μl של כל תמצית. בקרת המיצוי וההגברה הפנימית MS2 שעברה את פרוטוקול המיצוי המלא נכללה כבקרת המיצוי השלילית במינימום שתי בארות בצלחת qRT-PCR. כדי לקבוע זיהום פוטנציאלי בתהליך qRT-PCR, 5 μl nfH2O נכלל כבקרת ה-qRT-PCR השלילית. לאחר מכן, 5 μl של פלסמיד תבנית SARS-CoV-2 מתווסף נכלל בבאר בודדת כביקורת qRT-PCR חיובית. לאחר הוספת 5 μl מכל דגימה לבאר המיועדת לה, הצלחת נאטמה עם אטם פלסטיק שקוף אופטית. הצלחת עברה צנטריפוגה למשך דקה אחת ב-1×g (1,000 סל"ד) ב-4 מעלות צלזיוס ולאחר מכן הוכנס למכונת qRT-PCR (QuantStudio, Thermo Fisher Scientific) והריצה עברה פרמטרים. נרכשו אותות עבור פלואורסאין (FAM) ו-carboxyrhodamine (ROX). נעשה שימוש ב-ROX כדי לזהות את הבקרה הפנימית של MS2 ופלואורסצין שימש לזיהוי SARS-CoV-2 RNA. הבדיקה בוצעה למשך 2 דקות ב-25 מעלות צלזיוס, 15 דקות ב-50 מעלות צלזיוס (עבור התעתיק ההפוך), 2 דקות ב-90 מעלות צלזיוס, לפני 45 מחזורים של 95 מעלות צלזיוס למשך 3 שניות ולאחר מכן 60 מעלות צלזיוס למשך 30 שניות. . התוצאות נקבעו על ידי אישור של בקרות חיוביות נכונות (הגברה של הפלסמיד), בקרות מיצוי והגברה של כל הדגימות (ערוץ ROX), ללא הגברה בבקרים השליליים וערכים ממוצעים עקביים של בקרות. חיוביות של SARS-CoV-2 אושרה על ידי הגברה בתעלת הפלואורסצאין עם עקומה סיגמואידית מתאימה עם ערך CT של ≤36. ערכי ה-CT של בדיקה MS2 ו-MGB 3 נשמרו כדי לעקוב אחר האיכות והשחזור של הבדיקה44.
חיתוך RNase H ניתן לתכנות עבור nanobait
עבור חישה ננופורית, נעשה שימוש נוסף בבקרות SARS-CoV-2 RNA, תמציות חומצות גרעין (דגימות מטופלים) או RNA ויראלי MS2 לזיהוי עם ננובייט. ראשית, ערבבנו אוליגו מדריך עם המדגם וחיממנו אותה ל-70 מעלות צלזיוס למשך 5 דקות. RNase H (5,000 יחידות למ"ל; NEB) הוסף, ערבב וחומם במשך 20 דקות ב-37 מעלות צלזיוס כדי לאפשר לאנזים לחתוך RNA ב-DNA: היברידית RNA שמשחררת ביעילות את RNA המטרה. RNase H הושבת תרמית על ידי דגירה ב-65 מעלות צלזיוס למשך 10 דקות. אוליגו מדריך אומתו כך שלא יוצרים מבנים תוך-מולקולריים, הומו- או הטרודמרים באמצעות תוכנת NUPACK45. עבור המדידה עם היעד הנעדר, נעשה שימוש באותו פרוטוקול כולל אוליגו מדריך. מדידות הבקרה לא מציגות תזוזה, ומכאן, אנו יכולים להוציא כל קשר צולב מהותי מאוליגוס מדריך.
מאפייני רצף מטרה ויראלי עבור ננובייט
אורך המטרה, אורך אחיזת הרגליים ותכולת ה-GC נבחרו כדי להבטיח הכלאה מיטבית21. עבור עיצובי ה-DNA nanobait, רצפי המטרה נבחרו להיות באזורים המשומרים של גנום ויראלי והיו להם 40-60% תוכן GC כדי ליצור דופלקס יציב של 20 bp. אורך אחיזת הרגליים נבחר להיות באורך של 6 nt ובעל תכולת GC של 40-60%. בדקנו את כל הרצפים עבור אינטראקציות תוך-מולקולריות פוטנציאליות לא רצויות או הומודימרים באמצעות תוכנת NUPACK (יישום אינטרנט 2020)45. לאחר מכן, ביצענו בדיקת תגובתיות צולבת בין מספר אתרים המועסקים בכל ניסוי45.
הכנת פרח DNA עבור nanobait
עיצבנו פרח DNA כתווית נוספת לזיהוי SARS-CoV-2 RNA מדגימות המטופלים. שלושה פרחי DNA ספציפיים לכל יעד SARS-CoV-2 (צמתים שבעה כיוונים, 7WJa, 7WJb ו-7WJc) הוכנו בנפרד. אם לוקחים 7WJc כדוגמה, גדיל DNA של 4 מיקרומטר J1, J2, J3 ו-J4c (טבלה משלימה 1) ערבבו יחד במאגר TM (10 מ"מ Tris-HCl, 10 מ"מ MgCl2, pH 8.0) ומחומם ל-90 מעלות צלזיוס למשך 5 דקות, ואז מקורר ל-65 מעלות צלזיוס למשך 15 דקות, 45 מעלות צלזיוס למשך 15 דקות, 37 מעלות צלזיוס למשך 20 דקות, 25 מעלות צלזיוס למשך 20 דקות ולבסוף ל-4 מעלות C למשך 20 דקות. Strand J4c הוחלף על ידי J4b כדי להכין 7WJb. עבור 7WJa, כדי להימנע מקיפול עצמי באתר 43 על nanobait, J1, J2, J3 J4a ו-C43 ערבבו יחד לפני החישול.
הרכבה עצמית של DNA nanobait
ה-DNA nanobait הורכב על ידי ערבוב M13 DNA חד-גדילי ליניארי (M13mp18, 7,249 nt, Guild Biosciences, 100 nM) עם אוליגונוקלאוטידים משלימים קצרים12 (שחלקם הכיל מבני התייחסות ולכידת גדילים) ועל ידי הוספת גדילים משלימים חלקית שהיו 3′-biotinylated לתגובת עקירת גדיל בתיווך אחיזת רגליים. ה-DNA הלינאארי של M13 (באורך 7,228 נ"ט) הושלמה על ידי אוליגונוקלאוטידים, ובכך יצר ננו-בייט כפול גדילי עם חצי קצה של ארבעה דיאוקסיתימידין שמונעים מולטימריזציה12. התערובת הכילה 20 ננומטר של M13 DNA ליניארי, 60 ננומטר של אוליגונוקלאוטידים (פי שלושה עודף ל-M13 DNA), גדילים 3'-biotinylated בריכוז של 180 nM, 10 מM MgCl2 ו-1× TE (10 מ"מ Tris-HCl, 1 מ"מ EDTA, pH 8.0). הוא ערבב על ידי פיפטינג וסובב לפני חימום ל-70 מעלות צלזיוס למשך 30 שניות והתקרר במשך 45 דקות לטמפרטורת הסביבה. עודפי אוליגונוקלאוטידים הוסרו באמצעות מסננים צנטריפוגליים של Amicon Ultra 0.5 מ"ל עם חתך של 100 kDa עם מאגר כביסה (10.0 mM Tris-HCl pH 8.0, 0.5 mM MgCl2). אם פרחי DNA הועסקו כתווית, הגדילים המשלימים חלקית הנושאים אותו הודגרו ב-10 mM MgCl2 במשך 2 שעות בטמפרטורת הסביבה, ולאחר מכן בוצע סינון Amicon כמתואר לעיל. האסימטריה של עיצוב הננובייט מאפשרת זיהוי חד משמעי של אתרי הקישור. הננובייט אוחסן עד לשימוש לניסויים נוספים מתחת ל-4-10 מעלות צלזיוס ב-0.5 mM MgCl2, 10.0 mM Tris-HCl, pH 8.0. עיצוב nanobait נבדק על ידי קריאת nanopore לפני כל מדידה.
קריאת Nanopore של DNA Nanobait
הננובייט היה מעורבב עם דגימה (תמצית חומצת גרעין או מטרות ויראליות מטוהרות בעודף פי עשרה) ב-10 mM MgCl2 ו-100 מ"מ NaCl. התערובת (5 μl) הודגרה בטמפרטורת החדר (~10 דקות) עד להכנה למדידת ננו-פוריים. ההבדל בהרכב רצף המטרה והמאפיינים הפיזיים שלו עלול להוביל לשונות בהכלאה ומכאן ליעילות העקירה של אתרי חישה21. השתמשנו ב-htRNA (100 ng μl-1; Invitrogen) כרקע היכן שצוין, כדי להראות שאין אותות לא ספציפיים המושרים על ידי RNAs ילידים אנושיים. למדידת ננו-פוריים, הדגימה דוללה ל-<0.5 nM nanobait (עבור מטרות ויראליות מטוהרות) או ש-4.7 μl של דגימת מטופל חתוך RNase-H מעורבב עם 0.3 μl של streptavidin חד ערכי (SAe1D3)18 (1 μM), 5 μl של LiCl (4.0 M) ו-5.0 μl של LiCl (8.0 M). יצרנו ננו-נקבוביות של 14 ± 3 ננומטר (ממוצע ± סטיית תקן)12 שימוש בנימי זכוכית קוורץ בקוטר חיצוני של 0.5 מ"מ ובקוטר פנימי של 0.2 מ"מ (Sutter Instrument) על ידי חולץ בסיוע לייזר P-2000 (Sutter Instrument). התערובת הועברה לפיפטה בשבב polydimethylsiloxane nanopore, וכל המדידות בוצעו במתח קבוע של 600 mV. פרטי מדידת ננופור מוצגים בטבלה משלימה 30.
ניתוח נתונים ננופוריים בזמן אמת
ניתוח נתונים של Nanopore מוסבר בפירוט בסעיף המשלים 14. בקצרה, אירועי nanobait סוננו מתוך עקבות זרם יוני גולמי ולאחר מכן נקבע אזור הזיהוי, וחולץ מידע על נוכחות הספייק בכל אתר ספציפי. יעילות העקירה המתוכננת חושבה כיעילות תזוזה עבור מדידה שהופחתה לבקרה ללא יעד עבור כל אתר (50 אירועי ננו-בייט עבור כל אחת משלוש ההקלטות הננו-פוריות), אלא אם צוין אחרת:
$$begin{array}{l}{mathrm{Displacement}},{mathrm{efficiency}} =frac{1}{3}mathop {sum}limits_{n = 1}^3 נותרו{ {1 -frac{1 }{{50}}mathop {sum}limits_{n = 1}^{50} {left[ {fleft(n right) = left( {frac{{1,,mathrm{peak}}}{{0,, {mathrm{no}},{mathrm{peak}}}}} מימין]_{{{{mathrm{target}}}}}}} ימין}\ – frac{1}{3}mathop {sum }limits_{n = 1}^3 {left{ {1 – frac{1}{{50}}mathop {sum }limits_{n = 1}^{50} left[ {fleft(n right) = left( { frac{{1,,mathrm{peak}}}{{0,,mathrm{no}},{mathrm{peak}}}} ימין)} ימין]_{{{mathrm{no}}}},{ {{mathrm{target}}}}}} מימין}} סוף{מערך}.$$
אימתנו שהרשת העצבית הקונבולוציונית QuipuNet27 היה מסוגל לנתח בזמן אמת נתונים ננופוריים בעקבות ההליך המתואר. בעבר, הוכחנו שעם כעשרה אירועים, אנו מגיעים ל-99% אמון בזיהוי חיובי של מבני ה-DNA המעוצבים שלנו46.
הדמיית AFM
הדמיית AFM (Nanosurf Mobile S) של nanobaits בוצעה באוויר במצב ללא מגע. מבני nanobait היו מדוללים ל-1 ng μl-1 ב-1 mM MgCl2 ו- 10 μl נוספו לנציץ שנחתך טרי, הודגרה למשך דקה אחת, נשטף עם מי Milli-Q מסוננים ולאחר מכן ייבוש בנשיפה עם חנקן. לפני הסריקה, לוחית הנציץ הוצמדה לשלב המדגם של AFM באמצעות סרט דביק דו צדדי. הדמיה וניתוח תמונה בוצעו באמצעות Gwyddion (גרסה 1).
ניתוח סטטיסטי
עבור כל המדידות חושבו רווחי סמך של 99.9% ליעילות עקירה. מובהקות סטטיסטית בין שני אתרים ללא ועם המטרה נבדקה באמצעות סטודנט דו-צדדי t- מבחן.
סיכום הדיווח
מידע נוסף על עיצוב המחקר זמין ב סיכום דיווח תיקי טבע מקושר למאמר זה.
- הפצת תוכן ויחסי ציבור מופעל על ידי SEO. קבל הגברה היום.
- Platoblockchain. Web3 Metaverse Intelligence. ידע מוגבר. גישה כאן.
- מקור: https://www.nature.com/articles/s41565-022-01287-x
- 000
- 1
- 10
- 100
- 11
- 110
- 2014
- 2016
- 2017
- 2018
- 2020
- 2021
- 7
- 70
- 9
- a
- מֵעַל
- נעדר
- אקדמי
- נרכש
- הוסיף
- לאחר
- AIR
- תעשיות
- מאפשר
- אמביינט
- אנליזה
- עוגן
- ו
- אחר
- בקשה
- גישה
- מתאים
- סביב
- מאמר
- התאסף
- זמין
- בחזרה
- רקע
- לפני
- פעמון
- בֵּין
- כריכה
- לפוצץ
- BP
- בקצרה
- חיץ
- מחושב
- מסוגל
- ללכוד
- לשאת
- מוסמך
- ערוץ
- מאפיינים
- לבדוק
- חן
- שבב
- ברור
- קליני
- אוסף
- משולב
- משלימה
- ריכוז
- אמון
- מְאוּשָׁר
- עִקבִי
- קבוע
- תוכן
- לִשְׁלוֹט
- בקרות
- רשת עצבית convolutional
- יוצרים
- נוֹכְחִי
- זונה
- חותך
- גזירה
- מחזורי
- נתונים
- ניתוח נתונים
- מופגן
- מְתוּאָר
- עיצוב
- מעוצב
- עיצובים
- פרט
- פרטים
- איתור
- לקבוע
- נחוש
- סטייה
- הבדל
- באופן דיגיטלי
- ישירות
- הפצה
- ה-DNA
- מטה
- כל אחד
- יעילות
- יעילות
- יְעִילוּת
- הנדסה
- לְהַבטִיחַ
- שגיאה
- Ether (ETH)
- אֲפִילוּ
- אירועים
- דוגמה
- מוסבר
- תמצית
- הוֹצָאָה
- תמציות
- מסננים
- בסופו של דבר
- ראשון
- פרח
- בעקבות
- הבא
- טופס
- קדימה
- החל מ-
- מלא
- נוסף
- הגנום
- זכוכית
- מדריך
- מאוד
- HTTPS
- בן אנוש
- היברידי
- הזדהות
- תמונה
- הדמיה
- הפעלה
- in
- כלול
- כולל
- דגירה
- דגירה
- מידע
- מכשיר
- יחסי גומלין
- פנימי
- יונית
- IT
- KDA
- תווית
- עוֹפֶרֶת
- אורך
- קשר
- צמוד
- ארוך
- לונה
- מכונה
- אב
- חוֹמֶר
- מידות
- נָצִיץ
- יכול
- מינימום
- מעורב
- ערבוב
- תַעֲרוֹבֶת
- ML
- סלולרי
- מצב
- MOL
- מספר
- ננו
- ננופור
- יליד
- טבע
- שלילי
- רשת
- עצביים
- רשת עצבית
- לפתוח
- אופטימלי
- אַחֶרֶת
- זיווג
- חולה
- חולים
- ביצועים
- גופני
- תמונה
- פלסטי
- אפלטון
- מודיעין אפלטון
- אפלטון נתונים
- תיק עבודות
- חיובי
- חִיוּבִיוּת
- פוטנציאל
- להכין
- מוּכָן
- נוכחות
- למנוע
- קוֹדֶם
- תחל
- בדיקה
- תהליך
- נכסים
- חֶלְבּוֹן
- פרוטוקול
- איכות
- חי
- לְהַגִיעַ
- תגובה
- זמן אמת
- באזור
- אזורים
- עיתונות
- נותר
- הוסר
- דווח
- מחקר
- תוצאות
- להפוך
- רנ"א
- רוש
- חֶדֶר
- rt
- כללי
- הפעלה
- בְּטִיחוּת
- אותו
- סארס-CoV-2
- סריקה
- סעיף
- נבחר
- רצף
- קצר
- לְהַצִיג
- הראה
- אותות
- משמעות
- בו זמנית
- יחיד
- אתר
- אתרים
- קטן
- תוכנה
- כמה
- ספציפי
- הסתובב
- יציב
- התמחות
- לעמוד
- תֶקֶן
- אמור
- סטטיסטי
- צעדים
- מאוחסן
- קווצות
- כתוצאה מכך
- ניכר
- מערכות
- שולחן
- נטילת
- יעד
- מטרות
- תבנית
- עשר
- השמיים
- בכך
- שְׁלוֹשָׁה
- פִּי
- TM
- ל
- יַחַד
- סה"כ
- לעקוב
- Ultra
- תחת
- יחידות
- אוניברסלי
- תוקף
- ערך
- ערכים
- מְאוּמָת
- גרסה
- באמצעות
- וירוסים
- ראיה
- מתח
- W
- מים
- אינטרנט
- אפליקציית רשת
- ולס
- אשר
- לְלֹא
- wu
- זפירנט