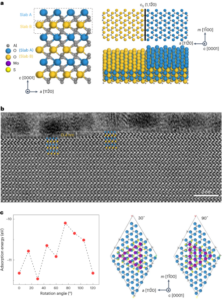
Sintesi dei nanobot
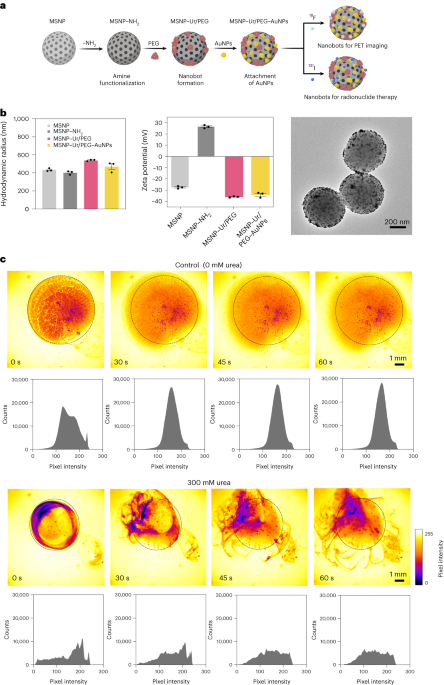
I nanobot sono stati preparati come descritto in precedenza33. In breve, gli MSNP sono stati sintetizzati utilizzando un metodo Stöber modificato41, facendo reagire trietanolammina (35 g), acqua ultrapura (20 ml) e bromuro di esadeciltrimetilammonio (CTAB; 570 mg) a 95 °C per 30 min sotto agitazione. Successivamente è stato aggiunto goccia a goccia tetraetil ortosilicato (1.5 ml); la miscela è stata lasciata reagire per 2 h a 95 °C e gli MSNP risultanti raccolti mediante centrifugazione e lavati in etanolo (tre volte, 2,500g, 5 minuti). Per rimuovere il modello CTAB, gli MSNP sono stati posti a riflusso in metanolo acido (1.8 ml di HCl, 30 ml di metanolo) per 24 ore. Quindi, gli MSNP sono stati raccolti mediante centrifugazione e lavati tre volte in etanolo (2,500g, 5 min) prima di incorporare la modificazione dell'ammina aggiungendo APTES (6 μl per mg di MSNP) agli MSNP (1 mg ml-1) in una soluzione etanolica al 70% a 70 °C, agitando vigorosamente per 1 h. MSNP-NH2 sono stati raccolti e lavati tre volte in etanolo e tre volte in acqua mediante centrifugazione (tre volte, 1,150g, 5 minuti). MSNP-NH2 sono stati risospesi in PBS ad una concentrazione di 1 mg ml-1 e un volume totale di 900 µl e attivato con glutaraldeide (100 µl) per 2.5 µl a temperatura ambiente. Gli MSNP-NH attivati2 sono stati raccolti e lavati in PBS tre volte mediante centrifugazione (1,150g, 5 min), risospesi in una soluzione di ureasi (3 mg ml-1) e PEG eterobifunzionale (1 μg PEG per mg di 5 kDa HS-MSNPs-NH2) in PBS e ha reagito per 24 ore a temperatura ambiente. I nanobot risultanti sono stati quindi raccolti e lavati tre volte in PBS mediante centrifugazione (1,150g, 5 min) prima di risospenderli in una dispersione di AuNP, preparata come precedentemente descritto51, lasciando reagire per 10 min, e lavando accuratamente mediante centrifugazione (tre volte, 1,150g, 5 minuti).
Distribuzione dimensionale idrodinamica e carica superficiale degli MSNP, MSNP-NH2, i nanobot e i nanobot decorati con AuNP sono stati determinati utilizzando rispettivamente un sistema di diffusione dinamica della luce Wyatt Mobius e un Malvern Zetasizer. In tutti i casi, la concentrazione era di 20 μg ml-1 e tempo di acquisizione 5 s, utilizzando tre esecuzioni per esperimento. Sono state eseguite tre misurazioni per tipo di particella.
Sintesi di MSNP FITC
Una miscela di FITC (2 mg), etanolo (5 ml) e APTES (400 μl) è stata preparata e agitata per 30 min. Quindi, è stato seguito il protocollo precedentemente descritto per la sintesi di MSNP, tranne per il fatto che abbiamo aggiunto goccia a goccia tetraetil ortosilicato (1.25 µl) in combinazione con la miscela FITC-APTES (250 µl). Le fasi di funzionalizzazione per ottenere nanobot marcati FITC erano le stesse sopra menzionate.
Sintesi di AuNP
Gli AuNP sono stati sintetizzati utilizzando un metodo riportato33. In breve, tutti i materiali sono stati puliti utilizzando acqua regia appena preparata, risciacquati accuratamente con acqua e asciugati all'aria. Successivamente, un AuCl da 1 mM4 la soluzione è stata riscaldata fino al punto di ebollizione sotto agitazione in un pallone a fondo tondo integrato in un sistema a riflusso. Successivamente, sono stati aggiunti 10 ml di soluzione di citrato di sodio (30.8 mM) e la soluzione è stata fatta bollire per 20 minuti, ottenendo un colore rosso. La soluzione è stata quindi lasciata raffreddare a temperatura ambiente agitando per 1 ora. Gli AuNP risultanti sono stati conservati al buio e la caratterizzazione è stata condotta utilizzando la microscopia elettronica a trasmissione.
Attività enzimatica
Attività enzimatica dei nanobot, 18F-nanobot e 131I-nanobots è stato misurato utilizzando il rosso fenolo. Per fare ciò, 2 μl di nanobot (1 mg ml-1) sono stati aggiunti a una piastra da 96 pozzetti e miscelati con 200 µl di diverse soluzioni di urea (0, 50, 100, 200 µM) in rosso fenolo 1.1 µM. L'assorbanza a 560 nm è stata misurata nel tempo a 37 °C.
Dinamica del movimento dei nanobot attraverso la microscopia ottica
I video ottici dei nanobot sono stati acquisiti utilizzando un microscopio Leica Thunder, abbinato a una fotocamera CCD ad alta velocità Hamamatsu e un obiettivo × 1.25. Per questo, i nanobot sono stati centrifugati e risospesi in 50 µl di PBS (concentrazione finale di 20 mg ml-1). Quindi, una capsula Petri è stata riempita con 3 ml di PBS o con una soluzione 300 mM di urea (in PBS) e osservata al microscopio. Una goccia da 5 μl con nanobot (20 mg ml-1) è stato quindi aggiunto alla capsula Petri piena di liquido e i video sono stati registrati a 25 fotogrammi al secondo. Le distribuzioni dell'intensità dei pixel video nelle ROI sono state analizzate a intervalli di 15 utilizzando il software ImageJ.
Radiomarcatura dei nanobot con [18F]F-PyTFP
Sintesi di [18F]F-PyTFP
[18F]F-PyTFP è stato sintetizzato in un modulo Neptis xSeed (Optimized Radiochemical Applications), seguendo un metodo precedentemente riportato33.
Sintesi di 18Nanobot etichettati F
I nanobot erano etichettati con [18F]F-PyTFP, sulla base di una procedura precedentemente stabilita con piccole modifiche33. In breve, 200 μl di soluzione di nanobot (1 mg ml-1) è stato centrifugato (10 min, 13,853g), risospesi in 10 µl di PBS (1 µM, pH 8) e incubati con 4 µl di [18F]F-PyTFP in acetonitrile (circa 37 MBq) per 35 min a temperatura ambiente. Dopo l'incubazione, la miscela di reazione è stata diluita con acqua (200 µl) e purificata mediante centrifugazione (5 min, 13,853g). Il pellet risultante è stato quindi risciacquato tre volte con acqua prima di essere misurato in un calibratore di dose (CPCRC-25R, Capintec). La resa radiochimica è stata calcolata come il rapporto tra la quantità di radioattività presente nei nanobot dopo il lavaggio e la quantità di radioattività iniziale. La purezza radiochimica dopo la purificazione era ≥99%, come determinato mediante radiocromatografia su strato sottile (radio-TLC) utilizzando carta per cromatografia iTLC-SG (Agilent Technologies) e diclorometano e metanolo (2:1) rispettivamente come fase stazionaria e mobile. Le piastre TLC sono state analizzate utilizzando un lettore TLC (MiniGITA, Raytest).
Stabilità di 18F-nanobot
La stabilità di 18I nanobot marcati con F sono stati determinati utilizzando i seguenti mezzi: (1) urea 300 mM, (2) acqua e (3) urina di animali portatori di tumore. 18I nanobot marcati con F (10 µl) sono stati incubati con la soluzione corrispondente (100 µl) per 1 ora a temperatura ambiente. Quindi, i nanobot e il supernatante sono stati separati mediante centrifugazione e raccolti, e la radioattività misurata in un calibratore di dose (CPCRC-25R).
Radiomarcatura dei nanobot con 131I
La radioiodurazione dei nanobot con ureasi è stata eseguita incubando i nanobot con [131Soluzione I]NaI (925 MBq ml-1; GE HealthCare). In breve, 400 μl di soluzione di nanobot di ureasi (1 mg ml-1) è stato centrifugato (13,853g, 5 min), risospesi in 100 μl di PBS (10 mM, pH 7.4) e incubati con 25 μl o 185 μl di soluzione iniettabile [131I]NaI (rispettivamente circa 42.55 o 277.5 MBq) per 30 min, a seconda dell'attività finale desiderata. Dopo l'incubazione, la miscela di reazione è stata purificata mediante centrifugazione (13,853g, 5 minuti). Il precipitato risultante è stato lavato tre volte con acqua (100 µl). La radioattività nel surnatante e nel precipitato è stata determinata utilizzando un calibratore di dose (CPCRC-25R) ed entrambe le frazioni sono state analizzate mediante radio-TLC, come per 18F-nanobot.
Sviluppo del modello animale
I topi sono stati mantenuti e gestiti in conformità con la Direttiva 2010/63/UE del Consiglio Europeo e le linee guida interne. Tutte le procedure sperimentali sono state approvate dal comitato etico CIC biomaGUNE e dalle autorità locali (Diputación Foral de Guipuzcoa, PRO-AE-SS-276). L'analisi delle immagini (sia PET che MRI) è stata effettuata in cieco rispetto alla distribuzione dei gruppi degli animali.
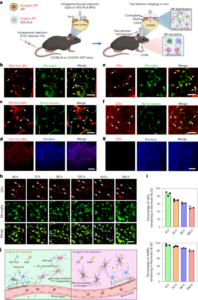
Il modello murino ortotopico di cancro della vescica è stato generato mediante somministrazione intravescicale di cellule MB49 (linea cellulare di carcinoma murino della vescica) a topi femmine C57BL/6JRj (8 settimane, Janvier). Per gli esperimenti volti a determinare l'accumulo di tumore (quattro gruppi; dettagli di seguito), sono stati inoculati sei animali per gruppo, come determinato utilizzando l'analisi di precisione, con le seguenti ipotesi: precisione richiesta, 20%; s.d. previsto, ±20%; fiducia, 95%; perdita di animali, 20%. Per gli esperimenti sull'efficacia terapeutica (sei gruppi; dettagli di seguito), sono stati inclusi dieci animali per gruppo, come calcolato utilizzando un metodo di Student a una coda t-test, differenza tra due medie indipendenti, con le seguenti ipotesi: ipotesi nulla, il trattamento non influenza la crescita del tumore; α, 0.05; 1 − β, 0.95; s.d., ±50%; differenze attese tra i gruppi, 50%; perdita di animali, 20%. Poiché l'esperimento è stato condotto in due lotti per ragioni operative, un gruppo di controllo è stato incluso in entrambi i lotti (Tabella 2), e poi tutti gli animali sono stati raggruppati. Per la stabilizzazione del tumore, i topi sono stati anestetizzati mediante inalazione di isoflurano al 3% in O puro2 e mantenuto dall'1.0–1.5% di isoflurano in O 100%.2. Quindi, la vescica è stata svuotata e sono state indotte lesioni chimiche sull'urotelio mediante instillazione intravescicale di 50 µl di poli-l-lisina (Sigma-Aldrich) attraverso un catetere di calibro 24 per 15 min. Successivamente, la vescica è stata nuovamente svuotata e le cellule MB49 (105 cellule) in DMEM ad alto contenuto di glucosio (100 µl) sono stati instillati per 1 ora prima di rimuovere il catetere e svuotare la vescica tramite massaggio addominale. Durante gli esperimenti, i topi sono stati monitorati e pesati per il monitoraggio della salute e del benessere. È stato applicato un endpoint umano se la perdita di peso superava il 20% o sulla base dei sintomi clinici, secondo i criteri del veterinario responsabile.
Monitoraggio delle dimensioni del tumore
Gli studi MRI sono stati condotti 7 e 14 giorni dopo l'induzione del tumore, utilizzando uno scanner Bruker BioSpec USR 7/70 da 30 T (Bruker BioSpin) dotato di un inserto con gradiente BGA-12S di 440 mT m-1 e un risonatore QSN 112/086 (T12053V3) per radiofrequenza14 trasmissione e una bobina di superficie del cervello di ratto (T11205V3) per la ricezione RF (entrambe funzionanti a 300 MHz). Gli animali sono stati anestetizzati con isoflurano (4% per l'induzione e 1.5% per il mantenimento in una soluzione al 50% di O2/50%N2 miscela) e posizionati su una culla compatibile con la RM. La temperatura corporea e la frequenza respiratoria sono state monitorate continuamente utilizzando un dispositivo di monitoraggio compatibile con la RM (modello 1030 SA, Small Animal Instruments), interfacciato con un sistema di riscaldamento dell'aria per piccoli roditori per mantenere la temperatura corporea. Dopo aver acquisito le immagini di riferimento, è stata utilizzata una sequenza di imaging pesata in diffusione basata su spin-echo per visualizzare i tumori, utilizzando i seguenti parametri: tempo di eco (TE) = 22.3 ms, tempo di ripetizione (TR) = 2,500 ms, n = 2 medie, un'immagine A0 (immagine basale con b = 0 secondi mm-2) e un'immagine DW acquisita utilizzando gradienti di diffusione nella direzione (1, 0, 0) con una durata del gradiente δ = 4.5 ms e una separazione gradiente Δ = 10.6 ms, dare b = 650 secondi mm-2, un 16 × 16 mm2 campo visivo, dimensione della matrice dell'immagine di 160 × 160 punti, 20 fette consecutive di 0.5 mm di spessore (nessun intervallo, acquisite in modalità interleaved) e una larghezza di banda di 192.9 Hz per pixel. Per visualizzare i tumori, le immagini sono state postelaborate con il software ImageJ, dividendo le immagini acquisite con un gradiente di diffusione (b = 650 secondi mm-2) da quelli acquisiti senza (b = 0 secondi mm-2) e applicando un filtro gaussiano 3D (σx = σy = σz = 0.7) al risultato. I tumori sono stati delineati manualmente per determinarne il volume.
Biodistribuzione in vivo
Il giorno 15 dopo l'induzione del tumore, i topi sono stati randomizzati in quattro gruppi per ottenere distribuzioni medie omogenee del volume del tumore tra i gruppi. Le scansioni PET-CT (scanner MOLECUBE β e X-CUBE) sono state acquisite 3 h dopo la somministrazione intravescicale di 100 μl di 18F-BSA (gruppi 1 e 2) o 18Nanobot F-ureasi (gruppi 3 e 4) ad una concentrazione di 200 μg ml-1, utilizzando acqua (gruppi 1 e 3) o 300 mM di urea in acqua (gruppi 2 e 4) come veicolo (Tabella 1). Per l'acquisizione delle immagini, gli animali sono stati indotti con anestesia (isoflurano al 5% in ossigeno puro) e posti in posizione supina prima di massaggiare la regione addominale per l'evacuazione della vescica. Subito dopo, il corrispondente 18Nanobot etichettati F (18F-BSA/18F-ureasi in acqua/urea) sono stati instillati nella vescica attraverso un catetere di calibro 24 e incubati per 1 ora, prima di rimuovere il catetere, svuotare la vescica e lasciare che i topi si riprendessero dall'anestesia. A t = 3 h dopo la somministrazione, gli animali sono stati ri-anestetizzati e sono state acquisite immagini PET statiche di tutto il corpo per 10 min, seguite da scansioni TC. Le immagini PET sono state ricostruite utilizzando l'algoritmo di ricostruzione di massimizzazione delle aspettative del sottoinsieme ordinato 3D con correzioni casuali, di dispersione e di attenuazione. Le immagini PET-CT dello stesso topo sono state co-registrate e analizzate utilizzando lo strumento di elaborazione delle immagini PMOD. I grafici della concentrazione della radioattività in funzione del tempo sono stati ottenuti creando un volume di interesse sulla regione superiore della vescica utilizzando uno strumento di contorno 3D e misurando l'attività (decadimento corretto) in kilobecquerel per organo. I risultati sono stati corretti applicando un fattore di calibrazione e quindi normalizzati dal volume del tumore derivato dalla risonanza magnetica.
Studi ex vivo
Analisi istopatologiche
Dopo aver completato tutte le immagini, le vesciche selezionate (n = 3 per gruppo) da animali portatori di tumore e sani sono stati rimossi in condizioni asettiche e immediatamente fissati in formaldeide al 4%. Quindi, le vesciche sono state incluse in paraffina prima di prelevare sezioni da 2–3 µm per la colorazione con ematossilina-eosina. Sono state ottenute immagini rappresentative di tutte le condizioni per l'esame istopatologico.
Analisi ICP-MS
Le misurazioni sono state eseguite su un Thermo iCAP Q ICP-MS (Thermo Fisher Scientific) accoppiato con un autocampionatore ASX-560 (CETAC Tech). Dopo aver completato tutte le immagini, gli animali sono stati uccisi e le vesciche selezionate (n = 2 per gruppo; quattro gruppi) raccolti e digeriti in 1 ml di HNO3:HCl (miscela 4:1). La dispersione è stata fatta bollire fino alla completa dissoluzione degli organi. Quindi, la soluzione è stata raffreddata a temperatura ambiente e analizzata utilizzando ICP-MS per determinare la concentrazione di Au in ciascun campione, trasformando i risultati in percentuali di dose iniettata per grammo di tessuto (%ID g-1).
Imaging immunoistochimico e microscopia confocale
Per le analisi immunoistochimiche, gli animali portatori di tumore hanno ricevuto nanobot marcati con FITC in acqua o urea 300 mM (n = 4 per gruppo), come descritto sopra, per gli studi PET-CT. Inoltre, gli animali portatori di tumore senza nanobot sono serviti come gruppo di controllo (n = 2). In tutti i casi, le vesciche sono state raccolte, congelate e tagliate in sezioni da 10 µm che sono state immediatamente fissate in formaldeide al 10% per 15 µm, lavate con PBS 10 µM e quindi incubate in NH 50 µM.4CI in PBS per 5 min prima di risciacquare nuovamente con PBS. La permeabilizzazione è stata eseguita con metanolo:acetone (1:1) per 5 minuti a temperatura ambiente e Triton allo 0.1% in PBS per 5 minuti. Dopo il lavaggio con PBS, i campioni sono stati saturati con una soluzione di Tween al 5% BSA–0.5% in PBS per 15 min a temperatura ambiente e incubati per 1 ora a temperatura ambiente con anti-FITC di topo (1:100, Abcam) in BSA al 5% –0.5% Interpolazione. Le sezioni sono state lavate tre volte con PBS 10 mM per 5 minuti e incubate per 30 minuti a temperatura ambiente con l'anticorpo secondario Alex Fluor 647 IgG anti-topo d'asino (Molecular Probes, Life Technologies, 1:1,000) in 5% BSA–0.5% Tween in PBS, lavati nuovamente in PBS (3 × 5 min) e montati con un kit antisbiadimento ProLong con 4,6-diamidino-2-fenilindolo (DAPI; Molecular Probes, Life Technologies). Le immagini sono state acquisite con un microscopio confocale Leica STELLARIS 5 (UPV/EHU Scientific Park) con impostazioni identiche per tutte le sezioni: ingrandimento ×10 con imaging e cucitura di piastrelle (tipicamente campo visivo 4 × 5). La linea laser e le finestre di rilevamento erano 405 nm e 440–503 nm per DAPI, 489 nm e 494–602 nm per il laser bianco FITC e 653 nm e 660–836 nm per il laser bianco Alexa647.
Cancellazione ottica
Dopo la perfusione con paraformaldeide al 4% e PBS, i campioni vescicali sono stati rimossi e ulteriormente fissati in paraformaldeide al 4% durante la notte a 4 °C, quindi incorporati in una siringa da 5 ml con agarosio allo 0.8% a basso punto di fusione per formare un blocco cilindrico e consentire una facile montaggio nella cuvetta di quarzo. L'intero blocco è stato progressivamente disidratato utilizzando metanolo:H2O a 4 °C (30%:70% per 1 h, 50%:50% per 1 h, 70%:30% per 1 h, 100%:0% per 1 h, quindi metanolo al 100% durante la notte e di nuovo per 4 h) e infine immerso in alcol benzilico-benzil benzoato (BABB) come soluzione di corrispondenza dell'indice di rifrazione per l'imaging. Per i confronti in vitro dei nanobot FITC verdi con particelle rosse commerciali, abbiamo utilizzato nanoparticelle di silice fluorescente rossa DiagNano (Creative Diagnostics), diametro 1 μm, resistenti alla pulizia BABB.
Autofluorescenza e imaging sLS polarizzato
L'imaging del foglio di luce è stato eseguito su MacroSPIM, un sistema personalizzato per l'imaging di interi organi sviluppato presso l'IRB di Barcellona44,45. In breve, i campioni vengono incorporati in un blocco di agarosio, purificati insieme al campione e sottoposti a imaging all'interno di una cuvetta di quarzo. L'imaging ad autofluorescenza utilizzava laser a 488, 561 o 638 nm che fornivano illuminazione attraverso una lente cilindrica doppietta acromatica da 50 mm (ACY254-050-A, Thorlabs). Per ridurre gli artefatti a strisce, il foglio luminoso viene ruotato con uno scanner risonante SC-10 (EOPC) lungo un telescopio 4f con lenti doppiette acromatiche G322288322 da 100 mm (QI Optic Photonics). L'autofluorescenza dei tessuti viene raccolta attraverso filtri di fluorescenza a banda o passa lungo e registrata con una fotocamera ORCA Flash v2 (Hamamatsu Photonics). L'imaging è stato eseguito a ×9.6 con uno zoom ×8, un obiettivo ×2 e un obiettivo a tubo ×0.6. Il foglio luminoso è stato appiattito attraverso il campo visivo, ottenendo 5–6 µm di risoluzione assiale. L'imaging 3D è stato eseguito in incrementi di 2.5 µm. L'imaging dell'intera vescica è stato eseguito in 2 × 3 o 3 × 4 XY tessere, a seconda delle dimensioni dell'organo.
L'imaging sLS è stato ottenuto rimuovendo il filtro a fluorescenza o utilizzando qualsiasi filtro che trasmette il laser. La rotazione del foglio leggero ha ridotto il rumore delle macchie laser, determinando una media temporale della coerenza laser, come mostrato in precedenza52. L'orientamento della polarizzazione lineare del foglio di luce nell'illuminazione è stato controllato ruotando una piastra a semionda (AHWP05M-600, Thorlabs) prima dello scanner pivot. Il segnale rilevato è stato selezionato nella polarizzazione utilizzando un polarizzatore lineare rotante (LPVISC100, Thorlabs) prima della ruota portafiltri durante il rilevamento, introducendo una perdita di intensità > 50% nel rilevamento della fluorescenza. Mentre la distribuzione del segnale sLS in generale cambia con l’orientamento del polarizzatore, il segnale di autofluorescenza del tessuto rimane inalterato dalla rotazione del polarizzatore. sLS produce una risoluzione spaziale di 2.4 ± 0.3 μm in BABB, che è paragonabile alla risoluzione nell'imaging con foglio di luce in fluorescenza (confermato adattando una funzione gaussiana al XY risposta dell'immagine di una singola particella, Figura supplementare. 8l–m) e vicino alla risoluzione teorica in aria (1.53 μm con apertura numerica (NA) = 0.2 con zoom macro massimo ×8).
Elaborazione delle immagini e analisi 3D
L'elaborazione delle immagini, la segmentazione e l'analisi dei set di dati di fogli leggeri sono state eseguite con ImageJ/Fiji, mentre le Figg. 3 ed 4 sono stati generati con Imaris Viewer 9.9 (https://imaris.oxinst.com/imaris-viewer) e video supplementare 3 è stato generato con Imaris 9 (https://imaris.oxinst.com/) (Bitplane, Oxford Instruments). I set di dati di fogli leggeri piastrellati sono stati uniti con MosaicExplorerJ53. La segmentazione 3D del tessuto vescicale è stata eseguita utilizzando macro ImageJ/Fiji personalizzate per l'annotazione 3D semiautomatica di grandi volumi in modalità virtuale. In breve, un primo script, "Macro1", carica stack di immagini 3D, consente all'utente di annotare le ROI su più piani e interpola automaticamente le ROI per generare ed esportare maschere 3D. Le ROI sono state disegnate ogni 15 piani (ogni 37.5 μm) per facilitare una buona continuità di segmentazione mantenendo le annotazioni al minimo ragionevole. Un secondo script, "Macro2", esegue le operazioni matematiche o booleane, piano per piano senza caricare in memoria l'intero stack, né tra maschere 3D né tra una maschera 3D e i dati originali, salvando il risultato come un nuovo stack. Tutte le maschere sono state generate annotando le immagini di autofluorescenza.
Sia gli strati superficiali del tumore che quelli del tessuto sano (Fig. 3) sono stati delineati utilizzando la bacchetta magica e gli strumenti del lazo delle Fiji sulla cavità vescicale in una maschera. Chiamando questa prima iterazione BC1, le esecuzioni successive di Macro1 dilatano quindi automaticamente questo contorno 3D di una quantità di pixel definita per produrre nuove iterazioni della maschera, BC2, BC3 e così via, con dilatazioni crescenti. Il primo strato contenente sia tumore che tessuto sano, maschera L1, si ottiene sottraendo la maschera BC1 da BC2 e così via, ottenendo L2 e L3 come strati concentrici. Il volume del tumore più vicino alla cavità è stato ottenuto annotando il tumore con strumenti bacchetta e lazo per creare una maschera T1, mentre lo strato 3D di urotelio sano è stato rilevato separatamente nella maschera U1. Sottraendo U1 da L1 si ottiene lo strato superficiale del tumore e così via: L2 − U1, L3 − U1. Viceversa, il primo strato dell'urotelio si ottiene sottraendo T1 da L1. Tutti gli strati in Fig. 3 sono stati definiti avere uno spessore di 33 μm.
La stessa suite di macro e procedure (strumento bacchetta ImageJ, erosione digitale di 500 µm e così via) è stata utilizzata per delineare e segmentare la parte interna del tessuto vescicale e quindi stimare il volume del tessuto interno della vescica (Fig. 4, vedere sopra per i dettagli). Gli istogrammi dell'intensità del segnale sparso sono stati creati nelle Fiji combinando il segnale sparso e la maschera.
RNT utilizzando 131I-nanobot
Tra i giorni 8 e 15 dopo l'impianto del tumore, gli animali sono stati divisi in sei gruppi (gruppi 1-6), cercando di ottenere volumi tumorali medi simili tra i gruppi (Tabella 2). Per gli esperimenti, gli animali sono stati indotti con anestesia (isoflurano al 5% in O2) e posizionati supini prima di svuotare la vescica massaggiando la regione addominale. Subito dopo, 100 μl del trattamento idoneo alla concentrazione di 400 μg ml-1 (Tavolo 2) è stato instillato nella vescica utilizzando un catetere di calibro 24. Il trattamento e il veicolo (acqua o urea) sono rimasti nella vescica per 1 ora prima della rimozione del catetere. La vescica è stata nuovamente svuotata mediante massaggio addominale e i topi si sono ripresi dall'anestesia nelle loro gabbie, sostituendo la segatura della gabbia degli animali 24 ore dopo il trattamento per rimuovere la contaminazione radioattiva.
Efficacia terapeutica determinata dalla risonanza magnetica
Sono stati eseguiti due studi MRI su ciascun topo: (1) tra i giorni 7 e 14 dopo l'inoculazione del tumore per randomizzare gli animali tra i gruppi e misurare i volumi tumorali iniziali (pretrattamento); (2) tra i giorni 16 e 21 dopo l'inoculazione del tumore (post-trattamento) per valutare l'efficacia terapeutica. La risonanza magnetica è stata condotta utilizzando gli scanner Bruker BioSpec da 7 T e Bruker BioSpec da 11.7 T (entrambi con il software ParaVision 7), a seconda della disponibilità. Ciò non ha influito sui risultati poiché il campo esterno non è fondamentale per l'imaging anatomico14. Gli esperimenti di imaging sono stati condotti utilizzando gli stessi parametri di imaging e l'elaborazione spiegata sopra (Monitoraggio delle dimensioni del tumore). Nel caso dello scanner da 11.7 T la configurazione consisteva in una bobina sulla superficie del cuore del mouse per la ricezione e una bobina volumetrica per la trasmissione. I volumi del tumore in ciascuna fetta sono stati determinati da volumi di interesse prelevati manualmente che coprivano l'area del tumore.
analisi statistica
Negli studi di imaging PET, le percentuali di dose iniettata (% ID) e dose iniettata per volume tumorale (% ID cm-3) sono stati confrontati utilizzando ANOVA unidirezionale. Le differenze tra i gruppi sono state determinate utilizzando il test dei confronti multipli di Tukey. NTV nella sezione RNT è stata ottenuta da a t-test di valori spaiati. Si presumeva che la distribuzione dei dati fosse normale, ma ciò non è stato formalmente testato. Le analisi statistiche sono state eseguite con GraphPad Prism v.8.
Reporting summary
Ulteriori informazioni sulla progettazione della ricerca sono disponibili nel Riepilogo dei rapporti sul portafoglio naturalistico collegato a questo articolo.
- Distribuzione di contenuti basati su SEO e PR. Ricevi amplificazione oggi.
- PlatoData.Network Generativo verticale Ai. Potenzia te stesso. Accedi qui.
- PlatoAiStream. Intelligenza Web3. Conoscenza amplificata. Accedi qui.
- PlatoneESG. Carbonio, Tecnologia pulita, Energia, Ambiente, Solare, Gestione dei rifiuti. Accedi qui.
- Platone Salute. Intelligence sulle biotecnologie e sulle sperimentazioni cliniche. Accedi qui.
- Fonte: https://www.nature.com/articles/s41565-023-01577-y
- :È
- :non
- ][P
- 000
- 1
- 10
- 100
- 11
- 13
- 14
- 15%
- 16
- 160
- 20
- 200
- 2015
- 2017
- 2018
- 2019
- 2020
- 2021
- 22
- 23
- 24
- 25
- 250
- 30
- 300
- 33
- 35%
- 3d
- 400
- 41
- 50
- 500
- 51
- 52
- 53
- 7
- 70
- 8
- 9
- 95%
- a
- WRI
- sopra
- accordo
- accumulazione
- Raggiungere
- raggiunto
- acquisito
- l'acquisizione di
- acquisizione
- operanti in
- attivato
- attivo
- attività
- aggiunto
- l'aggiunta di
- Inoltre
- amministrazione
- influenzare
- Dopo shavasana, sedersi in silenzio; saluti;
- dopo
- ancora
- Mirato
- ARIA
- AL
- alex
- algoritmo
- Tutti
- permesso
- lungo
- Alzheimer
- tra
- quantità
- amiloide
- an
- analisi
- .
- Presentatrice
- ed
- animali
- animali
- anticorpo
- in qualsiasi
- applicazioni
- applicato
- AMMISSIONE
- opportuno
- approvato
- acqua
- SONO
- RISERVATA
- articolo
- AS
- assunto
- ipotesi
- At
- Autorità
- automaticamente
- disponibilità
- disponibile
- media
- media
- b
- BABB
- Larghezza di banda
- base
- BE
- prima
- comportamento
- essendo
- sotto
- fra
- Bloccare
- stile di vita
- bollito
- entrambi
- Cervello
- onere
- ma
- by
- gabbie
- calcolato
- chiamata
- stanza
- Cancro
- Custodie
- casi
- CCD
- cella
- Celle
- Modifiche
- carica
- chimico
- Radura
- clicca
- Info su
- Chiudi
- bobina
- combinazione
- combinando
- comitato
- paragonabile
- rispetto
- confronto
- completamente
- completando
- concentrazione
- condizione
- condizioni
- condotto
- fiducia
- CONFERMATO
- consecutivo
- continuità
- continuamente
- di controllo
- controllata
- al contrario
- Froids
- corretto
- Correzioni
- Corrispondente
- Consiglio
- accoppiato
- copertura
- creare
- creato
- Creazione
- Creative
- criteri
- critico
- Scansioni CT
- costume
- taglio
- Scuro
- dati
- dataset
- giorno
- Giorni
- de
- definito
- consegna
- Dipendente
- descritta
- Design
- desiderato
- dettagli
- rilevato
- rivelazione
- Determinare
- determinato
- determinazione
- sviluppato
- dispositivo
- diagnostica
- DID
- differenza
- differenze
- diverso
- Emittente
- digitale
- diluito
- direzione
- Malattia
- piatto
- Dispersione
- distribuzione
- distribuzioni
- Diviso
- do
- effettua
- fatto
- dose
- disegnato
- Cadere
- durata
- dinamico
- dinamica
- e
- E&T
- ogni
- facile
- eco
- efficacia
- o
- incorporato
- enable
- Abilita
- endpoint
- migliorata
- Intero
- enzimatica
- attrezzato
- sviluppate
- stabilimento
- stima
- Etere (ETH)
- etica
- europeo
- valutare
- Ogni
- esame
- superato
- Tranne
- aspettativa
- previsto
- esperimento
- sperimentale
- esperimenti
- ha spiegato
- export
- esterno
- facilitare
- fattore
- la donna
- campo
- Fico
- figura
- pieno
- filtro
- filtri
- finale
- Infine
- Nome
- adatto
- fisso
- Cromatografia
- seguito
- i seguenti
- Nel
- modulo
- formaldeide
- formalmente
- via
- quattro
- da
- congelati
- function
- ulteriormente
- divario
- ge
- GE Healthcare
- Generale
- generare
- generato
- Dare
- buono
- gradienti
- Grammo
- Green
- Gruppo
- Gruppo
- Crescita
- linee guida
- Avere
- he
- Salute e benessere
- assistenza sanitaria
- sano
- Cuore
- HTTPS
- huang
- umano
- ICAP
- ID
- identico
- if
- Immagine
- analisi dell'immagine
- immagini
- Imaging
- subito
- immersa
- migliora
- in
- incluso
- incorporando
- crescente
- incubato
- Incubando
- INCUBAZIONE
- studente indipendente
- Index
- induzione
- informazioni
- inizialmente
- interno
- interno
- strumenti
- integrato
- interattivo
- interesse
- interno
- ai miglioramenti
- l'introduzione di
- iterazione
- iterazioni
- SUO
- KDA
- conservazione
- kit
- l2
- grandi
- laser
- Luxinar SR AOM
- strato
- galline ovaiole
- partenza
- a sinistra
- lente
- lenti
- Vita
- leggera
- linea
- LINK
- connesso
- Caricamento in corso
- carichi
- locale
- spento
- Macro
- macro
- mantenere
- manutenzione
- manualmente
- mask
- Mascherine
- corrispondenza
- materiale
- Materiale
- matematico
- Matrice
- massimo
- si intende
- misurare
- misurato
- misurazioni
- di misura
- Media
- Memorie
- merino
- Metanolo
- metodi
- topi
- Microscopio
- Microscopia
- verbale
- ordine
- minore
- misto
- miscela
- ML
- Mobile
- Moda
- modello
- modelli
- modificato
- modulo
- molecolare
- monitorati
- monitoraggio
- movimento
- mouse
- MRI
- MS
- MT
- multiplo
- nanotecnologia
- Natura
- reti
- Neutres
- New
- no
- Rumore
- normale
- obiettivo
- osservato
- ottenere
- ottenuto
- of
- Vecchio
- on
- ONE
- operativo
- operativa
- Operazioni
- ottimizzati
- or
- Orca
- i
- ancora
- per una notte
- Oxford
- Oxygen
- Carta
- parametri
- Parco
- parte
- particella
- PBS
- piolo
- penetrazione
- per
- eseguita
- esegue
- animale domestico
- Petri
- fasi
- Perno
- pixel
- posto
- piano
- Planes
- Platone
- Platone Data Intelligence
- PlatoneDati
- punto
- punti
- lavori
- posizione
- posizionato
- Precisione
- preparato
- presenti
- in precedenza
- procedura
- procedure
- lavorazione
- progressivamente
- protocollo
- Qi
- quantitativo
- radio
- casuale
- Randomizzato
- RAT
- tasso
- rapporto
- Reagire
- reazione
- Lettore
- ragionevole
- motivi
- ricevuto
- reception
- registrato
- Recuperare
- Rosso
- ridurre
- Ridotto
- riduce
- riferimento
- regione
- è rimasta
- resti
- rimuovere
- rimosso
- rimozione
- Segnalati
- Reportistica
- rappresentante
- necessario
- riparazioni
- resistente
- Risoluzione
- rispettivamente
- risposta
- colpevole
- risultante
- Risultati
- robot
- Prenotazione sale
- corre
- s
- SA
- stesso
- risparmio
- scansioni
- sparpagliato
- SCI
- scientifico
- copione
- Secondo
- secondario
- Sezione
- sezioni
- vedere
- segmento
- segmentazione
- selezionato
- Sequenza
- servito
- impostazioni
- alcuni
- foglio
- mostrato
- Signal
- Distribuzione del segnale
- simile
- da
- singolo
- SIX
- Taglia
- Taglia
- piccole
- So
- sodio
- Software
- soluzione
- Soluzioni
- Spaziale
- Stabilità
- pila
- Stacks
- statistiche
- Stellaris
- Passi
- memorizzati
- striscia
- studente
- studi
- successivo
- Successivamente
- suite
- superficie
- Sintomi
- sintesi
- sistema
- T
- T1
- tavolo
- presa
- Tech
- Tecnologie
- cannocchiale
- modello
- carnagione
- test
- testato
- che
- Il
- loro
- Li
- poi
- teorico
- Terapeutico
- terapia
- questo
- a fondo
- quelli
- tre
- tridimensionale
- Attraverso
- per tutto
- tempo
- volte
- fazzoletto di carta
- a
- insieme
- strumenti
- Totale
- verso
- verso
- trasformazione
- trattamento
- tritone
- cerca
- tumori
- seconda
- Digitare
- tipicamente
- non influenzato
- per
- fino a quando
- su
- utilizzato
- Utente
- utilizzando
- Valori
- veicolo
- contro
- via
- Video
- Video
- Visualizza
- virtuale
- visualizzare
- vivo
- volume
- volumi
- volumetrica
- camminatore
- bacchetta magica
- Prima
- lavaggio
- Water
- we
- Settimane
- peso
- Benessere
- sono stati
- Ruota
- quale
- while
- bianca
- finestre
- con
- entro
- senza
- X
- dare la precedenza
- cedevole
- i rendimenti
- zefiro
- zoom