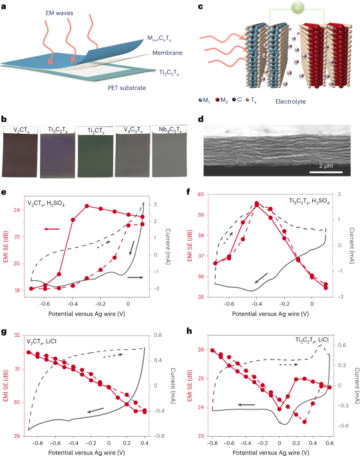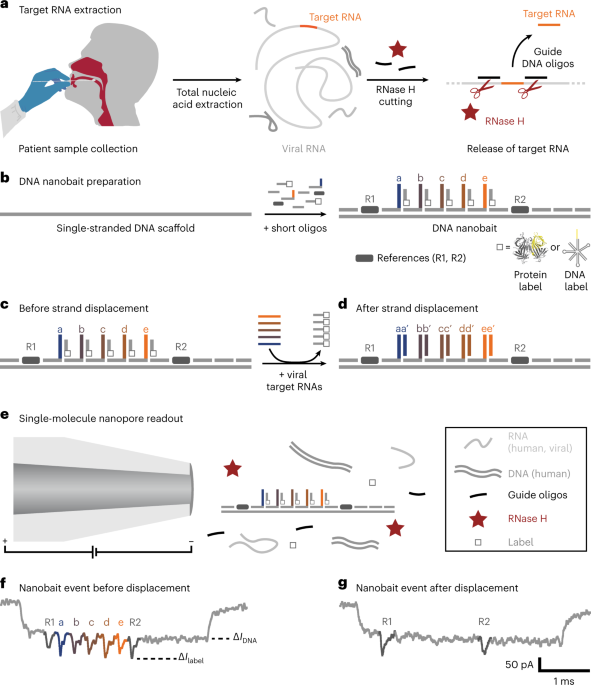
Pengambilan sampel pasien
Pengambilan sampel pasien dilakukan dengan cara usap bagian belakang tenggorokan (oropharyngeal swab) pasien, seperti yang telah dijelaskan sebelumnya26. Sampel dikumpulkan dari pasien dengan gambaran klinis mirip COVID-19 dan diuji dengan qRT-PCR setelah ekstraksi asam nukleat. Secara singkat, setelah pengumpulan, penyeka ditempatkan ke dalam tabung sampel berlabel yang berisi buffer lisis (4 M guanidine thiocyanate, 25 mM Tris-HCl, 0.5% β-mercaptoethanol dan MS2 RNA (200 ng µl-1; Roche)). Tabung diaduk perlahan untuk memastikan pemerataan buffer lisis. Langkah-langkah keamanan telah dijelaskan sebelumnya dan dilakukan di laboratorium CL2 bersertifikat26.
Ekstraksi asam nukleat
Asam nukleat total diekstraksi menggunakan sistem berbasis kolom spin dan seperti yang digunakan oleh pengujian qRT-PCR standar26. Kontrol amplifikasi internal (MS2 (~6 × 104 PFU ml-1) per 10 ml buffer lisis) ditambahkan pada top-up buffer lisis (25 µl per 10 ml buffer lisis). Sampel dielusi dalam 100 µl air bebas nuklease (nfH2HAI; Invitrogen) dan didiamkan selama 1 menit sebelum sentrifugasi selama 1 menit pada 21,130×g (15,000 rpm) dalam kompor mikro benchtop. Sampel yang dielusi langsung dikenai qRT-PCR. Ekstrak asam nukleat yang tersisa disimpan pada suhu −80 °C dan selanjutnya digunakan untuk penginderaan nanobait-nanopore.
qRT-PCR untuk SARS-CoV-2
Deteksi SARS-CoV-2 dilakukan seperti yang dijelaskan sebelumnya26. Per reaksi, master mix berisi 12.5 µl 2× Luna Universal Probe One-Step reaction mix, 0.5 µl 20 µM Wu forward primer (5′-ATGGGTTGGGATTATCCTAAATGTGA-3′), 0.5 µl 20 µM Wu reverse primer (5′ -GCAGTTGTGGCATCTCCTGATGAG-3′), 0.3 µl dari 10 µM MGB Probe 3 fluorescein (5′-ATGCTTAGAATTATGGCCTCAC-3′), 0.5 µl dari 10 µM primer maju kontrol internal untuk MS2 RNA, 0.5 µl dari 10 µM primer mundur kontrol internal untuk MS2 RNA, 0.3 µl probe internal 10 µM (MS2 ROX), 1 µl Luna WarmStart RT Enzyme Mix dan 3.9 µl nfH2O. Kemudian, 20 µl master mix dimasukkan ke dalam masing-masing sumur dari pelat 96-sumur dan kemudian digabungkan dengan 5 µl setiap ekstrak. Kontrol ekstraksi dan amplifikasi internal MS2 yang menjalani protokol ekstraksi penuh dimasukkan sebagai kontrol ekstraksi negatif dalam minimal dua sumur pada pelat qRT-PCR. Untuk menentukan potensi kontaminasi pada proses qRT-PCR, 5 µl nfH2O dimasukkan sebagai kontrol negatif qRT-PCR. Kemudian, 5 µl plasmid template SARS-CoV-2 berduri dimasukkan ke dalam satu sumur sebagai kontrol positif qRT-PCR. Setelah menambahkan 5 µl dari masing-masing sampel ke sumur yang ditentukan, pelat ditutup dengan segel plastik bening secara optik. Pelat disentrifugasi selama 1 menit pada 2,000 ×g (1,000 rpm) pada 4 °C dan kemudian dimasukkan ke dalam mesin qRT-PCR (QuantStudio, Thermo Fisher Scientific) dan prosesnya diparametrikan. Sinyal untuk fluorescein (FAM) dan carboxyrhodamine (ROX) diperoleh. ROX digunakan untuk mendeteksi kontrol MS2 internal dan fluorescein digunakan untuk mendeteksi RNA SARS-CoV-2. Pengujian dilakukan selama 2 menit pada 25 °C, 15 menit pada 50 °C (untuk reverse transcriptase), 2 menit pada 90 °C, sebelum 45 siklus 95 °C selama 3 detik diikuti oleh 60 °C selama 30 detik . Hasilnya ditentukan oleh konfirmasi kontrol positif yang benar (amplifikasi plasmid), kontrol ekstraksi dan amplifikasi semua sampel (saluran ROX), tidak ada amplifikasi pada kontrol negatif dan nilai rata-rata kontrol yang konsisten. Kepositifan SARS-CoV-2 dikonfirmasi dengan amplifikasi pada saluran fluorescein dengan kurva sigmoidal yang sesuai dengan nilai CT ≤36. Nilai CT dari probe MS2 dan MGB 3 dipertahankan untuk melacak kualitas dan reproduktifitas pengujian44.
Pemotongan RNase H yang dapat diprogram untuk nanobait
Untuk penginderaan nanopori, kontrol RNA SARS-CoV-2, ekstrak asam nukleat (sampel pasien) atau RNA virus MS2 digunakan lebih lanjut untuk deteksi dengan nanobait. Pertama, kami mencampur oligos pemandu dengan sampel dan memanaskannya hingga 70 ° C selama 5 menit. RNase H (5,000 unit per ml; NEB) ditambahkan, dicampur dan dipanaskan selama 20 menit pada suhu 37 °C untuk memungkinkan enzim memotong RNA dalam DNA: RNA hibrid yang secara efektif melepaskan target RNA. RNase H secara termal tidak aktif dengan inkubasi pada 65 ° C selama 10 menit. Panduan oligos divalidasi untuk tidak membentuk struktur intramolekul, homo atau heterodimer menggunakan perangkat lunak NUPACK45. Untuk pengukuran dengan target yang tidak ada, protokol yang sama termasuk panduan oligos digunakan. Pengukuran kontrol tidak menunjukkan perpindahan, dan karenanya, kami dapat mengecualikan ikatan silang yang substansial dari oligo pemandu.
Properti urutan target virus untuk nanobait
Panjang target, panjang tumpuan, dan konten GC dipilih untuk memastikan hibridisasi yang optimal21. Untuk desain nanobait DNA, sekuens target dipilih untuk berada di wilayah yang dilestarikan dari genom virus dan memiliki kandungan GC 40-60% untuk membentuk dupleks 20 bp yang stabil. Panjang tumpuan dipilih menjadi panjang 6 nt dan memiliki kandungan GC 40-60%. Kami menguji semua urutan untuk potensi interaksi intramolekul atau homodimer sangat stabil yang tidak diinginkan menggunakan perangkat lunak NUPACK (aplikasi web 2020)45. Kemudian, kami melakukan pemeriksaan reaktivitas silang antara beberapa situs yang digunakan dalam setiap percobaan45.
Persiapan bunga DNA untuk nanobait
Kami merancang bunga DNA sebagai label lain untuk deteksi RNA SARS-CoV-2 dari sampel pasien. Tiga bunga DNA spesifik untuk setiap target SARS-CoV-2 (persimpangan tujuh arah, 7WJa, 7WJb, dan 7WJc) disiapkan secara terpisah. Mengambil 7WJc sebagai contoh, 4 μM untai DNA J1, J2, J3 dan J4c (Tabel Tambahan 1) dicampur bersama dalam buffer TM (10 mM Tris-HCl, 10 mM MgCl2, pH 8.0) dan dipanaskan hingga 90 °C selama 5 menit, kemudian didinginkan hingga 65 °C selama 15 menit, 45 °C selama 15 menit, 37 °C selama 20 menit, 25 °C selama 20 menit dan terakhir hingga 4 ° C selama 20 menit. Strand J4c digantikan oleh J4b untuk menyiapkan 7WJb. Untuk 7WJa, untuk menghindari pelipatan sendiri di situs 43 pada nanobait, J1, J2, J3 J4a dan C43 dicampur bersama sebelum anil.
Perakitan sendiri nanobait DNA
Nanobait DNA dirakit dengan mencampur DNA M13 beruntai tunggal linier (M13mp18, 7,249 nt, Guild Biosciences, 100 nM) dengan oligonukleotida komplementer pendek12 (beberapa di antaranya menyimpan struktur referensi dan menangkap untaian) dan dengan menambahkan sebagian untai komplementer yang 3′-dibiotinilasi untuk reaksi perpindahan untai yang dimediasi tumpuan. DNA M13 yang dilinearisasi (panjang 7,228 nt) dilengkapi dengan oligonukleotida, sehingga menciptakan nanobait beruntai ganda dengan overhang dua terminal empat deoxythymidine yang mencegah multimerisasi12. Campuran tersebut mengandung 20 nM DNA M13 linier, 60 nM oligonukleotida (tiga kali lebih banyak dari DNA M13), untaian 3′-biotinilasi dalam konsentrasi 180 nM, 10 mM MgCl2 dan 1× TE (10 mM Tris–HCl, 1 mM EDTA, pH 8.0). Itu dicampur dengan pemipetan dan diputar sebelum dipanaskan hingga 70 ° C selama 30 detik dan didinginkan selama 45 menit hingga suhu sekitar. Kelebihan oligonukleotida dihilangkan menggunakan filter sentrifugal Amicon Ultra 0.5 ml dengan cutoff 100 kDa dengan bufer pencuci (10.0 mM Tris–HCl pH 8.0, 0.5 mM MgCl2). Jika bunga DNA digunakan sebagai label, helai komplementer sebagian yang membawanya diinkubasi dalam 10 mM MgCl2 selama 2 jam pada suhu sekitar, dan selanjutnya, filtrasi Amicon dilakukan seperti dijelaskan di atas. Asimetri desain nanobait memungkinkan identifikasi situs pengikatan yang tidak ambigu. Nanobait disimpan sampai digunakan untuk percobaan lebih lanjut di bawah 4–10 °C dalam 0.5 mM MgCl2, 10.0 mM Tris–HCl, pH 8.0. Desain nanobait diperiksa dengan pembacaan nanopore sebelum setiap pengukuran.
Pembacaan Nanopore dari DNA nanobait
Nanobait dicampur dengan sampel (ekstrak asam nukleat atau target virus yang dimurnikan dengan kelebihan sepuluh kali lipat) dalam 10 mM MgCl2 dan 100 mM NaCl. Campuran (5 μl) diinkubasi pada suhu kamar (~ 10 menit) sampai disiapkan untuk pengukuran nanopori. Perbedaan komposisi urutan target dan karakteristik fisiknya dapat menyebabkan variabilitas dalam hibridisasi dan karenanya efisiensi perpindahan situs penginderaan21. Kami telah menggunakan htRNA (100 ng μl-1; Invitrogen) sebagai latar belakang yang ditunjukkan, untuk menunjukkan bahwa tidak ada sinyal non-spesifik yang diinduksi oleh RNA asli manusia. Untuk pengukuran nanopore, sampel diencerkan menjadi <0.5 nM nanobait (untuk target virus yang dimurnikan) atau 4.7 μl sampel pasien yang dipotong RNase-H dicampur dengan 0.3 μl streptavidin monovalen (SAe1D3)18 (1 μM), 5 μl LiCl (4.0 M) dan 5.0 μl LiCl (8.0 M). Kami telah membuat nanopori 14 ± 3 nm (rata-rata ± standar deviasi).12 menggunakan kapiler kaca kuarsa dengan diameter luar 0.5 mm dan diameter dalam 0.2 mm (Sutter Instrument) dengan laser-assisted puller P-2000 (Sutter Instrument). Campuran tersebut dipipet dalam chip nanopore polydimethylsiloxane, dan semua pengukuran dilakukan pada tegangan konstan 600 mV. Detail pengukuran nanopori ditunjukkan pada Tabel Tambahan 30.
Analisis data nanopori waktu nyata
Analisis data nanopori dijelaskan secara rinci di Bagian Tambahan 14. Secara singkat, peristiwa nanobait disaring dari jejak arus ionik mentah dan kemudian wilayah deteksi ditentukan, dan informasi keberadaan lonjakan di setiap situs spesifik diekstraksi. Efisiensi perpindahan yang diplot dihitung sebagai efisiensi perpindahan untuk pengukuran dikurangi dengan kontrol tanpa target untuk setiap situs (50 peristiwa nanobait untuk masing-masing dari tiga rekaman nanopori), kecuali dinyatakan sebaliknya:
$$begin{array}{l}{mathrm{Displacement}},{mathrm{efisiensi}} =frac{1}{3}mathop {sum}limits_{n = 1}^3 tersisa{ {1 -frac{1 }{{50}}mathop {sum}limits_{n = 1}^{50} {left[ {fleft( n right) = left( {frac{{1,,mathrm{peak}}}{{0,, {mathrm{no}},{mathrm{peak}}}}} kanan)} benar]_{{{{mathrm{target}}}}}} } kanan}\ – frac{1}{3}mathop {sum }batas_{n = 1}^3 {kiri{ {1 – frac{1}{{50}}mathop {jumlah }batas_{n = 1}^{50} kiri[ {fleft( n kanan) = kiri( { frac{{1,,mathrm{peak}}}{{0,,mathrm{no}},{mathrm{peak}}}} kanan)} kanan]_{{{mathrm{no}}}},{ {{mathrm{target}}}}}} kanan}} end{array}.$$
Kami memverifikasi bahwa jaringan saraf convolutional QuipuNet27 mampu menganalisis data nanopori secara real-time mengikuti prosedur yang dijelaskan. Sebelumnya, kami mendemonstrasikan bahwa dengan sekitar sepuluh peristiwa, kami mencapai keyakinan 99% dalam deteksi positif struktur DNA rancangan kami46.
pencitraan AFM
Pencitraan nanobaits AFM (Nanosurf Mobile S) dilakukan di udara dalam mode non-kontak. Struktur nanobait diencerkan menjadi 1 ng μl-1 dalam 1 mM MgCl2 dan 10 μl ditambahkan ke mika yang baru dibelah, diinkubasi selama 1 menit, dibilas dengan air Milli-Q yang disaring dan kemudian dikeringkan dengan nitrogen. Sebelum dipindai, pelat mika ditempelkan pada tahap sampel AFM menggunakan pita perekat dua sisi. Visualisasi dan analisis gambar dilakukan menggunakan Gwyddion (versi 2.60).
Analisis statistik
Untuk semua pengukuran, interval kepercayaan 99.9% untuk efisiensi perpindahan dihitung. Signifikansi statistik antara dua situs tanpa dan dengan target diuji menggunakan Student's dua sisi t-uji.
Ringkasan pelaporan
Informasi lebih lanjut tentang desain penelitian tersedia di Ringkasan Pelaporan Portofolio Alam tertaut ke artikel ini.
- Konten Bertenaga SEO & Distribusi PR. Dapatkan Amplifikasi Hari Ini.
- Platoblockchain. Intelijen Metaverse Web3. Pengetahuan Diperkuat. Akses Di Sini.
- Sumber: https://www.nature.com/articles/s41565-022-01287-x
- 000
- 1
- 10
- 100
- 11
- 110
- 2014
- 2016
- 2017
- 2018
- 2020
- 2021
- 7
- 70
- 9
- a
- atas
- absen
- akademik
- diperoleh
- menambahkan
- Setelah
- UDARA
- Semua
- memungkinkan
- Ambient
- analisis
- Jangkar
- dan
- Lain
- Aplikasi
- pendekatan
- sesuai
- sekitar
- artikel
- dirakit
- tersedia
- kembali
- latar belakang
- sebelum
- Bel
- antara
- mengikat
- meniup
- BP
- secara singkat
- penyangga
- dihitung
- mampu
- menangkap
- membawa
- Tersertifikasi
- Saluran
- karakteristik
- memeriksa
- chen
- keping
- jelas
- Klinis
- koleksi
- bergabung
- komplementer
- konsentrasi
- kepercayaan
- DIKONFIRMASI
- konsisten
- konstan
- Konten
- kontrol
- kontrol
- jaringan saraf convolutional
- membuat
- terbaru
- melengkung
- Memotong
- pemotongan
- siklus
- data
- analisis data
- menunjukkan
- dijelaskan
- Mendesain
- dirancang
- desain
- rinci
- rincian
- Deteksi
- Menentukan
- ditentukan
- penyimpangan
- perbedaan
- digital
- langsung
- distribusi
- dna
- turun
- setiap
- efektif
- efisiensi
- efisiensi
- Teknik
- memastikan
- kesalahan
- Eter (ETH)
- Bahkan
- peristiwa
- contoh
- menjelaskan
- ekstrak
- ekstraksi
- Ekstrak
- filter
- Akhirnya
- Pertama
- bunga
- diikuti
- berikut
- bentuk
- Depan
- dari
- penuh
- lebih lanjut
- genom
- kaca
- membimbing
- sangat
- HTTPS
- manusia
- Hibrida
- Identifikasi
- gambar
- Pencitraan
- implementasi
- in
- termasuk
- Termasuk
- diinkubasi
- INKUBASI
- informasi
- instrumen
- interaksi
- intern
- Ionik
- IT
- KDA
- label
- memimpin
- Panjang
- LINK
- terkait
- Panjang
- luna
- mesin
- menguasai
- bahan
- pengukuran
- Mika
- mungkin
- minimum
- campur aduk
- Percampuran
- campuran
- ML
- mobil
- mode
- MOL
- beberapa
- nano
- nanopori
- asli
- Alam
- negatif
- jaringan
- saraf
- saraf jaringan
- Buka
- optimal
- jika tidak
- pasangan
- pasien
- pasien
- prestasi
- fisik
- gambar
- plastik
- plato
- Kecerdasan Data Plato
- Data Plato
- portofolio
- positif
- Kepositifan
- potensi
- Mempersiapkan
- siap
- kehadiran
- mencegah
- sebelumnya
- pertama
- penyelidikan
- proses
- properties
- Protein
- protokol
- kualitas
- Mentah
- mencapai
- reaksi
- real-time
- wilayah
- daerah
- Pers
- yang tersisa
- Dihapus
- Pelaporan
- penelitian
- Hasil
- membalikkan
- RNA
- roche
- Kamar
- rt
- aturan
- Run
- Safety/keselamatan
- sama
- SARS-CoV-2
- pemindaian
- Bagian
- terpilih
- Urutan
- Pendek
- Menunjukkan
- ditunjukkan
- sinyal
- makna
- serentak
- tunggal
- situs web
- Situs
- kecil
- Perangkat lunak
- beberapa
- tertentu
- pintal
- stabil
- Tahap
- berdiri
- standar
- menyatakan
- statistik
- Tangga
- tersimpan
- Helai
- Kemudian
- besar
- sistem
- tabel
- pengambilan
- target
- target
- Template
- sepuluh
- Grafik
- dengan demikian
- tiga
- kali
- TM
- untuk
- bersama
- Total
- jalur
- Ultra
- bawah
- unit
- Universal
- divalidasi
- nilai
- Nilai - Nilai
- diverifikasi
- versi
- melalui
- virus
- visualisasi
- Tegangan
- W
- air
- jaringan
- aplikasi web
- Wells
- yang
- tanpa
- wu
- zephyrnet.dll