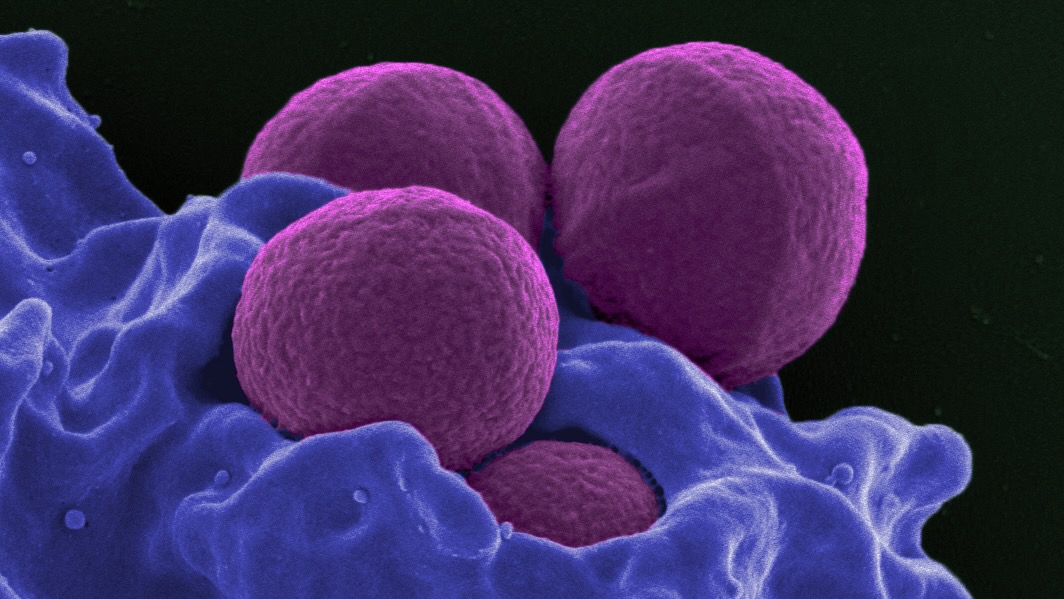
Les antibiotiques ont sauvé d’innombrables vies et constituent un outil crucial dans la médecine moderne. Mais nous perdons du terrain dans notre lutte contre les bactéries. Au milieu du siècle dernier, les scientifiques découvert de toutes nouvelles classes d'antibiotiques. Depuis lors, le rythme des découvertes a ralenti et la prévalence des bactéries résistantes aux antibiotiques a augmenté.
Il reste probablement des antibiotiques à découvrir, mais l’univers chimique est trop vaste pour que quiconque puisse le rechercher. Ces dernières années, les scientifiques se sont tournés vers l’IA. Les algorithmes d’apprentissage automatique peuvent réduire un nombre considérable de configurations chimiques potentielles à une poignée de candidats prometteurs à tester.
Jusqu’à présent, les scientifiques ont utilisé l’IA pour trouver des composés uniques dotés de propriétés antibiotiques. Mais dans une nouvelle étude, publié hier dans Nature, les chercheurs du MIT affirment avoir construit et testé un système capable d'identifier de toutes nouvelles classes d'antibiotiques et de prédire lesquels sont probablement sans danger pour les humains.
L'IA a passé au crible plus de 12 millions de composés et a découvert une classe inconnue d'antibiotiques qui s'est révélée efficace chez la souris contre le Staphylococcus aureus résistant à la méthicilline (SARM), une souche mortelle d'insecte résistant aux médicaments.
Bien que ces antibiotiques découverts par l’IA doivent encore prouver leur sécurité et leur efficacité chez l’homme en passant le test standard des tests cliniques, l’équipe pense que leur travail peut accélérer la découverte dès le début et, espérons-le, augmenter notre taux de réussite global.
Explorer l’espace de la drogue
Les scientifiques utilisent de plus en plus les assistants de l’IA pour accélérer le processus de découverte. Le plus célèbre est peut-être AlphaFold de DeepMind, un programme d’apprentissage automatique capable de modéliser la forme des protéines, les éléments de base de notre corps. L’idée est qu’AlphaFold et ses descendants peuvent accélérer le processus ardu de recherche sur les médicaments. Leur conviction est si forte que DeepMind a créé une filiale en 2021, Laboratoires isomorphes, dédié à faire exactement cela.
D’autres approches de l’IA se sont également révélées prometteuses. Un groupe du MIT, en particulier, s'est concentré sur le développement d'antibiotiques entièrement nouveaux pour lutter contre les superbactéries. Leur première étude, publiée en 2020, a établi que cette approche pourrait fonctionner, lorsqu'ils ont découvert l'halicine, un antibiotique jusqu'alors inconnu qui pourrait facilement éliminer E. coli résistant aux médicaments.
Lors d’un suivi plus tôt cette année, l’équipe a ciblé Acinetobacter baumannii, « l’ennemi public n°1 des infections bactériennes multirésistantes », selon Jonathan Stokes de l’Université McMaster, auteur principal de l’étude.
« Acinetobacter peut survivre sur les poignées de porte et les équipements des hôpitaux pendant de longues périodes, et il peut absorber les gènes de résistance aux antibiotiques de son environnement. Il est désormais très courant de trouver des isolats d’A. baumannii résistants à presque tous les antibiotiques », Stokes a dit à l'époque.
Après avoir passé au peigne fin 6,680 240 composés en seulement deux heures, l’IA a mis en évidence quelques centaines de candidats prometteurs. L’équipe en a testé XNUMX qui étaient structurellement différents des antibiotiques existants. Ils ont découvert neuf candidats prometteurs, dont un, l'abaucine, qui s'est révélé très efficace contre A. baumannii.
Les deux études ont montré que l’approche pouvait fonctionner, mais n’ont abouti qu’à des candidats uniques sans aucune information sur why ils étaient efficaces. Les algorithmes d’apprentissage automatique sont notoirement des boîtes noires : ce qui se passe « entre les oreilles » pour ainsi dire est souvent un mystère complet.
Dans la dernière étude, le groupe a visé un autre adversaire connu, le SARM, mais cette fois-ci, ils ont enchaîné plusieurs algorithmes pour améliorer les résultats et mieux éclairer le raisonnement de l’IA.
Basculer l'interrupteur
Le dernier limier antibiotique de l’équipe s’est entraîné sur quelque 39,000 XNUMX composés, y compris leur structure chimique et leur capacité à tuer le SARM. Ils ont également formé des modèles distincts pour prédire la toxicité d'un composé donné pour les cellules humaines.
"Vous pouvez représenter pratiquement n'importe quelle molécule sous la forme d'une structure chimique, et également indiquer au modèle si cette structure chimique est antibactérienne ou non", Felix Wong, postdoctorant à l'IMES et au Broad Institute du MIT et de Harvard, dit MIT Nouvelles. « Le modèle est formé sur de nombreux exemples comme celui-ci. Si vous lui donnez ensuite une nouvelle molécule, un nouvel arrangement d’atomes et de liaisons, il peut vous indiquer la probabilité que ce composé soit antibactérien.
Une fois terminé, l’équipe a introduit plus de 12 millions de composés dans le système. L'IA a réduit cette énorme liste à environ 3,600 283 composés organisés en cinq classes, en fonction de leurs structures, qui, selon elle, auraient une certaine activité contre le SARM et seraient peu toxiques pour les cellules humaines. L'équipe a arrêté une liste finale de XNUMX candidats à tester.
Parmi ceux-ci, ils en ont trouvé deux de la même classe, c’est-à-dire qu’ils avaient des composants structurels similaires censés contribuer à l’activité antimicrobienne, qui étaient très efficaces. Chez la souris, les antibiotiques ont combattu à la fois une infection cutanée et une infection systémique en éliminant 90 pour cent des bactéries SARM présentes.
Notamment, alors que leurs travaux antérieurs s'attaquaient aux bactéries Gram-négatives en perturbant les membranes cellulaires, le SARM est Gram-positif et possède des parois plus épaisses.
"Nous disposons de preuves assez solides que cette nouvelle classe structurelle est active contre les agents pathogènes à Gram positif en dissipant sélectivement la force motrice des protons dans les bactéries", a déclaré Wong. "Les molécules attaquent sélectivement les membranes cellulaires bactériennes, d'une manière qui n'entraîne pas de dommages substantiels aux membranes cellulaires humaines."
En rendant leur IA explicable, l’équipe espère se concentrer sur les structures qui pourraient éclairer les recherches futures ou contribuer à la conception d’antibiotiques plus efficaces en laboratoire.
Examens finaux
L’essentiel à noter ici est que même s’il semble que les nouveaux antibiotiques aient été efficaces chez les souris à très petite échelle, il reste un long chemin à parcourir avant qu’on vous en prescrive un.
Les nouveaux médicaments sont soumis à des tests et des essais cliniques rigoureux, et de nombreux candidats, même prometteurs, ne parviennent pas à passer de l’autre côté. Le domaine de la découverte de médicaments assistée par l’IA, plus généralement, est encore à ses débuts à cet égard. La première Les médicaments conçus par l'IA sont actuellement en cours d'essais cliniques, mais aucun n’a encore été approuvé.
L’espoir est néanmoins de pouvoir approvisionner plus rapidement le pipeline avec de meilleurs candidats.
Cela peut prendre de trois à six ans pour découvrir un nouvel antibiotique adapté aux essais cliniques, selon l'Université de Pennsylvanie César de la Fuente, dont le laboratoire effectue un travail similaire. Ensuite, il y a les épreuves elles-mêmes. Avec l’augmentation de la résistance aux antibiotiques, nous n’aurons peut-être pas ce genre de temps, sans parler du fait que les antibiotiques n’ont pas le retour sur investissement d’autres médicaments. Toute aide est la bienvenue.
« Maintenant, grâce aux machines, nous avons pu accélérer [le calendrier] » de la Fuente a dit Scientific American. « Dans mon propre travail et celui de mes collègues, par exemple, nous pouvons découvrir en quelques heures des milliers ou des centaines de milliers de candidats précliniques au lieu de devoir attendre trois à six ans. Je pense que l’IA en général a permis cela.
Il est encore tôt, mais si les antibiotiques découverts par l’IA s’avèrent utiles dans les années à venir, nous pourrons peut-être garder le dessus dans notre lutte de longue date contre les bactéries.
- Contenu propulsé par le référencement et distribution de relations publiques. Soyez amplifié aujourd'hui.
- PlatoData.Network Ai générative verticale. Autonomisez-vous. Accéder ici.
- PlatoAiStream. Intelligence Web3. Connaissance Amplifiée. Accéder ici.
- PlatonESG. Carbone, Technologie propre, Énergie, Environnement, Solaire, La gestion des déchets. Accéder ici.
- PlatoHealth. Veille biotechnologique et essais cliniques. Accéder ici.
- La source: https://singularityhub.com/2023/12/21/ai-discovers-a-new-class-of-antibiotics-after-scouring-12-million-compounds/
- :possède
- :est
- :ne pas
- $UP
- 000
- 1
- 12
- 2020
- 2021
- 39
- 600
- 90
- a
- capacité
- Capable
- accélérer
- Selon
- infection
- activité
- Après
- à opposer à
- AI
- objectif
- algorithmes
- aussi
- Bien que
- an
- ainsi que les
- Une autre
- Antibiotiques
- tous
- chacun.e
- apparaît
- une approche
- approches
- ,
- SONT
- autour
- arrangement
- AS
- At
- Attaquer
- auteur
- Bactéries
- Essentiel
- En gros
- Bataille
- BE
- était
- before
- cru
- croit
- Améliorée
- Big
- Noir
- Blocs
- sang
- Obligations
- tous les deux
- vaste
- Punaise
- Développement
- construit
- mais
- by
- CAN
- candidats
- cellule
- Cellules
- siècle
- enchaîné
- la chimie
- classe
- les classes
- Infos sur les
- essais cliniques
- Venir
- Commun
- complet
- composants électriques
- Composé
- contribuer
- conviction
- pourriez
- crédit
- crucial
- des dommages
- Date
- dévoué
- DeepMind
- Conception
- développement
- différent
- découvrez
- découvert
- Découvre
- découverte
- maladies
- do
- faire
- Ne pas
- down
- drogue
- découverte de médicament
- Médicaments
- e
- Plus tôt
- "Early Bird"
- Efficace
- activé
- fin
- énorme
- entièrement
- Environment
- l'équipements
- établies
- Pourtant, la
- Chaque
- preuve
- exemple
- exemples
- existant
- fait
- célèbre
- Fed
- few
- champ
- bats toi
- finale
- Trouvez
- Prénom
- cinq
- concentré
- Pour
- Force
- trouvé
- de
- avant
- L'extrémité avant
- avenir
- Gant
- Général
- généralement
- Donner
- donné
- Go
- Sol
- Réservation de groupe
- cultivé
- ait eu
- main
- poignée
- arrive
- harvard
- Vous avez
- ayant
- vous aider
- ici
- Surbrillance
- Frappé
- d'espérance
- Avec optimisme
- espoirs
- Hôpital
- HEURES
- HTTPS
- humain
- Les êtres humains
- cent
- Des centaines
- i
- idée
- identifier
- if
- éclairer
- améliorer
- in
- Y compris
- Améliore
- de plus en plus
- infection
- Infections
- Maladies infectieuses
- informer
- d'information
- plutôt ;
- Institut
- développement
- un investissement
- Isole
- IT
- SES
- jonathan
- juste
- ACTIVITES
- Tuer
- Genre
- connu
- laboratoire
- Nom de famille
- Nouveautés
- apprentissage
- comme
- Probable
- Liste
- Vit
- Location
- de longue date
- pas à perdre
- click
- machine learning
- Les machines
- maintenir
- a prendre une
- Fabrication
- de nombreuses
- Matière
- Mai..
- médecine
- mentionner
- souris
- Milieu
- pourrait
- million
- MIT
- modèle
- numériques jumeaux (digital twin models)
- Villas Modernes
- molécule
- PLUS
- (en fait, presque toutes)
- SARM
- my
- Mystère
- Nationales
- presque
- Besoin
- Nouveauté
- NIH
- neuf
- aucune
- Aucun
- noter
- maintenant
- numéros
- of
- souvent
- on
- ONE
- uniquement
- or
- Organisé
- Autre
- nos
- ande
- plus de
- global
- propre
- Rythme
- particulier
- En passant
- Personnes
- pour cent
- être
- périodes
- pipeline
- Platon
- Intelligence des données Platon
- PlatonDonnées
- défaillances
- préclinique
- prévoir
- prédit
- représentent
- assez
- prévalence
- précédent
- précédemment
- probabilité
- processus
- Programme
- PROMETTONS
- prometteur
- propriétés
- Protéines
- Prouver
- prouvé
- publié
- vite.
- assez
- Tarif
- facilement
- vraiment
- récent
- représentent
- un article
- chercheurs
- Résistance
- résistant
- Résultats
- retourner
- rigoureux
- Augmenter
- des
- Saïd
- même
- sauvé
- dire
- Escaliers intérieurs
- scientifiques
- Rechercher
- recherches
- supérieur
- séparé
- Établi
- plusieurs
- formes
- montré
- montré
- côté
- similaires
- depuis
- unique
- SIX
- Peau
- petit
- So
- quelques
- parler
- vitesse
- pivoté
- étapes
- Standard
- Encore
- stock
- STRONG
- de construction
- structurellement
- structure
- structures
- études
- Étude
- filiale
- Ces
- convient
- survivre
- combustion propre
- systémique
- Prenez
- prise
- équipe
- dire
- examiné
- Essais
- qui
- Les
- leur
- se
- puis
- Ces
- l'ont
- chose
- penser
- this
- cette année
- milliers
- trois
- Avec
- fiable
- calendrier
- à
- ensemble
- dit
- trop
- a
- outil
- qualifié
- essais cliniques
- Tourné
- deux
- subir
- non découvert
- Univers
- université
- d'utiliser
- en utilisant
- très
- attendez
- était
- Façon..
- we
- bienvenu
- ont été
- quand
- qui
- tout en
- blanc
- la totalité
- dont
- comprenant
- wong
- activités principales
- digne
- pourra
- an
- années
- hier
- encore
- a abouti
- you
- zéphyrnet
- zéro







