Incubación
Todos los procesos de incubación se llevaron a cabo en un ThermoMixer C (Eppendorf), excepto las Figs. 1 y 9 (Incubadora Dry Bath FB15103, Fisher Scientific) y para la Fig. 1c y las Figs suplementarias. 3–6 (QuantStudio5, Applied Biosystems de Thermo Fisher Scientific).
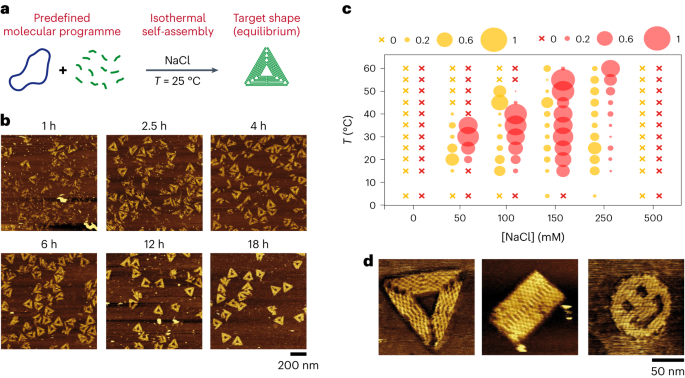
Autoensamblaje isotérmico de origamis de ADN 2D
Ver Fig. 1 y las Figs suplementarias. 1-9 y 11-14. Usamos el cóctel básico sin ningún pretratamiento térmico y lo mezclamos directamente con el tampón deseado antes de agitarlo brevemente y agregar la plantilla M13 (1 nM) a la solución y mezclar suavemente hacia arriba y hacia abajo con una pipeta. La solución se dejó incubar, sin mezclar más, a una temperatura fija durante el tiempo deseado.
Recocido térmico de origamis de ADN en tampón TANa
Consulte la figura complementaria. 10. Ensamblamos la plantilla M13 (1 nM) con una mezcla de grapas (40 nM cada grapa) en TANa suplementado con 100 mM de NaCl. La muestra se incubó durante 10 min a 90 °C y luego se sometió a una rampa térmica en un termociclador peqSTAR 2X (Peqlab) de 70 °C a 20 °C a una velocidad de -1 °C por 10 min.
Purificación por precipitación de PEG
Véanse las figuras complementarias. 13 y 14. Los origamis de ADN obtenidos por ensamblaje isotérmico en tampón TANa ([NaCl] = 100 mM) a 25 °C se purificaron a partir de sus hebras básicas mediante precipitación con PEG. El método se inspiró en el protocolo presentado en un informe anterior.35. Los origamis de ADN se diluyeron tres veces con una solución de PEG 8000 y NaCl para alcanzar concentraciones finales de 4 % p/v y 500 mM, respectivamente. Después de mezclar suavemente, la solución se dejó incubar durante 15 min a temperatura ambiente y se centrifugó a 15,000g durante 15 min. Se eliminó el sobrenadante y los origamis se resuspendieron a su volumen inicial en tampón TANa ([NaCl] = 100 mM). Si era necesario, se repetía el proceso para una segunda purificación consecutiva.
Electroforesis en gel de origamis purificados
Consulte la figura complementaria. 13. Preparamos 50 ml de gel de agarosa (tipo I bajo EEO, Sigma Aldrich) al 1.5% que contenía 4 μl de GR-Green 10,000x (Excellgen) en tampón TBE 1x. Una vez enfriado el gel, introducimos en cada pocillo 18 µl de DNA Ladder de 100 pb (New England Biolabs) o 18 µl de muestra suplementada con 1x de solución SDS de colorante de carga de ADN (Thermo Scientific). La migración se realizó a 100 V durante 1 h en una celda de electroforesis de 7 cm llena con tampón TBE 1X.
Montaje escalonado isotérmico
Consulte la figura complementaria. 24. Las grapas del triángulo se ensamblaron en tres lotes separados, cada uno codificando la esquina superior, la parte intermedia y el borde opuesto. Se mezcló un lote (40 nM de cada grapa) con M13 (1 nM) en tampón TANa ([NaCl] = 100 mM) y se dejó incubar el sistema a 25 °C sin mezclar más. Cada 24 h, retiramos el volumen necesario para la imagen AFM, agregamos un lote que codifica una parte adicional del triángulo (40 nM cada grapa) y dejamos que el sistema se incube a 25 °C en tampón TANa ([NaCl] = 100 mM) . Realizamos dos formas diferentes de montaje escalonado, de la esquina al lado opuesto y de un lado a la esquina opuesta.
Preparación isotérmica de triángulos modificados con estreptavidina
Ver Fig. 2a y las Figs suplementarias. 15 y 16. En el mismo tubo mezclamos 1 nM de M13, 40 nM de cada una de las grapas incluidas las biotiniladas y 2 µM de estreptavidina en tampón TANa suplementado con 100 mM de NaCl. La muestra se dejó incubar a 25 °C sin mezclar durante 24 h.
Preparación isotérmica de rectángulos SST R4
Ver Fig. 2b. Mezclamos todas las hebras del rectángulo R4 en el tampón hasta una concentración final de 100 nM en cada hebra en TANa complementado con NaCl 100 mM. La muestra se dejó incubar a 25 °C sin mezclar durante 24 h.
Electroforesis en gel de rectángulos SST R4
Ver Fig. 2b. Se preparó un gel de agarosa al 1.5 % (tipo I bajo EEO, Sigma Aldrich) en tampón TBE 0.5x suplementado con MgCl 11 mM.2 y tinción de ADN verde GB. La electroforesis en gel se realizó en un baño de agua con hielo durante 2 h a 100 V de voltaje utilizando una escalera de ADN de 1 kb. Para la purificación, la banda objetivo del gel se cortó en pedazos pequeños y se colocó en un tubo con una columna giratoria, y la columna se sometió a centrifugación a 5,000g durante 10 min. Para obtener imágenes de AFM, la muestra eluida se adsorbió directamente en una placa de mica durante 10 min dentro de la cámara de atmósfera controlada. A continuación, la muestra se enjuagó con 1 ml de TBE 0.5x + MgCl 11 mM.2 y observado usando AFM en 0.5× TBE + 11 mM MgCl2.
Preparación isotérmica de nanoredes de ADN
Ver Fig. 2c y Fig. Suplementaria. 17. Mezclamos los nueve oligonucleótidos (1 µM de cada nucleótido) en tampón TANa complementado con 100 mM o 150 mM de NaCl. La muestra se dejó incubar a 25 °C sin mezclar durante 24 h.
Recocido térmico de origamis 3D
Ver Fig. 3a y Fig. Suplementaria. 18. El andamiaje (7,560 nt M13 para Tb, 8,064 nt M13 para T1) y la mezcla de grapas (exceso de 10x en cada grapa) se mezclaron en un tampón que contenía Tris-HCl 5 mM, pH 8.0, EDTA 1 mM y MgCl 18 mM.2. La mezcla se calentó a 65 °C durante 15 min para desnaturalizar todas las cadenas de ADN antes de enfriarse lentamente en un gradiente de 60 °C a 40 °C, durante 41 h para recocer y ensamblar las nanoestructuras de origami 3D.
TEM de tinción negativa
Ver Fig. 3 y Fig. Suplementaria. 18. Para la caracterización de TEM, las nanoestructuras de ADN se purificaron primero a partir de agarosa al 1% (0.5x TBE, Tris-borato 45 mM, EDTA 1 mM, pH 8.3) complementado con MgCl 11 mM2 y 0.5 mg ml-1 Sybr SEGURO. Las muestras se migraron en el gel durante 3 h con un tampón de funcionamiento de 0.5x TBE, 11 mM MgCl2 a 2.85 Vcm-1 a temperatura ambiente. Las bandas correspondientes a las estructuras autoensambladas se escindieron y se transfirieron a una columna giratoria de extracción de ADN en gel (Merck) y se centrifugaron a 5,000 g durante 5 min a 4 °C. Luego, los origamis purificados se depositaron por adsorción en una rejilla recubierta de carbón descargada luminiscente (Quantifoil Micro Tools), se tiñeron durante 60 s con una solución acuosa de acetato de uranilo al 2% (p/v) (Merck) y luego se secaron con papel de filtro sin cenizas (VWR). ). Las observaciones TEM se realizaron en un microscopio JEM-1400 Flash Tungsten trabajando a 120 kV, equipado con una cámara Gatan OneView.
Competencia isotérmica
Consulte la figura complementaria. 20. Agitamos brevemente en vórtex las mezclas de dos conjuntos básicos que codifican triángulos y rectángulos (concentración final de 40 nM para cada elemento básico) con el tampón (TANa complementado con NaCl 100 mM) antes de agregar M13 a la mezcla (concentración final de 1 nM) y mezclar suavemente con una pipeta La muestra se dejó incubar a 25 °C sin mezclar más.
Observación AFM de nanoestructuras de ADN en líquido
Se utilizaron observaciones ambientales de AFM de alta resolución en el búfer de muestra para todos los datos e imágenes de AFM que se muestran en este artículo. Excepto por la Fig. 4 (consulte el protocolo específico a continuación), las nanoestructuras de ADN obtenidas en tampón TANa (origamis de ADN con o sin modificación de proteínas, rectángulos SST R4, nanoredes de ADN) se adsorbieron en discos de mica de 10 mm de diámetro recién cortados (Nano-Tec V-1 grado Moscovita, Micro to Nano Innovative Microscopy Supplies) previamente pegado a un disco metálico y tratado con 20 µl de una solución de tetracloruro de espermina (0.1 M en agua MilliQ) durante 10 min y lavado abundantemente, primero con agua MilliQ y luego con el tampón TANa. Para la adsorción de la muestra, se depositaron 15–20 µl de muestra sobre la mica recién tratada con espermina y se dejó adsorber durante 10 min, excepto en los experimentos de transformación isotérmica (Fig. 5 y las Figs suplementarias. 21–23) donde se aumentó el tiempo a 20 min debido a la menor concentración de origamis. A continuación, la placa de mica se enjuagó suavemente con 200 µl del tampón para eliminar el exceso de grapas y objetos no adsorbidos. Para evitar que la muestra se seque durante la manipulación de la mica, dejamos una fina capa de tampón encima de la muestra adsorbida y la mantuvimos a temperatura ambiente en una cámara de atmósfera controlada (un recipiente sellado que contenía un trozo de toallita Kimtech humedecida con MilliQ agua). Se realizó el mismo protocolo con muestras que contenían magnesio (Fig. 1, tampones TAEMg y TAMg), excepto que las muestras se adsorbieron directamente durante 5 min en la mica recién escindida sin ningún tratamiento. Las muestras se observaron con un microscopio de fuerza atómica Cypher ES (Oxford Instruments) en modo tapping con una frecuencia de resonancia de 17–45 kHz en líquido y una luz de 0.09 N·m.-1 Punta de fuerza constante (BL-AC40TS, Olympus), utilizando el modo de excitación fototérmica blueDrive. Las imágenes sin procesar se sometieron a sustracción de fondo polinomial, corrección de nivel de plano, alineación de filas utilizando varios métodos y corrección de cicatriz horizontal en Gwyddion.
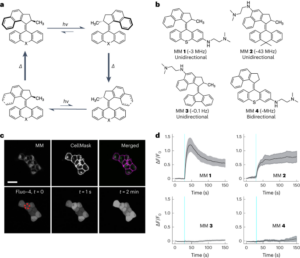
Imágenes en tiempo real de la evolución isotérmica Λ → Δ en una superficie de bicapa lipídica
Ver Fig. 4, Texto Suplementario 3 y películas complementarias 1-4. Las bicapas lipídicas soportadas (SLB) se obtuvieron a partir de liposomas DOPC a través de nuestro método descrito anteriormente37. Se prepararon vesículas a partir de una reserva de cloroformo de DOPC. Después de la evaporación del cloroformo bajo una corriente de gas nitrógeno, los lípidos se rehidrataron en agua MilliQ para alcanzar una concentración final de lípidos de 2 mg ml-1. A continuación, la mezcla de lípidos se agitó en un vórtex y se sometió a ultrasonidos durante 60 min para producir pequeñas vesículas unilamelares. Para evitar el secado de las bicapas, se llevaron a cabo los siguientes pasos en una cámara de atmósfera controlada. Los SLB se formaron depositando 2 ml de la solución de vesículas en discos de mica recién cortados (previamente pegados en una placa metálica magnética con pegamento), seguidos de 2 µl de TAEMg (Tris–acetato 1×, [EDTA] = 1 mM, [MgCl2] = 12.5 mM). Después de 30 min de adsorción, la muestra se enjuagó con 2 µl de tampón TAEMg para eliminar los liposomas no adsorbidos y este proceso de adsorción se repitió una segunda vez para asegurar una cobertura óptima de la superficie de mica por la bicapa. Al final del proceso de adsorción, la muestra se enjuagó con 5 µl de TAENa (Tris–acetato 1×, [EDTA] = 1 mM, [NaCl] = 100 mM) para asegurar que no quedara Mg libre.2+ iones en la muestra, lo que evitaría el plegamiento isotérmico de los origamis.
Origamis en forma de Λ modificados con colesterol (Fig. 19) se prepararon mezclando una solución de 10 nM de M13 en el tampón TAENa con 20 nM de cada una de las grapas y recociendo reduciendo la temperatura de 70 °C a 4 °C a una velocidad de -0.1 °C min-1. La solución de origami resultante se usó sin más purificación. Luego, se depositaron 2 µl de la solución de origamis Λ modificada con colesterol sobre el SLB preformado, seguidos de 2 µl de tampón TAENa. La muestra se incubó durante 60 min a temperatura ambiente en la cámara de atmósfera controlada, y luego se tomaron imágenes de la superficie directamente a temperatura ambiente (T = 26 °C) por AFM en 20 µl de tampón TAENa sin enjuagar la superficie. Después de seleccionar una posición que contenía un número suficiente de origamis Λ adsorbidos en el SLB, se agregaron 8 µl de las grapas del lado A en TAENa a la muestra sin moverla ni sacarla de la etapa AFM. Luego, la solución que sobresalía de la muestra se mezcló suavemente moviendo lentamente la sonda AFM hacia arriba y hacia abajo varias veces para acelerar la difusión en las grapas del lado A hacia la superficie de la mica. Luego se escaneó la misma posición en promedio cada 3 min durante 223 min. La resolución de la imagen fue de 512 × 512 de t = 0 a t = 41 min y luego se cambió a 640 × 640. El z escala de las imágenes fue de 0 a 10 nm de t = 0 a t = 47 min y 0-7 nm después. Debido a la evaporación durante el proceso de formación de imágenes, se añadió tampón suplementario (8 µl) en t = 63 minutos, t = 144 minutos y t = 161 min, y además se añadieron 8 µl de las grapas del lado A después t = 170 min.
Las imágenes AFM se obtuvieron en TAENa a temperatura ambiente usando un microscopio de fuerza atómica Brücker Fast Scan en modo tapping. Todos los experimentos de AFM se realizaron utilizando sondas Olympus. Las imágenes obtenidas por este protocolo se muestran en la Fig. 4 y películas complementarias 1–4.
Transformación morfológica isotérmica
Ver Fig. 5 y las Figs suplementarias. 21-23. Los origamis rectangulares de ADN, sin o con grapas acortadas, se prepararon primero mediante hibridación térmica: después del ensamblaje de 1 nM de la plantilla M13 con una mezcla de grapas (40 nM cada una) en tampón TANa (con NaCl 100 mM o 150 mM), el la muestra se incubó durante 10 min a 90 °C y luego se sometió a una rampa térmica en un termociclador peqSTAR 2X (Peqlab) de 70 °C a 20 °C a una velocidad de -0.1 °C por 2 min. Luego, las grapas triangulares se mezclaron directamente con la muestra, sin ninguna purificación (las grapas rectangulares se mantienen en el sistema), con una pipeta a la concentración deseada (10 o 100 nM de cada grapa) con una concentración final de 0.25 nM en [M13] y 10 nM en cada grapa rectangular. La muestra resultante se dejó incubar a 25 °C o 30 °C sin mezclar más.
Estadística y reproducibilidad
Excepto por el experimento que se muestra en la Fig. 4, que solo se realizó una vez debido a la complejidad de la configuración, todas las demás investigaciones se replicaron varias veces para garantizar la reproducibilidad. Todos los análisis de imágenes AFM se realizaron en un gran número n de origamis individuales tomados de diferentes imágenes, en diferentes posiciones de muestras obtenidas en las mismas condiciones. Se analizaron todos los origamis detectados. Ningún origami, bien doblado o no, fue excluido de estos análisis. El número n de objetos analizados en cada condición que se muestra en las diferentes figuras se muestra en las tablas complementarias 1–5. No se utilizó ningún método estadístico para predeterminar el tamaño de la muestra. No se excluyeron datos de los análisis. Los experimentos no fueron aleatorios. Los investigadores no estaban cegados a la asignación durante los experimentos y la evaluación de los resultados.
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Automoción / vehículos eléctricos, Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- Desplazamientos de bloque. Modernización de la propiedad de compensaciones ambientales. Accede Aquí.
- Fuente: https://www.nature.com/articles/s41565-023-01468-2
- :es
- :no
- :dónde
- $ UP
- 1
- 10
- 100
- 11
- 12
- 15%
- 2%
- 20
- 200
- 2014
- 2015
- 24
- 25
- 26
- 2D
- 30
- 3d
- 40
- 50
- 500
- 60
- 65
- 7
- 70
- 8
- 90
- a
- acelerar
- adicional
- la adición de
- adición
- Adicionales
- Después
- después
- Todos
- asignación
- cantidad
- an
- Comercial
- y
- cualquier
- aplicada
- somos
- artículo
- ensamblada
- Asamblea
- evaluación
- At
- promedio
- fondo
- BANDA
- BE
- porque
- "Ser"
- a continuación
- BP
- brevemente
- buffer
- by
- cámara
- llevado a
- (SCD por sus siglas en inglés),
- Cámara
- cambiado
- clic
- cóctel
- Codificación
- Columna
- complejidad
- concentración
- condición
- condiciones
- consecutivo
- constante
- Envase
- Esquina
- Correspondiente
- cobertura
- Corte
- cifrar
- datos
- denso
- depositado
- descrito
- deseado
- detectado
- una experiencia diferente
- Difusión
- directamente
- aquí
- ADN
- hecho
- DE INSCRIPCIÓN
- secas
- dos
- durante
- e
- cada una
- ed
- Southern Implants
- final
- England
- garantizar
- equipado
- Éter (ETH)
- Cada
- evolución
- Excepto
- exceso
- excluidos
- experimento
- experimentos
- RÁPIDO
- Higo
- Figura
- Figuras
- lleno
- filtrar
- final
- Nombre
- fijas
- Flash
- seguido
- siguiendo
- FORCE
- formado
- Gratis
- Frecuencia
- Desde
- promover
- Además
- GAS
- suaves
- grado
- Verde
- Cuadrícula
- tenido
- de alta resolución
- Horizontal
- HTTPS
- i
- if
- imagen
- imágenes
- Proyección de imagen
- in
- Incluye
- aumentado
- incubado
- Incubación
- incubadora
- INSTRUMENTO individual
- inicial
- originales
- dentro
- inspirado
- instrumentos
- Intermedio
- dentro
- Introducido
- Investigaciones
- Los investigadores
- IT
- mantenido
- ladder
- large
- .
- izquierda
- LINK
- Líquido
- carga
- Lote
- Baja
- inferior
- Manipulación
- Martin
- materiales
- Merck
- metal
- Método
- métodos
- Mica
- micro
- Microscopio
- Microscopía
- migrado
- migración
- min
- mezcla
- mezclado
- Mezcla
- mezcla
- ML
- Moda
- Películas
- emocionante
- nano
- nanotecnología
- Naturaleza
- necesario
- Nuevo
- no
- número
- objetos
- observado
- obtenido
- of
- Olimpo
- on
- una vez
- ONE
- las
- , solamente
- opuesto
- óptimo
- or
- Otro
- "nuestr
- salir
- Resultado
- Más de
- Oxford
- Papel
- parte
- clavija
- para
- realizado
- pieza
- piezas
- Platón
- Inteligencia de datos de Platón
- PlatónDatos
- posición
- abiertas
- preparación
- preparado
- evitar
- anterior
- previamente
- Anterior
- sonda
- en costes
- producir
- correctamente
- Proteínas
- protocolo
- poner
- Rampa
- Aleatorizado
- Rate
- Crudo
- en comunicarse
- la reducción de
- restante
- remove
- Remoto
- la eliminación de
- repetido
- replicado
- Resolución
- resonancia
- respectivamente
- resultante
- Conferencia
- FILA
- correr
- s
- ambiente seguro
- mismo
- escalable
- Escala
- escanear
- científico
- Segundo
- ver
- seleccionar
- separado
- Sets
- Varios
- acortado
- mostrado
- lado
- Sigma
- Tamaño
- Despacio
- chica
- a medida
- Soluciones
- soluciones y
- Girar
- Etapa
- estadístico
- pasos
- en stock
- hebras
- stream
- suficiente
- Soportado
- Superficie
- te
- T
- T1
- toma
- tocando
- Target
- plantilla
- esa
- La
- su
- luego
- Ahí.
- térmico
- Estas
- así
- Tres
- equipo
- veces
- de estilista
- a
- parte superior
- hacia
- transferido
- tratados
- del mismo día
- dos
- tipo
- bajo
- usado
- usando
- diversos
- vía
- Tensión
- volumen
- fue
- Agua
- formas
- we
- WELL
- tuvieron
- que
- seguirá
- limpiar
- sin
- trabajando
- se
- zephyrnet