16. Mai 2023 (Nanowerk-Neuigkeiten) Eine neue Studie trägt zu einem völlig neuen Bild darüber bei, wie Bakterienzellen kontinuierlich fehlerhafte Abschnitte ihrer DNA reparieren. In der Zeitschrift veröffentlicht Zelle („RNA-Polymerase treibt die DNA-Reparatur durch Ribonukleotid-Exzision voran E. coli") beschreibt der Bericht den molekularen Mechanismus hinter einem DNA-Reparaturweg, der dem irrtümlichen Einbau einer bestimmten Art molekularer Bausteine, Ribonukleotiden, in genetische Codes entgegenwirkt. Solche Fehler kommen beim Code-Kopieren in Bakterien und anderen Organismen häufig vor. Da ein falscher Einbau von Ribonukleotiden zu schädlichen Veränderungen des DNA-Codes (Mutationen) und DNA-Brüchen führen kann, haben alle Organismen einen DNA-Reparaturweg namens Ribonukleotid-Exzisionsreparatur (RER) entwickelt, der solche Fehler schnell behebt.
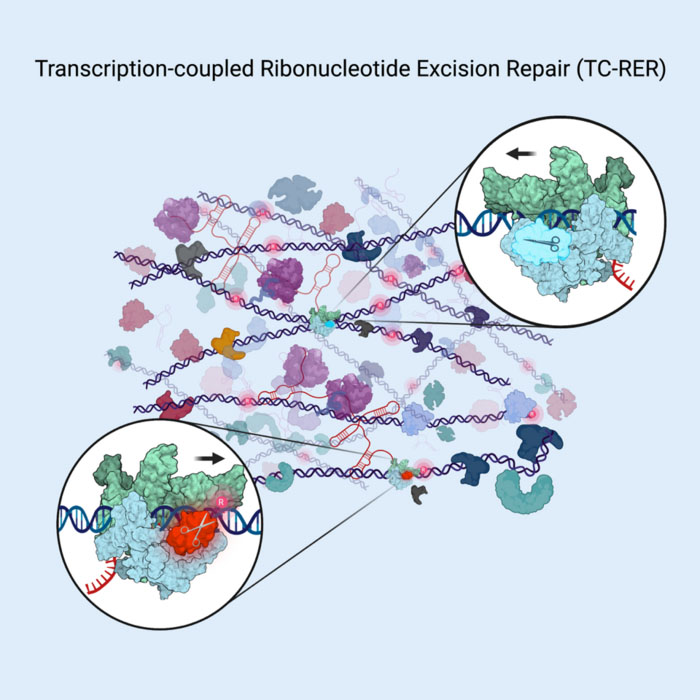 Das Studienenzym RNAseHII repariert DNA, indem es auf dem Enzym RNA-Polymerase mitfährt, das den genetischen Code liest, und falsch platzierte Codebuchstaben herausschneidet (siehe Schere), wenn es sie im genetischen Material von Bakterien „sieht“. (© Cell Press) Letztes Jahr veröffentlichte ein Team unter der Leitung von Evgeny Nudler, PhD, Julie Wilson Anderson Professor in der Abteilung für Biochemie und Molekulare Pharmakologie an der NYU Langone Health, zwei Analysen der DNA-Reparatur bei Lebewesen E. coli Zellen. Sie fanden heraus, dass die meisten Reparaturen bestimmter Arten von DNA-Schäden (große Läsionen), wie sie beispielsweise durch UV-Strahlung verursacht werden, dadurch erfolgen können, dass beschädigte Codeabschnitte zunächst von einer Proteinmaschine namens RNA-Polymerase identifiziert wurden. Die RNA-Polymerase bewegt sich entlang der DNA-Kette und liest den Code der DNA-„Buchstaben“, während sie Anweisungen in RNA-Moleküle umschreibt, die dann den Proteinaufbau steuern. Nudler und Mitarbeiter fanden heraus, dass die RNA-Polymerase während dieses Transkriptionsprozesses auch DNA-Läsionen findet und dann als Plattform für den Aufbau einer DNA-Reparaturmaschine namens Nucleotide Excision Repair (NER)-Komplex dient. NER schneidet dann die gefundene fehlerhafte DNA heraus und ersetzt sie durch eine genaue Kopie. Ohne die Wirkung der RNA-Polymerase tritt in lebenden Bakterien, wenn überhaupt, nur wenig NER auf. Jetzt liefert die neue Studie in Cell den ersten Beweis dafür, dass RER, wie im NER-Signalweg, eng an die Transkription gekoppelt ist. Die Autoren der Studie fanden Hinweise darauf, dass das an RER beteiligte Schlüsselenzym, RNaseHII, auch mit der RNA-Polymerase zusammenarbeitet, während es nach falsch eingebauten Ribonukleotiden in den DNA-Ketten lebender Bakterienzellen sucht. „Unsere Ergebnisse regen weiterhin zum Überdenken bestimmter Grundprinzipien im Bereich der DNA-Reparatur an“, sagt Nudler, ebenfalls Forscher am Howard Hughes Medical Institute. „Für die Zukunft plant unser Team zu untersuchen, ob die RNA-Polymerase die DNA auf alle Arten von Problemen scannt und genomweite Reparaturen auslöst, nicht nur in Bakterien, sondern auch in menschlichen Zellen.“
Das Studienenzym RNAseHII repariert DNA, indem es auf dem Enzym RNA-Polymerase mitfährt, das den genetischen Code liest, und falsch platzierte Codebuchstaben herausschneidet (siehe Schere), wenn es sie im genetischen Material von Bakterien „sieht“. (© Cell Press) Letztes Jahr veröffentlichte ein Team unter der Leitung von Evgeny Nudler, PhD, Julie Wilson Anderson Professor in der Abteilung für Biochemie und Molekulare Pharmakologie an der NYU Langone Health, zwei Analysen der DNA-Reparatur bei Lebewesen E. coli Zellen. Sie fanden heraus, dass die meisten Reparaturen bestimmter Arten von DNA-Schäden (große Läsionen), wie sie beispielsweise durch UV-Strahlung verursacht werden, dadurch erfolgen können, dass beschädigte Codeabschnitte zunächst von einer Proteinmaschine namens RNA-Polymerase identifiziert wurden. Die RNA-Polymerase bewegt sich entlang der DNA-Kette und liest den Code der DNA-„Buchstaben“, während sie Anweisungen in RNA-Moleküle umschreibt, die dann den Proteinaufbau steuern. Nudler und Mitarbeiter fanden heraus, dass die RNA-Polymerase während dieses Transkriptionsprozesses auch DNA-Läsionen findet und dann als Plattform für den Aufbau einer DNA-Reparaturmaschine namens Nucleotide Excision Repair (NER)-Komplex dient. NER schneidet dann die gefundene fehlerhafte DNA heraus und ersetzt sie durch eine genaue Kopie. Ohne die Wirkung der RNA-Polymerase tritt in lebenden Bakterien, wenn überhaupt, nur wenig NER auf. Jetzt liefert die neue Studie in Cell den ersten Beweis dafür, dass RER, wie im NER-Signalweg, eng an die Transkription gekoppelt ist. Die Autoren der Studie fanden Hinweise darauf, dass das an RER beteiligte Schlüsselenzym, RNaseHII, auch mit der RNA-Polymerase zusammenarbeitet, während es nach falsch eingebauten Ribonukleotiden in den DNA-Ketten lebender Bakterienzellen sucht. „Unsere Ergebnisse regen weiterhin zum Überdenken bestimmter Grundprinzipien im Bereich der DNA-Reparatur an“, sagt Nudler, ebenfalls Forscher am Howard Hughes Medical Institute. „Für die Zukunft plant unser Team zu untersuchen, ob die RNA-Polymerase die DNA auf alle Arten von Problemen scannt und genomweite Reparaturen auslöst, nicht nur in Bakterien, sondern auch in menschlichen Zellen.“
 Das Studienenzym RNAseHII repariert DNA, indem es auf dem Enzym RNA-Polymerase mitfährt, das den genetischen Code liest, und falsch platzierte Codebuchstaben herausschneidet (siehe Schere), wenn es sie im genetischen Material von Bakterien „sieht“. (© Cell Press) Letztes Jahr veröffentlichte ein Team unter der Leitung von Evgeny Nudler, PhD, Julie Wilson Anderson Professor in der Abteilung für Biochemie und Molekulare Pharmakologie an der NYU Langone Health, zwei Analysen der DNA-Reparatur bei Lebewesen E. coli Zellen. Sie fanden heraus, dass die meisten Reparaturen bestimmter Arten von DNA-Schäden (große Läsionen), wie sie beispielsweise durch UV-Strahlung verursacht werden, dadurch erfolgen können, dass beschädigte Codeabschnitte zunächst von einer Proteinmaschine namens RNA-Polymerase identifiziert wurden. Die RNA-Polymerase bewegt sich entlang der DNA-Kette und liest den Code der DNA-„Buchstaben“, während sie Anweisungen in RNA-Moleküle umschreibt, die dann den Proteinaufbau steuern. Nudler und Mitarbeiter fanden heraus, dass die RNA-Polymerase während dieses Transkriptionsprozesses auch DNA-Läsionen findet und dann als Plattform für den Aufbau einer DNA-Reparaturmaschine namens Nucleotide Excision Repair (NER)-Komplex dient. NER schneidet dann die gefundene fehlerhafte DNA heraus und ersetzt sie durch eine genaue Kopie. Ohne die Wirkung der RNA-Polymerase tritt in lebenden Bakterien, wenn überhaupt, nur wenig NER auf. Jetzt liefert die neue Studie in Cell den ersten Beweis dafür, dass RER, wie im NER-Signalweg, eng an die Transkription gekoppelt ist. Die Autoren der Studie fanden Hinweise darauf, dass das an RER beteiligte Schlüsselenzym, RNaseHII, auch mit der RNA-Polymerase zusammenarbeitet, während es nach falsch eingebauten Ribonukleotiden in den DNA-Ketten lebender Bakterienzellen sucht. „Unsere Ergebnisse regen weiterhin zum Überdenken bestimmter Grundprinzipien im Bereich der DNA-Reparatur an“, sagt Nudler, ebenfalls Forscher am Howard Hughes Medical Institute. „Für die Zukunft plant unser Team zu untersuchen, ob die RNA-Polymerase die DNA auf alle Arten von Problemen scannt und genomweite Reparaturen auslöst, nicht nur in Bakterien, sondern auch in menschlichen Zellen.“
Das Studienenzym RNAseHII repariert DNA, indem es auf dem Enzym RNA-Polymerase mitfährt, das den genetischen Code liest, und falsch platzierte Codebuchstaben herausschneidet (siehe Schere), wenn es sie im genetischen Material von Bakterien „sieht“. (© Cell Press) Letztes Jahr veröffentlichte ein Team unter der Leitung von Evgeny Nudler, PhD, Julie Wilson Anderson Professor in der Abteilung für Biochemie und Molekulare Pharmakologie an der NYU Langone Health, zwei Analysen der DNA-Reparatur bei Lebewesen E. coli Zellen. Sie fanden heraus, dass die meisten Reparaturen bestimmter Arten von DNA-Schäden (große Läsionen), wie sie beispielsweise durch UV-Strahlung verursacht werden, dadurch erfolgen können, dass beschädigte Codeabschnitte zunächst von einer Proteinmaschine namens RNA-Polymerase identifiziert wurden. Die RNA-Polymerase bewegt sich entlang der DNA-Kette und liest den Code der DNA-„Buchstaben“, während sie Anweisungen in RNA-Moleküle umschreibt, die dann den Proteinaufbau steuern. Nudler und Mitarbeiter fanden heraus, dass die RNA-Polymerase während dieses Transkriptionsprozesses auch DNA-Läsionen findet und dann als Plattform für den Aufbau einer DNA-Reparaturmaschine namens Nucleotide Excision Repair (NER)-Komplex dient. NER schneidet dann die gefundene fehlerhafte DNA heraus und ersetzt sie durch eine genaue Kopie. Ohne die Wirkung der RNA-Polymerase tritt in lebenden Bakterien, wenn überhaupt, nur wenig NER auf. Jetzt liefert die neue Studie in Cell den ersten Beweis dafür, dass RER, wie im NER-Signalweg, eng an die Transkription gekoppelt ist. Die Autoren der Studie fanden Hinweise darauf, dass das an RER beteiligte Schlüsselenzym, RNaseHII, auch mit der RNA-Polymerase zusammenarbeitet, während es nach falsch eingebauten Ribonukleotiden in den DNA-Ketten lebender Bakterienzellen sucht. „Unsere Ergebnisse regen weiterhin zum Überdenken bestimmter Grundprinzipien im Bereich der DNA-Reparatur an“, sagt Nudler, ebenfalls Forscher am Howard Hughes Medical Institute. „Für die Zukunft plant unser Team zu untersuchen, ob die RNA-Polymerase die DNA auf alle Arten von Problemen scannt und genomweite Reparaturen auslöst, nicht nur in Bakterien, sondern auch in menschlichen Zellen.“
Modernste Techniken
Ribonukleotide (die Bausteine der RNA) und Desoxyribonukleotide (DNA-Bestandteile) sind verwandte Verbindungen. Während Zellen DNA-Ketten in Bakterienzellen kopieren und aufbauen, bauen sie oft fälschlicherweise Ribonukleotide anstelle von Desoxyribonukleotiden in DNA-Ketten ein, weil sie sich nur durch ein einziges Sauerstoffatom unterscheiden, sagen die Autoren der Studie. In Bakterienzellen macht die DNA-Polymerase III bekanntermaßen jedes Mal etwa 2,000 dieser Fehler, wenn sie das genetische Material einer Zelle kopiert. Um die Integrität des Genoms aufrechtzuerhalten, wird der Großteil der fehlplatzierten Ribonukleotide über den RER-Weg entfernt. Eine wichtige Frage war jedoch, wie RNaseHII relativ seltene Ribonukleotidläsionen inmitten eines „Ozeans“ intakter zellulärer DNA-Codes so schnell findet. Wie bereits in ihren Studien im Jahr 2022 verwendeten die Forscher quantitative Massenspektrometrie und In-vivo-Protein-Protein-Vernetzung, um die Abstände zwischen chemisch verbundenen Proteinen abzubilden und so die Schlüsseloberflächen von RNaseHII und RNA-Polymerase zu bestimmen, während sie in lebenden Bakterienzellen interagieren. Auf diese Weise stellten sie fest, dass die meisten RNaseHII-Moleküle mit der RNA-Polymerase koppeln. Darüber hinaus nutzten sie kryogene Elektronenmikroskopie (CryoEM), um die hochauflösenden Strukturen von an RNA-Polymerase gebundenem RNaseHII zu erfassen und so die Protein-Protein-Wechselwirkungen aufzudecken, die den RER-Komplex definieren. Darüber hinaus beeinträchtigten strukturgesteuerte genetische Experimente, die die RNA-Polymerase/RNaseHII-Wechselwirkung schwächten, die RER. „Diese Arbeit unterstützt ein Modell, bei dem RNaseHII die DNA nach falsch platzierten Ribonukleotiden durchsucht, indem es auf der RNA-Polymerase reitet, während sie sich entlang der DNA bewegt“, sagt der Erstautor der Studie, Zhitai Hao, ein Postdoktorand in Nudlers Labor. „Diese Arbeit ist für unser grundlegendes Verständnis des DNA-Reparaturprozesses von entscheidender Bedeutung und hat weitreichende klinische Auswirkungen.“- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoAiStream. Web3-Datenintelligenz. Wissen verstärkt. Hier zugreifen.
- Die Zukunft prägen mit Adryenn Ashley. Hier zugreifen.
- Kaufen und verkaufen Sie Anteile an PRE-IPO-Unternehmen mit PREIPO®. Hier zugreifen.
- Quelle: https://www.nanowerk.com/news2/biotech/newsid=63002.php
- :hast
- :Ist
- :nicht
- :Wo
- 000
- 10
- 11
- 12
- 2022
- 2023
- 7
- 9
- a
- Über Uns
- genau
- Action
- Zusatz
- Fügt
- Alle
- entlang
- ebenfalls
- inmitten
- an
- und
- jedem
- SIND
- AS
- Versammlung
- At
- Atom
- Autor
- Autoren
- Bakterien
- basic
- weil
- war
- hinter
- zwischen
- Blockieren
- Blockiert
- gebunden
- geht kaputt
- bauen
- Building
- aber
- by
- namens
- CAN
- Erfassung
- verursacht
- Die Zellen
- Center
- sicher
- Kette
- Ketten
- Änderungen
- Klinische
- Code
- Komplex
- Komponenten
- Kompromittiert
- ständig
- fortsetzen
- Kopien
- Zähler
- Paar
- gekoppelt
- kryogen
- Schneiden
- Datum
- Abteilung
- entschlossen
- DID
- abweichen
- Direkt
- Frau
- nach unten
- im
- Edge
- aufstrebenden
- Fehler
- Jedes
- Beweis
- entwickelt
- Experimente
- weit reichend
- fehlerhaft
- Feld
- findet
- Vorname
- Aussichten für
- vorwärts
- gefunden
- häufig
- weiter
- Genom
- gegeben
- hätten
- Haben
- Gesundheit
- hochauflösenden
- Ultraschall
- HTTPS
- human
- identifiziert
- if
- Image
- Auswirkungen
- in
- Aufnahme
- integrieren
- inspirieren
- Institut
- Anleitung
- Integrität
- interagieren
- Interaktion
- Interaktionen
- in
- untersuchen
- beteiligt
- IT
- Zeitschrift
- jpg
- Wesentliche
- bekannt
- Labor
- Nachname
- Letztes Jahr
- geführt
- Gefällt mir
- verknüpft
- wenig
- Leben
- Maschine
- halten
- um
- Karte
- Masse
- Ihres Materials
- Mechanismus
- sowie medizinische
- Mikroskopie
- Mitte
- unangebracht
- Fehler
- Modell
- Molekular-
- vor allem warme
- Motoren
- bewegt sich
- Neu
- jetzt an
- NYU
- of
- vorgenommen,
- on
- einzige
- Andere
- UNSERE
- Sauerstoff
- ein Bild
- Ort
- Pläne
- Plattform
- Plato
- Datenintelligenz von Plato
- PlatoData
- Presse
- Grundsätze
- Probleme
- Prozessdefinierung
- Professor
- Proteine
- Proteine
- bietet
- veröffentlicht
- quantitativ
- Frage
- schnell
- radikal
- RARE
- Lesebrillen
- bezogene
- verhältnismäßig
- Entfernt
- Knorpel zu reparieren,
- berichten
- Forscher
- Folge
- Die Ergebnisse
- zeigen
- Reiten
- RNA
- sagt
- Abschnitte
- sehen
- dient
- Single
- So
- Es wurden Studien
- Studie
- so
- Unterstützt
- Team
- zur Verbesserung der Gesundheitsgerechtigkeit
- Das
- ihr
- Sie
- dann
- Diese
- vom Nutzer definierten
- fehlen uns die Worte.
- diejenigen
- dicht
- Zeit
- zu
- XNUMX
- tippe
- Typen
- Verständnis
- benutzt
- lebenswichtig
- vivo
- Weg..
- GUT
- wann
- ob
- welche
- während
- Wilson
- mit
- ohne
- Arbeiten
- Jahr
- Zephyrnet