16 maggio 2023 (Notizie Nanowerk) Un nuovo studio si aggiunge a un quadro emergente e radicalmente nuovo di come le cellule batteriche riparano continuamente sezioni difettose del loro DNA. Pubblicato sulla rivista Cella (“RNA Polymerase Drives Ribonucleotide Excision DNA Repair in E. coli"), la relazione descrive il meccanismo molecolare alla base di un percorso di riparazione del DNA che contrasta l'errata inclusione di un certo tipo di elementi costitutivi molecolari, i ribonucleotidi, nei codici genetici. Tali errori sono frequenti nel processo di copia del codice nei batteri e in altri organismi. Dato che l’incorporazione errata del ribonucleotide può provocare cambiamenti dannosi del codice del DNA (mutazioni) e rotture del DNA, tutti gli organismi si sono evoluti per avere un percorso di riparazione del DNA chiamato riparazione per escissione del ribonucleotide (RER) che corregge rapidamente tali errori.
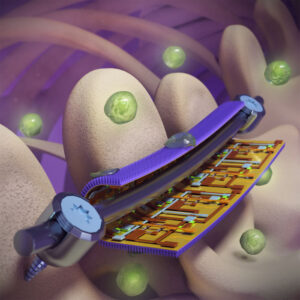
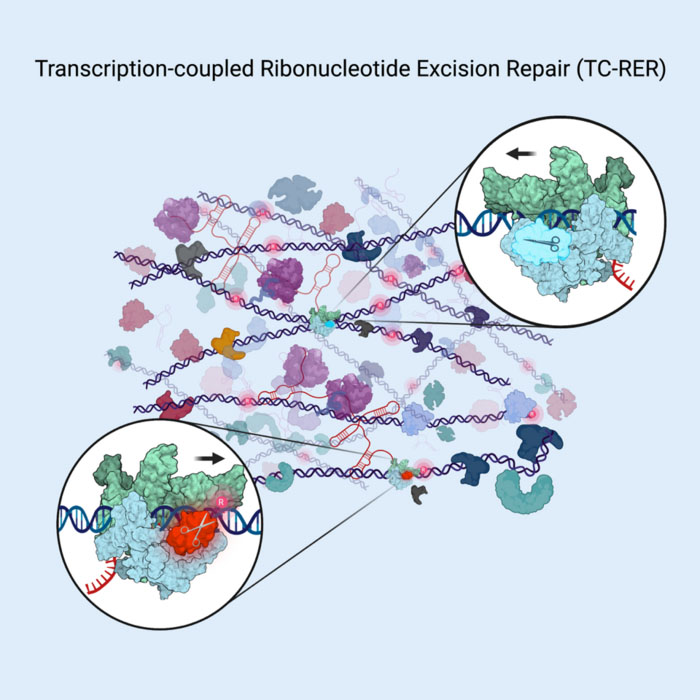 L'enzima in studio RNAseHII ripara il DNA cavalcando l'enzima che legge il codice genetico, l'RNA polimerasi, e ritagliando (vedi forbici) le lettere del codice fuori posto quando le "vede" nel materiale genetico batterico. (© Cell Press) L'anno scorso un team guidato da Evgeny Nudler, PhD, professore Julie Wilson Anderson presso il Dipartimento di Biochimica e Farmacologia Molecolare della NYU Langone Health, ha pubblicato due analisi sulla riparazione del DNA negli organismi viventi E. coli cellule. Hanno scoperto che la maggior parte della riparazione di alcuni tipi di danni al DNA (lesioni voluminose), come quelli causati dall’irradiazione UV, può avvenire perché le sezioni del codice danneggiate sono state prima identificate da una macchina proteica chiamata RNA polimerasi. La RNA polimerasi si muove lungo la catena del DNA, leggendo il codice delle “lettere” del DNA mentre trascrive le istruzioni nelle molecole di RNA, che poi dirigono la costruzione delle proteine. Nudler e colleghi hanno scoperto che durante questo processo di trascrizione, l'RNA polimerasi trova anche lesioni del DNA e quindi funge da piattaforma per l'assemblaggio di una macchina di riparazione del DNA chiamata complesso di riparazione per escissione nucleotidica (NER). Il NER quindi elimina il DNA difettoso trovato e lo sostituisce con una copia accurata. Senza l’azione della RNA polimerasi, nei batteri viventi è presente una quantità minima di NER, se non addirittura nulla. Ora il nuovo studio su Cell fornisce la prima prova che, come nel percorso NER, il RER è strettamente legato alla trascrizione. Gli autori dello studio hanno trovato prove che l’enzima chiave coinvolto nel RER, RNaseHII, coopera anche con l’RNA polimerasi mentre ricerca ribonucleotidi incorporati erroneamente nelle catene di DNA delle cellule batteriche viventi. "I nostri risultati continuano a ispirare un ripensamento di alcuni principi di base nel campo della riparazione del DNA", afferma Nudler, anche lui ricercatore presso l'Howard Hughes Medical Institute. “Andando avanti, il nostro team prevede di indagare se l’RNA polimerasi scansiona il DNA per tutti i tipi di problemi e innesca la riparazione a livello dell’intero genoma, non solo nei batteri, ma anche nelle cellule umane”.
L'enzima in studio RNAseHII ripara il DNA cavalcando l'enzima che legge il codice genetico, l'RNA polimerasi, e ritagliando (vedi forbici) le lettere del codice fuori posto quando le "vede" nel materiale genetico batterico. (© Cell Press) L'anno scorso un team guidato da Evgeny Nudler, PhD, professore Julie Wilson Anderson presso il Dipartimento di Biochimica e Farmacologia Molecolare della NYU Langone Health, ha pubblicato due analisi sulla riparazione del DNA negli organismi viventi E. coli cellule. Hanno scoperto che la maggior parte della riparazione di alcuni tipi di danni al DNA (lesioni voluminose), come quelli causati dall’irradiazione UV, può avvenire perché le sezioni del codice danneggiate sono state prima identificate da una macchina proteica chiamata RNA polimerasi. La RNA polimerasi si muove lungo la catena del DNA, leggendo il codice delle “lettere” del DNA mentre trascrive le istruzioni nelle molecole di RNA, che poi dirigono la costruzione delle proteine. Nudler e colleghi hanno scoperto che durante questo processo di trascrizione, l'RNA polimerasi trova anche lesioni del DNA e quindi funge da piattaforma per l'assemblaggio di una macchina di riparazione del DNA chiamata complesso di riparazione per escissione nucleotidica (NER). Il NER quindi elimina il DNA difettoso trovato e lo sostituisce con una copia accurata. Senza l’azione della RNA polimerasi, nei batteri viventi è presente una quantità minima di NER, se non addirittura nulla. Ora il nuovo studio su Cell fornisce la prima prova che, come nel percorso NER, il RER è strettamente legato alla trascrizione. Gli autori dello studio hanno trovato prove che l’enzima chiave coinvolto nel RER, RNaseHII, coopera anche con l’RNA polimerasi mentre ricerca ribonucleotidi incorporati erroneamente nelle catene di DNA delle cellule batteriche viventi. "I nostri risultati continuano a ispirare un ripensamento di alcuni principi di base nel campo della riparazione del DNA", afferma Nudler, anche lui ricercatore presso l'Howard Hughes Medical Institute. “Andando avanti, il nostro team prevede di indagare se l’RNA polimerasi scansiona il DNA per tutti i tipi di problemi e innesca la riparazione a livello dell’intero genoma, non solo nei batteri, ma anche nelle cellule umane”.
 L'enzima in studio RNAseHII ripara il DNA cavalcando l'enzima che legge il codice genetico, l'RNA polimerasi, e ritagliando (vedi forbici) le lettere del codice fuori posto quando le "vede" nel materiale genetico batterico. (© Cell Press) L'anno scorso un team guidato da Evgeny Nudler, PhD, professore Julie Wilson Anderson presso il Dipartimento di Biochimica e Farmacologia Molecolare della NYU Langone Health, ha pubblicato due analisi sulla riparazione del DNA negli organismi viventi E. coli cellule. Hanno scoperto che la maggior parte della riparazione di alcuni tipi di danni al DNA (lesioni voluminose), come quelli causati dall’irradiazione UV, può avvenire perché le sezioni del codice danneggiate sono state prima identificate da una macchina proteica chiamata RNA polimerasi. La RNA polimerasi si muove lungo la catena del DNA, leggendo il codice delle “lettere” del DNA mentre trascrive le istruzioni nelle molecole di RNA, che poi dirigono la costruzione delle proteine. Nudler e colleghi hanno scoperto che durante questo processo di trascrizione, l'RNA polimerasi trova anche lesioni del DNA e quindi funge da piattaforma per l'assemblaggio di una macchina di riparazione del DNA chiamata complesso di riparazione per escissione nucleotidica (NER). Il NER quindi elimina il DNA difettoso trovato e lo sostituisce con una copia accurata. Senza l’azione della RNA polimerasi, nei batteri viventi è presente una quantità minima di NER, se non addirittura nulla. Ora il nuovo studio su Cell fornisce la prima prova che, come nel percorso NER, il RER è strettamente legato alla trascrizione. Gli autori dello studio hanno trovato prove che l’enzima chiave coinvolto nel RER, RNaseHII, coopera anche con l’RNA polimerasi mentre ricerca ribonucleotidi incorporati erroneamente nelle catene di DNA delle cellule batteriche viventi. "I nostri risultati continuano a ispirare un ripensamento di alcuni principi di base nel campo della riparazione del DNA", afferma Nudler, anche lui ricercatore presso l'Howard Hughes Medical Institute. “Andando avanti, il nostro team prevede di indagare se l’RNA polimerasi scansiona il DNA per tutti i tipi di problemi e innesca la riparazione a livello dell’intero genoma, non solo nei batteri, ma anche nelle cellule umane”.
L'enzima in studio RNAseHII ripara il DNA cavalcando l'enzima che legge il codice genetico, l'RNA polimerasi, e ritagliando (vedi forbici) le lettere del codice fuori posto quando le "vede" nel materiale genetico batterico. (© Cell Press) L'anno scorso un team guidato da Evgeny Nudler, PhD, professore Julie Wilson Anderson presso il Dipartimento di Biochimica e Farmacologia Molecolare della NYU Langone Health, ha pubblicato due analisi sulla riparazione del DNA negli organismi viventi E. coli cellule. Hanno scoperto che la maggior parte della riparazione di alcuni tipi di danni al DNA (lesioni voluminose), come quelli causati dall’irradiazione UV, può avvenire perché le sezioni del codice danneggiate sono state prima identificate da una macchina proteica chiamata RNA polimerasi. La RNA polimerasi si muove lungo la catena del DNA, leggendo il codice delle “lettere” del DNA mentre trascrive le istruzioni nelle molecole di RNA, che poi dirigono la costruzione delle proteine. Nudler e colleghi hanno scoperto che durante questo processo di trascrizione, l'RNA polimerasi trova anche lesioni del DNA e quindi funge da piattaforma per l'assemblaggio di una macchina di riparazione del DNA chiamata complesso di riparazione per escissione nucleotidica (NER). Il NER quindi elimina il DNA difettoso trovato e lo sostituisce con una copia accurata. Senza l’azione della RNA polimerasi, nei batteri viventi è presente una quantità minima di NER, se non addirittura nulla. Ora il nuovo studio su Cell fornisce la prima prova che, come nel percorso NER, il RER è strettamente legato alla trascrizione. Gli autori dello studio hanno trovato prove che l’enzima chiave coinvolto nel RER, RNaseHII, coopera anche con l’RNA polimerasi mentre ricerca ribonucleotidi incorporati erroneamente nelle catene di DNA delle cellule batteriche viventi. "I nostri risultati continuano a ispirare un ripensamento di alcuni principi di base nel campo della riparazione del DNA", afferma Nudler, anche lui ricercatore presso l'Howard Hughes Medical Institute. “Andando avanti, il nostro team prevede di indagare se l’RNA polimerasi scansiona il DNA per tutti i tipi di problemi e innesca la riparazione a livello dell’intero genoma, non solo nei batteri, ma anche nelle cellule umane”.
Tecniche all'avanguardia
I ribonucleotidi (gli elementi costitutivi dell'RNA) e i desossiribonucleotidi (componenti del DNA) sono composti correlati. Mentre le cellule copiano e costruiscono catene di DNA nelle cellule batteriche, spesso incorporano erroneamente ribonucleotidi nelle catene di DNA al posto dei desossiribonucleotidi perché differiscono solo per un singolo atomo di ossigeno, affermano gli autori dello studio. È noto che nelle cellule batteriche la DNA polimerasi III commette circa 2,000 di questi errori ogni volta che copia il materiale genetico di una cellula. Per mantenere l’integrità del genoma, la maggior parte dei ribonucleotidi fuori posto viene rimossa dalla via RER, ma una domanda chiave era su come RNaseHII trova lesioni ribonucleotidiche relativamente rare in mezzo a un “oceano” di codici di DNA cellulare intatto così rapidamente. Come hanno fatto nei loro studi del 2022, i ricercatori hanno utilizzato la spettrometria di massa quantitativa e la reticolazione proteina-proteina in vivo per mappare le distanze tra le proteine chimicamente legate, e così hanno determinato le superfici chiave della RNasiHII e della RNA polimerasi mentre interagiscono nelle cellule batteriche viventi. In questo modo hanno determinato che la maggior parte delle molecole di RNasiHII si accoppiano con la RNA polimerasi. Inoltre, hanno utilizzato la microscopia elettronica criogenica (CryoEM) per catturare le strutture ad alta risoluzione della RNasiHII legata alla RNA polimerasi per rivelare le interazioni proteina-proteina che definiscono il complesso RER. Inoltre, gli esperimenti genetici guidati dalla struttura che hanno indebolito l'interazione RNA polimerasi/RNasiHII hanno compromesso il RER. "Questo lavoro supporta un modello in cui RNaseHII scansiona il DNA alla ricerca di ribonucleotidi fuori posto cavalcando la RNA polimerasi mentre si muove lungo il DNA", afferma il primo autore dello studio Zhitai Hao, uno studioso post-dottorato nel laboratorio di Nudler. “Questo lavoro è vitale per la nostra comprensione di base del processo di riparazione del DNA e ha implicazioni cliniche di vasta portata”.- Distribuzione di contenuti basati su SEO e PR. Ricevi amplificazione oggi.
- PlatoAiStream. Intelligenza dei dati Web3. Conoscenza amplificata. Accedi qui.
- Coniare il futuro con Adryenn Ashley. Accedi qui.
- Acquista e vendi azioni in società PRE-IPO con PREIPO®. Accedi qui.
- Fonte: https://www.nanowerk.com/news2/biotech/newsid=63002.php
- :ha
- :È
- :non
- :Dove
- 000
- 10
- 11
- 12
- 2022
- 2023
- 7
- 9
- a
- WRI
- preciso
- Action
- aggiunta
- Aggiunge
- Tutti
- lungo
- anche
- fra
- an
- ed
- in qualsiasi
- SONO
- AS
- montaggio
- At
- atomo
- autore
- gli autori
- batteri
- basic
- perché
- stato
- dietro
- fra
- Bloccare
- Blocchi
- legato
- pause
- costruire
- Costruzione
- ma
- by
- detto
- Materiale
- catturare
- ha causato
- Celle
- centro
- certo
- catena
- Catene
- Modifiche
- Info su
- codice
- complesso
- componenti
- Compromissione
- continuamente
- continua
- copie
- contatori
- Coppia
- accoppiato
- criogenico
- taglio
- Data
- Shirts Department
- determinato
- DID
- differire
- dirette
- dna
- giù
- durante
- bordo
- emergenti del mondo
- errori
- Ogni
- prova
- si è evoluta
- esperimenti
- di vasta portata
- difettoso
- campo
- trova
- Nome
- Nel
- Avanti
- essere trovato
- frequente
- ulteriormente
- genoma
- dato
- ha avuto
- Avere
- Salute e benessere
- ad alta risoluzione
- Come
- HTTPS
- umano
- identificato
- if
- Immagine
- implicazioni
- in
- inclusione
- incorporare
- ispirare
- Istituto
- istruzioni
- interezza
- interagire
- interazione
- interazioni
- ai miglioramenti
- indagare
- coinvolto
- IT
- rivista
- jpg
- Le
- conosciuto
- laboratorio
- Cognome
- L'anno scorso
- Guidato
- piace
- connesso
- piccolo
- vita
- macchina
- mantenere
- make
- carta geografica
- Massa
- materiale
- meccanismo
- medicale
- Microscopia
- In mezzo
- fuori luogo
- errori
- modello
- molecolare
- maggior parte
- motori
- si muove
- New
- adesso
- NYU
- of
- di frequente
- on
- esclusivamente
- Altro
- nostro
- su
- Oxygen
- partner
- immagine
- posto
- piani
- piattaforma
- Platone
- Platone Data Intelligence
- PlatoneDati
- stampa
- principi
- problemi
- processi
- Insegnante
- Proteine
- Proteine
- fornisce
- pubblicato
- quantitativo
- domanda
- rapidamente
- radicalmente
- RARO
- Lettura
- relazionato
- relativamente
- rimosso
- riparazione
- rapporto
- ricercatori
- colpevole
- Risultati
- rivelare
- equitazione
- RNA
- dire
- dice
- sezioni
- vedere
- serve
- singolo
- So
- studi
- Studio
- tale
- supporti
- team
- che
- Il
- loro
- Li
- poi
- Strumenti Bowman per analizzare le seguenti finiture:
- di
- questo
- quelli
- strettamente
- tempo
- a
- seconda
- Digitare
- Tipi di
- e una comprensione reciproca
- utilizzato
- importantissima
- vivo
- Modo..
- WELL
- quando
- se
- quale
- while
- Wilson
- con
- senza
- Lavora
- anno
- zefiro