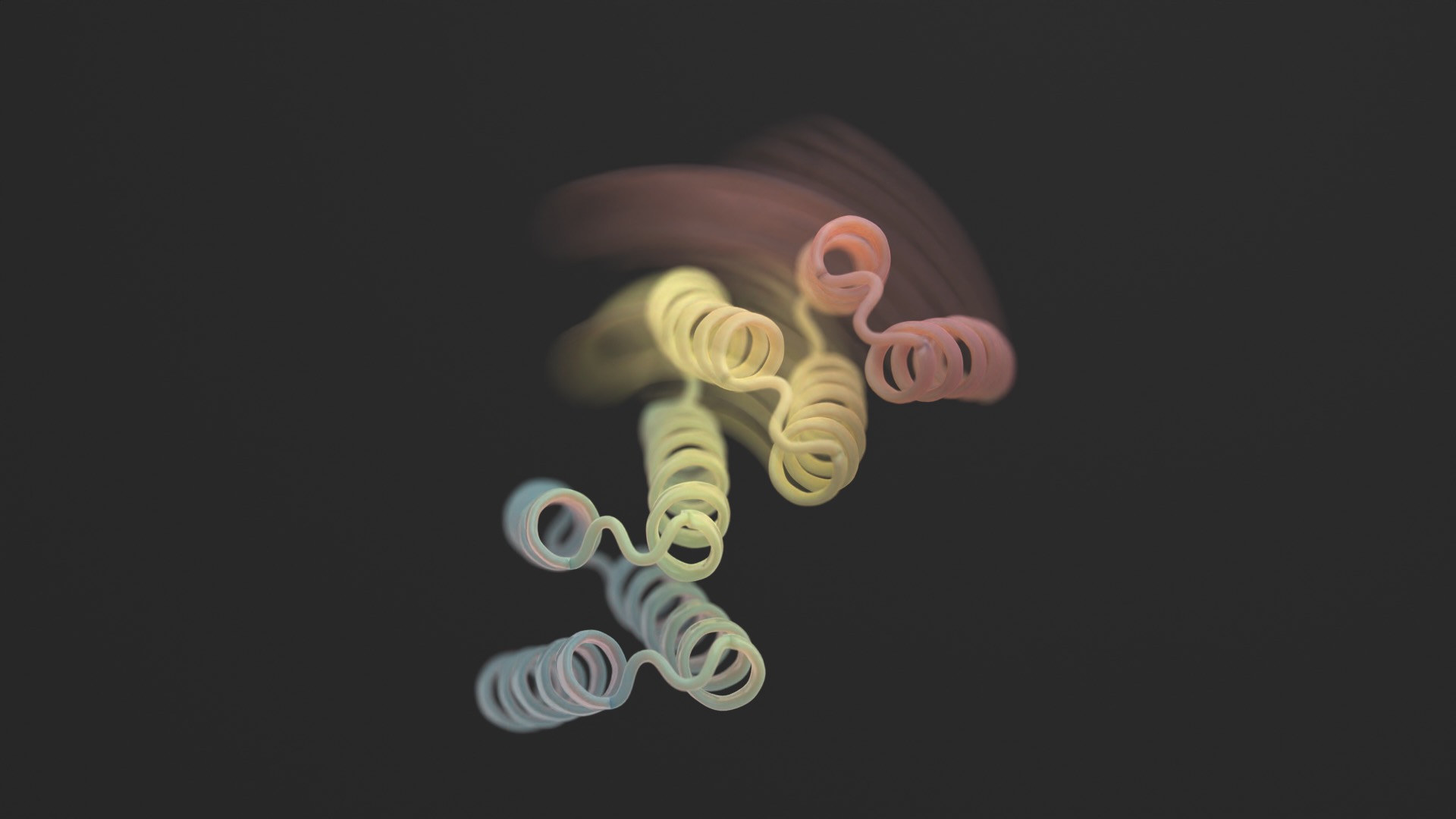
A menudo pensamos en las proteínas como esculturas tridimensionales inmutables.
Eso no es del todo bien. Muchas proteínas son transformadores que se retuercen y cambian de forma según las necesidades biológicas. Una configuración puede propagar señales dañinas de un derrame cerebral o un ataque cardíaco. Otro podría bloquear la cascada molecular resultante y limitar el daño.
En cierto modo, las proteínas actúan como transistores biológicos: interruptores de encendido y apagado en la raíz de la "computadora" molecular del cuerpo que determina cómo reacciona a las fuerzas y la retroalimentación externas e internas. Los científicos han estudiado durante mucho tiempo estas proteínas que cambian de forma para descifrar cómo funcionan nuestros cuerpos.
Pero, ¿por qué confiar solo en la naturaleza? ¿Podemos crear “transistores” biológicos, desconocidos para el universo biológico, desde cero?
Introduzca IA. Múltiples métodos de aprendizaje profundo ya pueden predecir con precisión las estructuras de las proteínas:un gran avance tras medio siglo de gestación. Estudios posteriores que utilizan algoritmos cada vez más potentes han alucinado estructuras de proteínas libres de las fuerzas de la evolución.
Sin embargo, estas estructuras generadas por IA tienen una desventaja: aunque son muy complejas, la mayoría son completamente estáticas, esencialmente, una especie de escultura de proteína digital congelada en el tiempo.
Un nuevo estudio in Ciencia: este mes rompió moldes al agregar flexibilidad a las proteínas de diseño. Las nuevas estructuras no son contorsionistas sin límites. Sin embargo, las proteínas de diseño pueden estabilizarse en dos formas diferentes (piense en una bisagra en una configuración abierta o cerrada) dependiendo de un "bloqueo" biológico externo. Cada estado es análogo al “0” o “1” de una computadora, que posteriormente controla la salida de la celda.
"Antes, solo podíamos crear proteínas que tuvieran una configuración estable", dijo el autor del estudio, el Dr. Florian Praetorius, de la Universidad de Washington. "Ahora, finalmente podemos crear proteínas que se mueven, lo que debería abrir una extraordinaria gama de aplicaciones".
El autor principal, el Dr. David Baker, tiene ideas: "Desde la formación de nanoestructuras que responden a los productos químicos en el medio ambiente hasta las aplicaciones en la administración de fármacos, apenas estamos comenzando a aprovechar su potencial".
Un matrimonio proteico hecho en IA
Un poco de biología 101.
Las proteínas construyen y hacen funcionar nuestros cuerpos. Estas macromoléculas comienzan su viaje a partir del ADN. La información genética se traduce en aminoácidos, los componentes básicos de una proteína: cuentas de imágenes en una cuerda. Luego, cada cuerda se dobla en intrincadas formas 3D, con algunas partes pegadas a otras. Llamadas estructuras secundarias, algunas configuraciones parecen Twizzlers. Otros se tejen en sábanas parecidas a alfombras. Estas formas se complementan entre sí, formando arquitecturas de proteínas altamente sofisticadas.
Al comprender cómo las proteínas adquieren su forma, potencialmente podemos diseñar otras nuevas desde cero, expandiendo el universo biológico y creando nuevas armas contra infecciones virales y otras enfermedades.
En 2020, AlphaFold de DeepMind y RoseTTAFold del laboratorio de David Baker rompieron el Internet de la biología estructural al predecir con precisión las estructuras de las proteínas basándose únicamente en sus secuencias de aminoácidos.
Desde entonces, los modelos de IA han predicho la forma de casi todas las proteínas conocidas y desconocidas para la ciencia. Estas poderosas herramientas ya están remodelando la investigación biológica, ayudando a los científicos a identificar rápidamente objetivos potenciales para combatir la resistencia a los antibióticos, estudiar el “alojamiento” de nuestro ADN, desarrollar nuevas vacunas o incluso arrojar luz sobre enfermedades que devastan el cerebro, como Enfermedad de Parkinson.
Luego llegó una bomba: los modelos de IA generativa, como DALL-E y ChatGPT, ofrecían una perspectiva tentadora. En lugar de simplemente predecir las estructuras de las proteínas, ¿por qué no? haz que la IA sueñe completamente novedoso estructuras de proteínas en su lugar? Desde una proteína que se une a las hormonas para regular los niveles de calcio hasta enzimas artificiales que catalizan la bioluminiscencia, los resultados iniciales despertaron entusiasmo y el potencial de las proteínas diseñadas por IA parecía infinito.
A la cabeza de estos descubrimientos está el laboratorio de Baker. Poco después de lanzar RoseTTAFold, desarrollaron aún más el algoritmo para identificar sitios funcionales en una proteína, donde interactúa con otras proteínas, medicamentos o anticuerpos, allanando el camino para que los científicos inventar nuevos medicamentos que aún no han imaginado.
Sin embargo, faltaba una cosa: flexibilidad. Un gran número de proteínas “cambian de código” de forma para cambiar su mensaje biológico. El resultado podría ser literalmente de vida o muerte: una proteína llamada Bax, por ejemplo, altera su forma en una conformación que desencadena la muerte celular. La beta amiloide, una proteína involucrada en la enfermedad de Alzheimer, toma una forma notoriamente diferente a medida que daña las células cerebrales.
Una IA que alucine proteínas flip-flop similares podría acercarnos más a la comprensión y recapitulación de estos enigmas biológicos, lo que conduciría a nuevas soluciones médicas.
Bisagra, línea y plomada
Diseñar una proteína a nivel atómico y esperar que funcione en una célula viva es difícil. Diseñar uno con dos configuraciones es una pesadilla.
Como una analogía suelta, piense en los cristales de hielo en una nube que eventualmente se convierten en copos de nieve, cada uno con una estructura diferente. El trabajo de la IA es hacer proteínas que puedan cambiar entre dos "copos de nieve" diferentes utilizando los mismos "cristales de hielo" de aminoácidos, con cada estado correspondiente a un interruptor de "encendido" o "apagado". Además, la proteína tiene que jugar bien dentro de las células vivas.
El equipo comenzó con varias reglas. En primer lugar, cada estructura debería verse muy diferente entre los dos estados, como un perfil humano de pie o sentado. Podrían comprobarlo midiendo las distancias entre los átomos, explicó el equipo. En segundo lugar, el cambio debe ocurrir rápido. Esto significa que la proteína no puede desplegarse por completo antes de volver a ensamblarse y tomar otra forma, lo que lleva tiempo.
Luego, hay algunas pautas básicas para una proteína funcional: debe funcionar bien con los líquidos corporales en ambos estados. Finalmente, tiene que actuar como un interruptor, cambiando su forma dependiendo de las entradas y salidas.
Cumplir con todas "estas propiedades en un sistema de proteínas es un desafío", dijo el equipo.
Usando una mezcla de AlphaFold, Rosetta y proteinMPNN, el diseño final parece una bisagra. Tiene dos partes rígidas que pueden moverse entre sí, mientras que otra pieza permanece plegada. Normalmente la proteína está cerrada. El desencadenante es un pequeño péptido (una cadena corta de aminoácidos) que se une a las bisagras y provoca su cambio de forma. Estos llamados "péptidos efectores" se diseñaron cuidadosamente para lograr especificidad, lo que reduce las posibilidades de que se adhieran a partes no objetivo.
El equipo primero añadió péptidos desencadenantes que brillan en la oscuridad a múltiples diseños de bisagras. Un análisis posterior encontró que el gatillo se agarró fácilmente a la bisagra. La configuración de la proteína cambió. Como prueba de cordura, la forma fue predicha previamente mediante análisis de IA.
Estudios adicionales que utilizaron estructuras cristalizadas de los diseños de proteínas, con o sin el efector, validaron aún más los resultados. Estas pruebas también buscaron los principios de diseño que hicieron que la bisagra funcionara y los parámetros que inclinan un estado hacia el otro.
¿La comida para llevar? La IA ahora puede diseñar proteínas con dos estados diferentes, esencialmente construyendo transistores biológicos para la biología sintética. Por ahora, el sistema sólo utiliza péptidos efectores diseñados a medida en sus estudios, lo que puede limitar la investigación y el potencial clínico. Pero según el equipo, la estrategia también puede extenderse a los péptidos naturales, como los que se unen a las proteínas implicadas en la regulación del azúcar en sangre, regulan el agua en los tejidos o influyen en la actividad cerebral.
“Al igual que los transistores en los circuitos electrónicos, podemos acoplar los interruptores a salidas y entradas externas para crear dispositivos de detección e incorporarlos a sistemas de proteínas más grandes”, dijo el equipo.
El autor del estudio, el Dr. Philip Leung, añade: "Esto podría revolucionar la biotecnología de la misma manera que los transistores transformaron la electrónica".
Crédito de la imagen: Ian C Haydon/ Instituto UW para el Diseño de Proteínas
- Distribución de relaciones públicas y contenido potenciado por SEO. Consiga amplificado hoy.
- PlatoData.Network Vertical Generativo Ai. Empodérate. Accede Aquí.
- PlatoAiStream. Inteligencia Web3. Conocimiento amplificado. Accede Aquí.
- PlatoESG. Automoción / vehículos eléctricos, Carbón, tecnología limpia, Energía, Ambiente, Solar, Gestión de residuos. Accede Aquí.
- PlatoSalud. Inteligencia en Biotecnología y Ensayos Clínicos. Accede Aquí.
- ChartPrime. Eleve su juego comercial con ChartPrime. Accede Aquí.
- Desplazamientos de bloque. Modernización de la propiedad de compensaciones ambientales. Accede Aquí.
- Fuente: https://singularityhub.com/2023/08/22/ai-can-now-design-proteins-that-behave-like-biological-transistors/
- :posee
- :es
- :no
- $ UP
- 2020
- 3d
- a
- AC
- Conforme
- precisamente
- Actúe
- actividad
- adicional
- la adición de
- Adicionalmente
- Añade
- Después
- en contra
- AI
- Modelos AI
- algoritmo
- algoritmos
- Todos
- solo
- ya haya utilizado
- también
- Aunque
- Alzheimer
- amiloide
- an
- análisis
- y
- Otra
- aplicaciones
- somos
- AS
- At
- atacar
- autor
- lejos
- Atrás
- panadero
- basado
- BE
- antes
- comenzó
- comenzar
- beta
- entre
- se unen
- biología
- biotecnología
- Poco
- Bloquear
- Bloques
- sangre
- cuerpos
- ambas
- Cerebro
- Actividad cerebral
- células del cerebro
- ruptura
- Rompió
- build
- Construir la
- pero
- by
- , que son
- llegó
- PUEDEN
- estudiar cuidadosamente
- cascadas
- catalizar
- (SCD por sus siglas en inglés),
- Células
- Siglo
- cadena
- desafiante
- posibilidades
- el cambio
- cambiado
- cambio
- ChatGPT
- comprobar
- productos químicos
- Clínico
- cerrado
- más cerca
- Soluciones
- completamente
- Configuración
- controles
- Correspondiente
- podría
- Parejas
- Para crear
- Creamos
- crédito
- de dall
- perjudicial
- David
- Muerte
- Descifrar
- profundo
- deep learning
- Mente profunda
- entrega
- Dependiente
- Diseño
- principios de diseño
- diseñado
- Designer
- diseño
- diseños
- determinar
- desarrollado
- Dispositivos
- una experiencia diferente
- digital
- Enfermedades
- enfermedad
- ADN
- DE INSCRIPCIÓN
- caída
- dr
- sueño
- droga
- Entrega de medicamentos
- Drogas
- cada una
- pasan fácilmente
- Southern Implants
- Efector
- ya sea
- Electronic
- Electrónica
- Sin fin
- ingeniero
- entusiasmo
- Entorno
- Incluso
- finalmente
- Cada
- evolución
- ejemplo
- en expansión
- explicado
- ampliar
- externo
- un recuerdo extraordinario
- RÁPIDO
- realimentación
- final
- Finalmente
- Nombre
- Flexibilidad
- Fuerzas
- formulario
- encontrado
- Desde
- congelado
- función
- funcional
- promover
- Obtén
- generativo
- IA generativa
- orientaciones
- tenido
- A Mitad
- suceder
- Difícil
- daño
- daños
- Tienen
- Corazón
- Ataque del Corazón
- ayudando
- altamente
- Bisagra
- con la esperanza
- Cómo
- Sin embargo
- HTTPS
- humana
- HIELO
- ideas
- imaginado
- inmutable
- in
- incorporar
- cada vez más
- Infecciones
- influir
- información
- inicial
- entradas
- dentro
- Innovadora
- interactúa
- interno
- Internet
- dentro
- involucra
- IT
- SUS
- sí mismo
- Trabajos
- solo
- el lab
- large
- mayores
- aprendizaje
- Vida
- luz
- como
- LIMITE LAS
- límites
- línea
- alga viva
- Largo
- Mira
- parece
- MIRADAS
- bajando
- hecho
- para lograr
- muchos
- Puede..
- significa
- medición
- servicios
- mensaje
- métodos
- que falta
- mezcla
- modelos
- molecular
- Mes
- MEJOR DE TU
- movimiento
- múltiples
- Natural
- Naturaleza
- Nuevo
- agradable
- NIH
- normalmente
- ahora
- número
- of
- Ofrecido
- a menudo
- on
- ONE
- las
- , solamente
- habiertos
- or
- Otro
- Otros
- "nuestr
- salida
- parámetros
- partes
- pieza
- Platón
- Inteligencia de datos de Platón
- PlatónDatos
- Jugar
- posible
- la posibilidad
- poderoso
- predecir
- previsto
- predecir
- previamente
- principios
- Mi Perfil
- propiedades
- perspectiva
- Proteínas
- Proteínas
- Búsqueda
- con rapidez
- distancia
- más bien
- Reacciona
- Regula tu
- reguladora
- relativo
- la liberación de
- confiar
- permanece
- la investigación
- Responder
- resultado
- resultante
- Resultados
- revolucionar
- Derecho
- rígido
- raíz
- reglas
- Ejecutar
- Said
- mismo
- Ciencia:
- los científicos
- rayar
- Segundo
- secundario
- parecía
- Varios
- Forma
- formas
- cobertizo
- Turno
- En Corto
- Dentro de poco
- tienes
- señales
- similares
- simplemente
- Sitios Web
- Sentado
- chica
- copos de nieve
- Soluciones
- algo
- sofisticado
- provocado
- especificidad
- estabilizar
- estable
- Comience a
- Estado
- Zonas
- pega
- Estrategia
- Cordón
- estructural
- estructura
- estudiado
- estudios
- ESTUDIO
- posterior
- Después
- tal
- azúcar
- Switch
- sintético
- te
- Todas las funciones a su disposición
- ¡Prepárate!
- toma
- Pulsa para buscar
- tiene como objetivo
- equipo
- pruebas
- que
- esa
- La
- su
- Les
- luego
- Ahí.
- Estas
- ellos
- cosa
- pensar
- así
- aquellos
- equipo
- de estilista
- tejidos
- a
- juntos
- transformado
- transformers
- detonante
- torcedura
- dos
- comprensión
- Universo
- universidad
- Universidad de Washington
- desconocido
- us
- usos
- usando
- validado
- virales
- fue
- Washington
- Agua
- Camino..
- we
- Armas
- Acabado
- web
- tuvieron
- que
- mientras
- porque
- sin
- Actividades:
- funciona
- aún
- zephyrnet