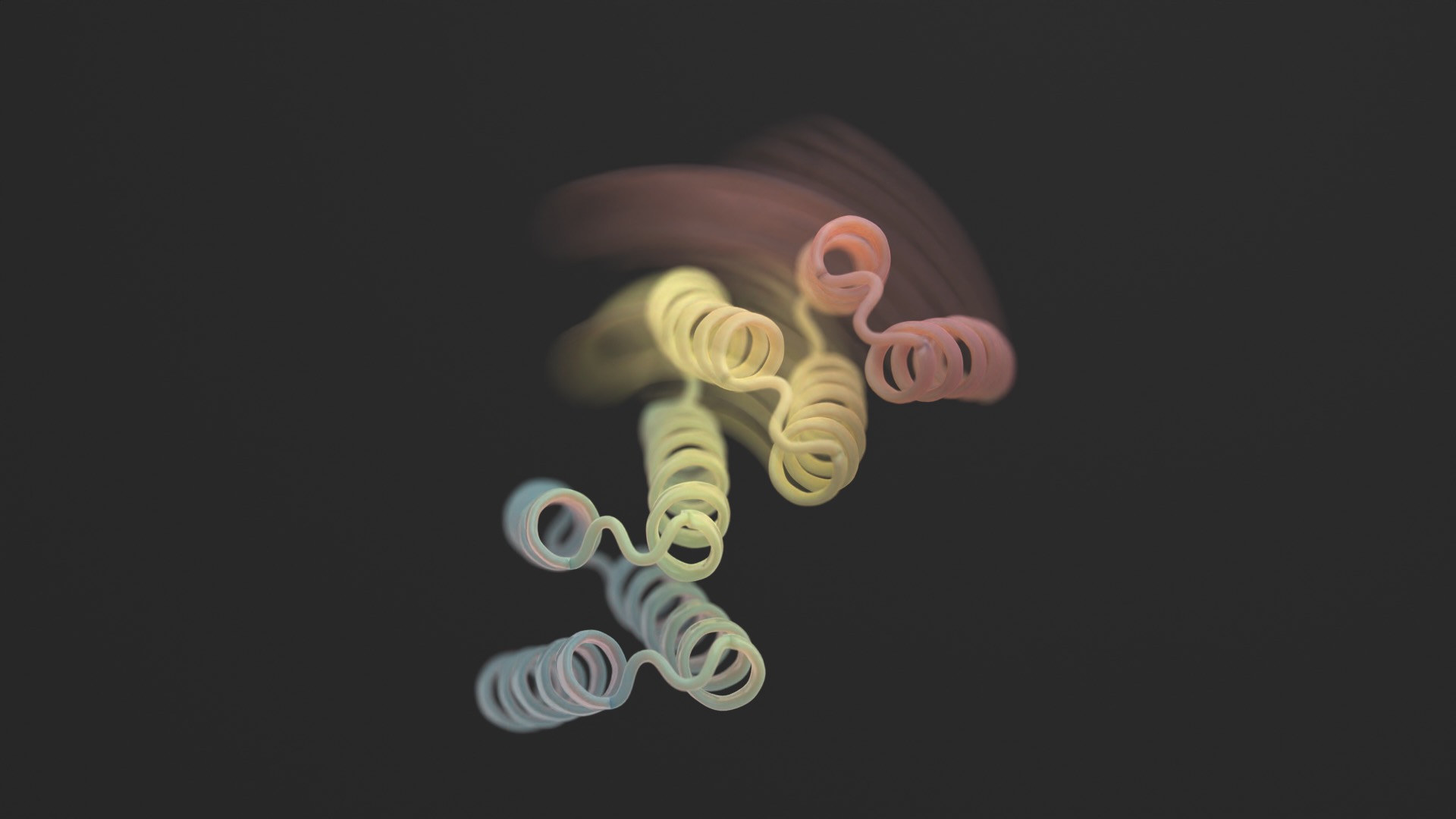
Vi tænker ofte på proteiner som uforanderlige 3D-skulpturer.
Det er ikke helt rigtigt. Mange proteiner er transformatorer, der vrider og ændrer deres former afhængigt af biologiske behov. En konfiguration kan udbrede skadelige signaler fra et slagtilfælde eller hjerteanfald. En anden kan blokere den resulterende molekylære kaskade og begrænse skaden.
På en måde fungerer proteiner som biologiske transistorer - tænd-sluk-kontakter ved roden af kroppens molekylære "computer", der bestemmer, hvordan den reagerer på eksterne og interne kræfter og feedback. Forskere har længe undersøgt disse formskiftende proteiner for at afkode, hvordan vores kroppe fungerer.
Men hvorfor stole på naturen alene? Kan vi skabe biologiske "transistorer", ukendte for det biologiske univers, fra bunden?
Indtast AI. Flere dyb læringsmetoder kan allerede præcist forudsige proteinstrukturer—et gennembrud et halvt århundrede undervejs. Efterfølgende undersøgelser, der anvender stadig stærkere algoritmer, har hallucinerede proteinstrukturer, der ikke er bundet af evolutionens kræfter.
Alligevel har disse AI-genererede strukturer en undergang: Selvom de er meget indviklede, er de fleste fuldstændig statiske - i det væsentlige en slags digital proteinskulptur frosset i tid.
En ny undersøgelse in Videnskab denne måned brød formen ved at tilføje fleksibilitet til designerproteiner. De nye strukturer er ikke kontortionister uden grænser. Designerproteinerne kan dog stabilisere sig i to forskellige former - tænk et hængsel i enten en åben eller lukket konfiguration - afhængigt af en ekstern biologisk "lås". Hver tilstand er analog med en computers "0" eller "1", som efterfølgende styrer cellens output.
"Før kunne vi kun skabe proteiner, der havde én stabil konfiguration," sagde undersøgelsesforfatter Dr. Florian Praetorius ved University of Washington. "Nu kan vi endelig skabe proteiner, der bevæger sig, hvilket burde åbne op for en ekstraordinær række anvendelser."
Hovedforfatter Dr. David Baker har ideer: "Fra at danne nanostrukturer, der reagerer på kemikalier i miljøet, til anvendelser i lægemiddellevering, er vi lige begyndt at udnytte deres potentiale."
Et proteinægteskab lavet i AI
En hurtig bid af biologi 101.
Proteiner bygger og driver vores krop. Disse makromolekyler begynder deres rejse fra DNA. Genetisk information omsættes til aminosyrer, byggestenene i et protein - billedperler på en snor. Hver streng foldes derefter til indviklede 3D-former, hvor nogle dele klæber til andre. Kaldet sekundære strukturer, nogle konfigurationer ligner Twizzlers. Andre væver ind i tæppelignende lagner. Disse former bygger yderligere på hinanden og danner meget sofistikerede proteinarkitekturer.
Ved at forstå, hvordan proteiner får deres former, kan vi potentielt konstruere nye fra bunden, udvide det biologiske univers og skabe nye våben mod virusinfektioner og andre sygdomme.
Tilbage i 2020 brød DeepMinds AlphaFold og David Baker labs RoseTTAFold det strukturelle biologiske internet ved nøjagtigt at forudsige proteinstrukturer baseret på deres aminosyresekvenser alene.
Siden da har AI-modellerne forudsagt formen af næsten alle proteiner, der er kendt - og ukendt - for videnskaben. Disse kraftfulde værktøjer er allerede ved at omforme den biologiske forskning og hjælper videnskabsmænd med hurtigt at finde potentielle mål bekæmpe antibiotikaresistens, studere "husning" af vores DNA, udvikle nye vacciner eller endda kaste lys over sygdomme, der hærger hjernen, som f.eks Parkinsons sygdom.
Så kom en bombe: Generative AI-modeller, såsom DALL-E og ChatGPT, tilbød en fristende udsigt. I stedet for blot at forudsige proteinstrukturer, hvorfor ikke har AI drømt op helt ny proteinstrukturer i stedet? Fra et protein, der binder hormoner til at regulere calciumniveauet til kunstige enzymer som katalyserer bioluminescens, udløste de første resultater entusiasme, og potentialet for AI-designede proteiner syntes uendeligt.
I spidsen for disse opdagelser er Bakers laboratorium. Kort efter udgivelsen af RoseTTAFold videreudviklede de algoritmen til at fastgøre funktionelle steder på et protein – hvor det interagerer med andre proteiner, lægemidler eller antistoffer – og baner vejen for videnskabsmænd til at drømme op ny medicin, de endnu ikke har forestillet sig.
Alligevel manglede én ting: fleksibilitet. Et stort antal proteiner "kodeskift" i form for at ændre deres biologiske budskab. Resultatet kunne bogstaveligt talt være liv eller død: et protein kaldet Bax, for eksempel, ændrer sin form til en konformation, der udløser celledød. Amyloid beta, et protein involveret i Alzheimers sygdom, antager notorisk en anden form, da det skader hjerneceller.
En kunstig intelligens, der hallucinerer lignende flip-flop-proteiner, kan bringe os tættere på at forstå og rekapitulere disse biologiske gåder – hvilket fører til nye medicinske løsninger.
Hinge, Line og Sinker
At designe et protein på atomniveau - og håbe på, at det virker i en levende celle - er svært. At designe en med to konfigurationer er et mareridt.
Som en løs analogi, tænk på iskrystaller i en sky, der til sidst dannes til snefnug, hver især forskellig i struktur. AI's job er at lave proteiner, der kan skifte mellem to forskellige "snefnug" ved hjælp af de samme aminosyre-"iskrystaller", hvor hver tilstand svarer til en "tændt" eller "sluk"-knap. Derudover skal proteinet spille godt inde i levende celler.
Holdet begyndte med flere regler. For det første skal hver struktur se vidt forskellig ud mellem de to stater - som en menneskelig profil stående eller siddende. De kunne kontrollere dette ved at måle afstande mellem atomer, forklarede holdet. For det andet skal forandringen ske hurtigt. Det betyder, at proteinet ikke kan udfolde sig fuldstændigt, før det stykker sig sammen igen til en anden form, hvilket tager tid.
Så er der nogle gode retningslinjer for et funktionelt protein: det skal spille godt med kropsvæsker i begge tilstande. Endelig skal den fungere som en kontakt, der ændrer form afhængigt af input og output.
At møde alle "disse egenskaber i ét proteinsystem er udfordrende," sagde teamet.
Ved at bruge en blanding af AlphaFold, Rosetta og proteinMPNN ligner det endelige design et hængsel. Den har to stive dele, der kan bevæge sig i forhold til hinanden, mens et andet stykke forbliver foldet. Normalt er proteinet lukket. Udløseren er et lille peptid - en kort kæde af aminosyrer - der binder sig til hængslerne og udløser formændringen. Disse såkaldte "effektorpeptider" blev omhyggeligt designet til specificitet, hvilket sænker deres chancer for at gribe fat i dele uden for målet.
Holdet tilføjede først glød-i-mørke-trigger-peptider til flere hængseldesigns. Efterfølgende analyse viste, at aftrækkeren nemt greb ind i hængslet. Proteinets konfiguration ændrede sig. Som en fornuftskontrol var formen en tidligere forudsagt ved hjælp af AI-analyse.
Yderligere undersøgelser ved hjælp af krystalliserede strukturer af proteindesignerne, enten med eller uden effektoren, validerede resultaterne yderligere. Disse tests jagtede også designprincipper, der fik hængslet til at fungere, og parametre, der tipper den ene tilstand til den anden.
Take away? AI kan nu designe proteiner med to forskellige tilstande - i det væsentlige bygge biologiske transistorer til syntetisk biologi. Indtil videre bruger systemet kun specialdesignede effektorpeptider i deres undersøgelser, hvilket kan begrænse forskning og klinisk potentiale. Men ifølge teamet kan strategien også strække sig til naturlige peptider, såsom dem, der binder proteiner involveret i regulering af blodsukker, regulerer vand i væv eller påvirker hjerneaktivitet.
"Som transistorer i elektroniske kredsløb kan vi koble switchene til eksterne udgange og indgange for at skabe sensorenheder og inkorporere dem i større proteinsystemer," sagde teamet.
Studieforfatter Dr. Philip Leung tilføjer: "Dette kunne revolutionere bioteknologien på samme måde som transistorer transformerede elektronik."
Billedkredit: Ian C Haydon/UW Institute for Protein Design
- SEO Powered Content & PR Distribution. Bliv forstærket i dag.
- PlatoData.Network Vertical Generative Ai. Styrk dig selv. Adgang her.
- PlatoAiStream. Web3 intelligens. Viden forstærket. Adgang her.
- PlatoESG. Automotive/elbiler, Kulstof, CleanTech, Energi, Miljø, Solenergi, Affaldshåndtering. Adgang her.
- PlatoHealth. Bioteknologiske og kliniske forsøgs intelligens. Adgang her.
- ChartPrime. Løft dit handelsspil med ChartPrime. Adgang her.
- BlockOffsets. Modernisering af miljømæssig offset-ejerskab. Adgang her.
- Kilde: https://singularityhub.com/2023/08/22/ai-can-now-design-proteins-that-behave-like-biological-transistors/
- :har
- :er
- :ikke
- $OP
- 2020
- 3d
- a
- AC
- Ifølge
- præcist
- Lov
- aktivitet
- tilføjet
- tilføje
- Derudover
- Tilføjer
- Efter
- mod
- AI
- AI modeller
- algoritme
- algoritmer
- Alle
- alene
- allerede
- også
- Skønt
- Alzheimers
- amyloid
- an
- analyse
- ,
- En anden
- applikationer
- ER
- AS
- At
- angribe
- forfatter
- væk
- tilbage
- bager
- baseret
- BE
- før
- begyndte
- begynde
- beta
- mellem
- binde
- biologi
- bioteknologi
- Bit
- Bloker
- Blocks
- blod
- organer
- både
- Brain
- Hjerneaktivitet
- hjerneceller
- gennembrud
- Broke
- bygge
- Bygning
- men
- by
- kaldet
- kom
- CAN
- omhyggeligt
- vandfald
- katalyserer
- celle
- Celler
- Århundrede
- kæde
- udfordrende
- odds
- lave om
- ændret
- skiftende
- ChatGPT
- kontrollere
- kemikalier
- Klinisk
- lukket
- tættere
- Cloud
- fuldstændig
- Konfiguration
- kontrol
- Tilsvarende
- kunne
- Par
- skabe
- Oprettelse af
- kredit
- dalls
- beskadige
- David
- Død
- dechifrere
- dyb
- dyb læring
- DeepMind
- levering
- Afhængigt
- Design
- design principper
- konstrueret
- Designer
- designe
- designs
- bestemmelse
- udviklet
- Enheder
- forskellige
- digital
- Sygdom
- sygdomme
- dna
- ned
- fald
- dr
- drøm
- medicin
- Levering af lægemidler
- Narkotika
- hver
- nemt
- Edge
- effektor
- enten
- elektronisk
- Elektronik
- Endless
- ingeniør
- entusiasme
- Miljø
- Endog
- til sidst
- Hver
- evolution
- eksempel
- ekspanderende
- forklarede
- udvide
- ekstern
- ekstraordinære
- FAST
- tilbagemeldinger
- endelige
- Endelig
- Fornavn
- Fleksibilitet
- Til
- Forces
- formular
- fundet
- fra
- frosset
- funktion
- funktionel
- yderligere
- Gevinst
- generative
- Generativ AI
- retningslinjer
- havde
- Halvdelen
- ske
- Hård Ost
- skade
- skader
- Have
- Hjerte
- Heart Attack
- hjælpe
- stærkt
- Hængsel
- håber
- Hvordan
- Men
- HTTPS
- menneskelig
- ICE
- ideer
- forestillet
- uforanderlige
- in
- indarbejde
- stigende
- Infektioner
- indflydelse
- oplysninger
- initial
- indgange
- indvendig
- i stedet
- Institut
- interagerer
- interne
- Internet
- ind
- involverede
- IT
- ITS
- selv
- Job
- rejse
- lige
- lab
- stor
- større
- læring
- niveauer
- Livet
- lys
- ligesom
- GRÆNSE
- grænser
- Line (linje)
- levende
- Lang
- Se
- ligner
- UDSEENDE
- sænkning
- lavet
- lave
- mange
- Kan..
- midler
- måling
- medicinsk
- besked
- metoder
- mangler
- blande
- modeller
- molekylær
- Måned
- mest
- bevæge sig
- flere
- Natural
- Natur
- behov
- Ny
- rart
- NIH
- Normalt
- nu
- nummer
- of
- tilbydes
- tit
- on
- ONE
- dem
- kun
- åbent
- or
- Andet
- Andre
- vores
- output
- parametre
- dele
- stykke
- plato
- Platon Data Intelligence
- PlatoData
- Leg
- potentiale
- potentielt
- vigtigste
- forudsige
- forudsagde
- forudsige
- tidligere
- principper
- Profil
- egenskaber
- udsigten
- Protein
- Proteiner
- Hurtig
- hurtigt
- rækkevidde
- hellere
- reagerer
- Regulere
- regulerings-
- relative
- frigive
- stole
- resterne
- forskning
- Svar
- resultere
- resulterer
- Resultater
- revolutionere
- højre
- stiv
- rod
- regler
- Kør
- Said
- samme
- Videnskab
- forskere
- ridse
- Anden
- sekundær
- syntes
- flere
- Shape
- former
- kaste
- skifte
- Kort
- Inden længe
- bør
- signaler
- lignende
- ganske enkelt
- Websteder
- Siddende
- lille
- snefnug
- Løsninger
- nogle
- sofistikeret
- udløst
- specificitet
- stabilisere
- stabil
- Starter
- Tilstand
- Stater
- stikning
- Strategi
- String
- strukturel
- struktur
- studeret
- undersøgelser
- Studere
- efterfølgende
- Efterfølgende
- sådan
- sukker
- Kontakt
- syntetisk
- systemet
- Systemer
- Tag
- tager
- Tryk på
- mål
- hold
- tests
- end
- at
- deres
- Them
- derefter
- Der.
- Disse
- de
- ting
- tror
- denne
- dem
- tid
- tip
- væv
- til
- sammen
- værktøjer
- omdannet
- transformers
- udløse
- Twist
- to
- forståelse
- Universe
- universitet
- University of Washington
- ukendt
- us
- bruger
- ved brug af
- valideret
- viral
- var
- washington
- Vand
- Vej..
- we
- Våben
- Weave
- web
- var
- som
- mens
- hvorfor
- med
- uden
- Arbejde
- virker
- endnu
- zephyrnet